[日本語] English
 万見
万見- EMDB-28586: Cryo-EM structure of the organic cation transporter 1 in the apo state -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the organic cation transporter 1 in the apo state | |||||||||
 マップデータ マップデータ | main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | organic cation / transport / membrane protein / TRANSPORT PROTEIN | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.57 Å | |||||||||
 データ登録者 データ登録者 | Suo Y / Wright NJ / Lee S-Y | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 | ジャーナル: bioRxiv / 年: 2023 タイトル: Molecular basis of polyspecific drug binding and transport by OCT1 and OCT2. 著者: Yang Suo / Nicholas J Wright / Hugo Guterres / Justin G Fedor / Kevin John Butay / Mario J Borgnia / Wonpil Im / Seok-Yong Lee /  要旨: A wide range of endogenous and xenobiotic organic ions require facilitated transport systems to cross the plasma membrane for their disposition . In mammals, organic cation transporter subtypes 1 ...A wide range of endogenous and xenobiotic organic ions require facilitated transport systems to cross the plasma membrane for their disposition . In mammals, organic cation transporter subtypes 1 and 2 (OCT1 and OCT2, also known as SLC22A1 and SLC22A2, respectively) are polyspecific transporters responsible for the uptake and clearance of structurally diverse cationic compounds in the liver and kidneys, respectively . Notably, it is well established that human OCT1 and OCT2 play central roles in the pharmacokinetics, pharmacodynamics, and drug-drug interactions (DDI) of many prescription medications, including metformin . Despite their importance, the basis of polyspecific cationic drug recognition and the alternating access mechanism for OCTs have remained a mystery. Here, we present four cryo-EM structures of apo, substrate-bound, and drug-bound OCT1 and OCT2 in outward-facing and outward-occluded states. Together with functional experiments, docking, and molecular dynamics simulations, these structures uncover general principles of organic cation recognition by OCTs and illuminate unexpected features of the OCT alternating access mechanism. Our findings set the stage for a comprehensive structure-based understanding of OCT-mediated DDI, which will prove critical in the preclinical evaluation of emerging therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
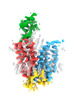
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28586.map.gz emd_28586.map.gz | 20.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28586-v30.xml emd-28586-v30.xml emd-28586.xml emd-28586.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28586.png emd_28586.png | 44.2 KB | ||
| その他 |  emd_28586_half_map_1.map.gz emd_28586_half_map_1.map.gz emd_28586_half_map_2.map.gz emd_28586_half_map_2.map.gz | 28.3 MB 28.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28586 http://ftp.pdbj.org/pub/emdb/structures/EMD-28586 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28586 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28586 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28586_validation.pdf.gz emd_28586_validation.pdf.gz | 873.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28586_full_validation.pdf.gz emd_28586_full_validation.pdf.gz | 872.7 KB | 表示 | |
| XML形式データ |  emd_28586_validation.xml.gz emd_28586_validation.xml.gz | 10.7 KB | 表示 | |
| CIF形式データ |  emd_28586_validation.cif.gz emd_28586_validation.cif.gz | 12.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28586 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28586 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28586 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28586 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28586.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28586.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map 2
| ファイル | emd_28586_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
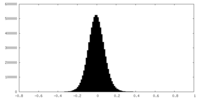
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_28586_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
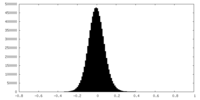
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : OCT1
| 全体 | 名称: OCT1 |
|---|---|
| 要素 |
|
-超分子 #1: OCT1
| 超分子 | 名称: OCT1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 60 kDa/nm |
-分子 #1: OCT1
| 分子 | 名称: OCT1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 61.299562 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MPTVDDVLEQ VGEFGWFQKQ AFLTLCLLSA AFAPIYVGIV FLGFTPDHRC RSPGVAELSQ RCGWSLAEEL NYTVPGLGAA GEAFPRQCR RYEVDWNQSA LSCVDPLASL AANRSHLPLG PCQHGWVYDT PGSSIVTEFN LVCADSWKVD LFQSCVNVGF F LGSLGVGY ...文字列: MPTVDDVLEQ VGEFGWFQKQ AFLTLCLLSA AFAPIYVGIV FLGFTPDHRC RSPGVAELSQ RCGWSLAEEL NYTVPGLGAA GEAFPRQCR RYEVDWNQSA LSCVDPLASL AANRSHLPLG PCQHGWVYDT PGSSIVTEFN LVCADSWKVD LFQSCVNVGF F LGSLGVGY IADRFGRKLC LLATTLISAV SGVLMAVAPD YTSMLLFRLL QGLVSKGSWM SGYTLITEFV GSGYRRTVAI LY QMAFTVG LVLLSGVAYA IPHWRWLQLA VSLPTFLFLL YYWCVPESPR WLLSQKRNTQ AIKIMDHIAQ KNGKLPPADL KML SLKEDS TEKLSPSFAD LFRTPQLRKH TFILMYLWFT SSVLYQGLIM HMGATGGNLY LDFFYSALVE FPAAFIILVT IDRV GRIYP LAVSNLVAGA ACLIMIFISQ DLHWLNITVA CVGRMGITIV FQMVCLVNAE LYPTFIRNLG VMVCSSLCDL GGIIT PFLV FRLMEVWQGL PLILFTVVGL VAGGMTLLLP ETKGVALPET IEDAENLGRK AKPKENTIYL QVQTSELPGT |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 240 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 5993 / 平均露光時間: 3.7 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 152.3 |
|---|---|
| 得られたモデル |  PDB-8et6: |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X