+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Sub-tomogram averaging of erythrocyte ankyrin-1 complex | |||||||||
 マップデータ マップデータ | Main map (sub-tomogram average of ankyrin complex particles). | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Membrane protein / ankyrin complex / TRANSPORT PROTEIN-STRUCTURAL PROTEIN complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報methylammonium transmembrane transport / Defective RHAG causes regulator type Rh-null hemolytic anemia (RHN) / Rhesus blood group biosynthesis / methylammonium transmembrane transporter activity / Rhesus glycoproteins mediate ammonium transport / carbon dioxide transmembrane transport / carbon dioxide transmembrane transporter activity / ammonium homeostasis / spectrin-associated cytoskeleton / hemoglobin metabolic process ...methylammonium transmembrane transport / Defective RHAG causes regulator type Rh-null hemolytic anemia (RHN) / Rhesus blood group biosynthesis / methylammonium transmembrane transporter activity / Rhesus glycoproteins mediate ammonium transport / carbon dioxide transmembrane transport / carbon dioxide transmembrane transporter activity / ammonium homeostasis / spectrin-associated cytoskeleton / hemoglobin metabolic process / positive regulation of organelle organization / protein-glutamine gamma-glutamyltransferase activity / leak channel activity / maintenance of epithelial cell apical/basal polarity / pH elevation / Defective SLC4A1 causes hereditary spherocytosis type 4 (HSP4), distal renal tubular acidosis (dRTA) and dRTA with hemolytic anemia (dRTA-HA) / negative regulation of urine volume / NrCAM interactions / Neurofascin interactions / ammonium transmembrane transport / Bicarbonate transporters / intracellular monoatomic ion homeostasis / ammonium channel activity / ankyrin-1 complex / CHL1 interactions / plasma membrane phospholipid scrambling / monoatomic anion transmembrane transporter activity / cytoskeletal anchor activity / chloride:bicarbonate antiporter activity / solute:inorganic anion antiporter activity / bicarbonate transport / bicarbonate transmembrane transporter activity / M band / monoatomic anion transport / inorganic cation transmembrane transport / Interaction between L1 and Ankyrins / chloride transport / chloride transmembrane transporter activity / ankyrin binding / negative regulation of glycolytic process through fructose-6-phosphate / hemoglobin binding / spectrin binding / cortical cytoskeleton / erythrocyte maturation / exocytosis / axolemma / endoplasmic reticulum to Golgi vesicle-mediated transport / erythrocyte development / COPI-mediated anterograde transport / protein-membrane adaptor activity / spleen development / chloride transmembrane transport / cytoskeleton organization / sarcoplasmic reticulum / protein localization to plasma membrane / regulation of intracellular pH / Cell surface interactions at the vascular wall / carbon dioxide transport / Erythrocytes take up oxygen and release carbon dioxide / Erythrocytes take up carbon dioxide and release oxygen / cell morphogenesis / transmembrane transport / sarcolemma / structural constituent of cytoskeleton / Z disc / multicellular organismal-level iron ion homeostasis / cytoplasmic side of plasma membrane / blood coagulation / virus receptor activity / regulation of cell shape / ATPase binding / basolateral plasma membrane / protein phosphatase binding / postsynaptic membrane / blood microparticle / transmembrane transporter binding / cytoskeleton / neuron projection / structural molecule activity / enzyme binding / signal transduction / protein homodimerization activity / extracellular exosome / nucleoplasm / ATP binding / identical protein binding / membrane / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
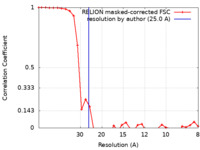
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 25.0 Å | |||||||||
 データ登録者 データ登録者 | Vallese F / Kim K / Yen LY / Johnston JD / Noble AJ / Cali T / Clarke OB | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Architecture of the human erythrocyte ankyrin-1 complex. 著者: Francesca Vallese / Kookjoo Kim / Laura Y Yen / Jake D Johnston / Alex J Noble / Tito Calì / Oliver Biggs Clarke /   要旨: The stability and shape of the erythrocyte membrane is provided by the ankyrin-1 complex, but how it tethers the spectrin-actin cytoskeleton to the lipid bilayer and the nature of its association ...The stability and shape of the erythrocyte membrane is provided by the ankyrin-1 complex, but how it tethers the spectrin-actin cytoskeleton to the lipid bilayer and the nature of its association with the band 3 anion exchanger and the Rhesus glycoproteins remains unknown. Here we present structures of ankyrin-1 complexes purified from human erythrocytes. We reveal the architecture of a core complex of ankyrin-1, the Rhesus proteins RhAG and RhCE, the band 3 anion exchanger, protein 4.2, glycophorin A and glycophorin B. The distinct T-shaped conformation of membrane-bound ankyrin-1 facilitates recognition of RhCE and, unexpectedly, the water channel aquaporin-1. Together, our results uncover the molecular details of ankyrin-1 association with the erythrocyte membrane, and illustrate the mechanism of ankyrin-mediated membrane protein clustering. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26965.map.gz emd_26965.map.gz | 1.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26965-v30.xml emd-26965-v30.xml emd-26965.xml emd-26965.xml | 30.9 KB 30.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26965_fsc.xml emd_26965_fsc.xml | 2.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26965.png emd_26965.png | 34.3 KB | ||
| マスクデータ |  emd_26965_msk_1.map emd_26965_msk_1.map | 2 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-26965.cif.gz emd-26965.cif.gz | 9.1 KB | ||
| その他 |  emd_26965_half_map_1.map.gz emd_26965_half_map_1.map.gz emd_26965_half_map_2.map.gz emd_26965_half_map_2.map.gz | 1.4 MB 1.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26965 http://ftp.pdbj.org/pub/emdb/structures/EMD-26965 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26965 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26965 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26965_validation.pdf.gz emd_26965_validation.pdf.gz | 789.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26965_full_validation.pdf.gz emd_26965_full_validation.pdf.gz | 789.4 KB | 表示 | |
| XML形式データ |  emd_26965_validation.xml.gz emd_26965_validation.xml.gz | 7.9 KB | 表示 | |
| CIF形式データ |  emd_26965_validation.cif.gz emd_26965_validation.cif.gz | 10.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26965 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26965 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26965 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26965 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8cslMC  7uz3C  7uzeC  7uzqC  7uzsC  7uzuC  7uzvC  7v07C  7v0kC  7v0mC  7v0qC  7v0sC  7v0tC  7v0uC  7v0xC  7v0yC  7v19C  8crqC  8crrC  8crtC  8cs9C  8csvC  8cswC  8csxC  8csyC  8ct2C  8ct3C  8cteC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26965.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26965.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map (sub-tomogram average of ankyrin complex particles). | ||||||||||||||||||||||||||||||||||||
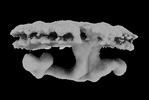
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26965_msk_1.map emd_26965_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_26965_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_26965_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Sub-tomogram average of ankyrin-1 complexes from native erythrocy...
| 全体 | 名称: Sub-tomogram average of ankyrin-1 complexes from native erythrocyte vesicles |
|---|---|
| 要素 |
|
-超分子 #1: Sub-tomogram average of ankyrin-1 complexes from native erythrocy...
| 超分子 | 名称: Sub-tomogram average of ankyrin-1 complexes from native erythrocyte vesicles タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Ankyrin-1
| 分子 | 名称: Ankyrin-1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 206.522625 KDa |
| 配列 | 文字列: MPYSVGFREA DAATSFLRAA RSGNLDKALD HLRNGVDINT CNQNGLNGLH LASKEGHVKM VVELLHKEII LETTTKKGNT ALHIAALAG QDEVVRELVN YGANVNAQSQ KGFTPLYMAA QENHLEVVKF LLENGANQNV ATEDGFTPLA VALQQGHENV V AHLINYGT ...文字列: MPYSVGFREA DAATSFLRAA RSGNLDKALD HLRNGVDINT CNQNGLNGLH LASKEGHVKM VVELLHKEII LETTTKKGNT ALHIAALAG QDEVVRELVN YGANVNAQSQ KGFTPLYMAA QENHLEVVKF LLENGANQNV ATEDGFTPLA VALQQGHENV V AHLINYGT KGKVRLPALH IAARNDDTRT AAVLLQNDPN PDVLSKTGFT PLHIAAHYEN LNVAQLLLNR GASVNFTPQN GI TPLHIAS RRGNVIMVRL LLDRGAQIET KTKDELTPLH CAARNGHVRI SEILLDHGAP IQAKTKNGLS PIHMAAQGDH LDC VRLLLQ YDAEIDDITL DHLTPLHVAA HCGHHRVAKV LLDKGAKPNS RALNGFTPLH IACKKNHVRV MELLLKTGAS IDAV TESGL TPLHVASFMG HLPIVKNLLQ RGASPNVSNV KVETPLHMAA RAGHTEVAKY LLQNKAKVNA KAKDDQTPLH CAARI GHTN MVKLLLENNA NPNLATTAGH TPLHIAAREG HVETVLALLE KEASQACMTK KGFTPLHVAA KYGKVRVAEL LLERDA HPN AAGKNGLTPL HVAVHHNNLD IVKLLLPRGG SPHSPAWNGY TPLHIAAKQN QVEVARSLLQ YGGSANAESV QGVTPLH LA AQEGHAEMVA LLLSKQANGN LGNKSGLTPL HLVAQEGHVP VADVLIKHGV MVDATTRMGY TPLHVASHYG NIKLVKFL L QHQADVNAKT KLGYSPLHQA AQQGHTDIVT LLLKNGASPN EVSSDGTTPL AIAKRLGYIS VTDVLKVVTD ETSFVLVSD KHRMSFPETV DEILDVSEDE GEELISFKAE RRDSRDVDEE KELLDFVPKL DQVVESPAIP RIPCAMPETV VIRSEEQEQA SKEYDEDSL IPSSPATETS DNISPVASPV HTGFLVSFMV DARGGSMRGS RHNGLRVVIP PRTCAAPTRI TCRLVKPQKL S TPPPLAEE EGLASRIIAL GPTGAQFLSP VIVEIPHFAS HGRGDRELVV LRSENGSVWK EHRSRYGESY LDQILNGMDE EL GSLEELE KKRVCRIITT DFPLYFVIMS RLCQDYDTIG PEGGSLKSKL VPLVQATFPE NAVTKRVKLA LQAQPVPDEL VTK LLGNQA TFSPIVTVEP RRRKFHRPIG LRIPLPPSWT DNPRDSGEGD TTSLRLLCSV IGGTDQAQWE DITGTTKLVY ANEC ANFTT NVSARFWLSD CPRTAEAVNF ATLLYKELTA VPYMAKFVIF AKMNDPREGR LRCYCMTDDK VDKTLEQHEN FVEVA RSRD IEVLEGMSLF AELSGNLVPV KKAAQQRSFH FQSFRENRLA MPVKVRDSSR EPGGSLSFLR KAMKYEDTQH ILCHLN ITM PPCAKGSGAE DRRRTPTPLA LRYSILSEST PGSLSGTEQA EMKMAVISEH LGLSWAELAR ELQFSVEDIN RIRVENP NS LLEQSVALLN LWVIREGQNA NMENLYTALQ SIDRGEIVNM LEGSGRQSRN LKPDRRHTDR DYSLSPSQMN GYSSLQDE L LSPASLGCAL SSPLRADQYW NEVAVLDAIP LAATEHDTML EMSDMQVWSA GLTPSLVTAE DSSLECSKAE DSDATGHEW KLEGALSEEP RGPELGSLEL VEDDTVDSDA TNGLIDLLEQ EEGQRSEEKL PGSKRQDDAT GAGQDSENEV SLVSGHQRGQ ARITHSPTV SQVTERSQDR LQDWDADGSI VSYLQDAAQG SWQEEVTQGP HSFQGTSTMT EGLEPGGSQE YEKVLVSVSE H TWTEQPEA ESSQADRDRR QQGQEEQVQE AKNTFTQVVQ GNEFQNIPGE QVTEEQFTDE QGNIVTKKII RKVVRQIDLS SA DAAQEHE EVTVEGPLED PSELEVDIDY FMKHSKDHTS TPNP UniProtKB: Ankyrin-1 |
-分子 #2: Band 3 anion transport protein
| 分子 | 名称: Band 3 anion transport protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 101.883859 KDa |
| 配列 | 文字列: MEELQDDYED MMEENLEQEE YEDPDIPESQ MEEPAAHDTE ATATDYHTTS HPGTHKVYVE LQELVMDEKN QELRWMEAAR WVQLEENLG ENGAWGRPHL SHLTFWSLLE LRRVFTKGTV LLDLQETSLA GVANQLLDRF IFEDQIRPQD REELLRALLL K HSHAGELE ...文字列: MEELQDDYED MMEENLEQEE YEDPDIPESQ MEEPAAHDTE ATATDYHTTS HPGTHKVYVE LQELVMDEKN QELRWMEAAR WVQLEENLG ENGAWGRPHL SHLTFWSLLE LRRVFTKGTV LLDLQETSLA GVANQLLDRF IFEDQIRPQD REELLRALLL K HSHAGELE ALGGVKPAVL TRSGDPSQPL LPQHSSLETQ LFCEQGDGGT EGHSPSGILE KIPPDSEATL VLVGRADFLE QP VLGFVRL QEAAELEAVE LPVPIRFLFV LLGPEAPHID YTQLGRAAAT LMSERVFRID AYMAQSRGEL LHSLEGFLDC SLV LPPTDA PSEQALLSLV PVQRELLRRR YQSSPAKPDS SFYKGLDLNG GPDDPLQQTG QLFGGLVRDI RRRYPYYLSD ITDA FSPQV LAAVIFIYFA ALSPAITFGG LLGEKTRNQM GVSELLISTA VQGILFALLG AQPLLVVGFS GPLLVFEEAF FSFCE TNGL EYIVGRVWIG FWLILLVVLV VAFEGSFLVR FISRYTQEIF SFLISLIFIY ETFSKLIKIF QDHPLQKTYN YNVLMV PKP QGPLPNTALL SLVLMAGTFF FAMMLRKFKN SSYFPGKLRR VIGDFGVPIS ILIMVLVDFF IQDTYTQKLS VPDGFKV SN SSARGWVIHP LGLRSEFPIW MMFASALPAL LVFILIFLES QITTLIVSKP ERKMVKGSGF HLDLLLVVGM GGVAALFG M PWLSATTVRS VTHANALTVM GKASTPGAAA QIQEVKEQRI SGLLVAVLVG LSILMEPILS RIPLAVLFGI FLYMGVTSL SGIQLFDRIL LLFKPPKYHP DVPYVKRVKT WRMHLFTGIQ IICLAVLWVV KSTPASLALP FVLILTVPLR RVLLPLIFRN VELQCLDAD DAKATFDEEE GRDEYDEVAM PV UniProtKB: Band 3 anion transport protein |
-分子 #3: Blood group Rh(CE) polypeptide
| 分子 | 名称: Blood group Rh(CE) polypeptide / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 45.598918 KDa |
| 配列 | 文字列: MSSKYPRSVR RCLPLWALTL EAALILLFYF FTHYDASLED QKGLVASYQV GQDLTVMAAL GLGFLTSNFR RHSWSSVAFN LFMLALGVQ WAILLDGFLS QFPPGKVVIT LFSIRLATMS AMSVLISAGA VLGKVNLAQL VVMVLVEVTA LGTLRMVISN I FNTDYHMN ...文字列: MSSKYPRSVR RCLPLWALTL EAALILLFYF FTHYDASLED QKGLVASYQV GQDLTVMAAL GLGFLTSNFR RHSWSSVAFN LFMLALGVQ WAILLDGFLS QFPPGKVVIT LFSIRLATMS AMSVLISAGA VLGKVNLAQL VVMVLVEVTA LGTLRMVISN I FNTDYHMN LRHFYVFAAY FGLTVAWCLP KPLPKGTEDN DQRATIPSLS AMLGALFLWM FWPSVNSPLL RSPIQRKNAM FN TYYALAV SVVTAISGSS LAHPQRKISM TYVHSAVLAG GVAVGTSCHL IPSPWLAMVL GLVAGLISIG GAKCLPVCCN RVL GIHHIS VMHSIFSLLG LLGEITYIVL LVLHTVWNGN GMIGFQVLLS IGELSLAIVI ALTSGLLTGL LLNLKIWKAP HVAK YFDDQ VFWKFPHLAV GF UniProtKB: Blood group Rh(CE) polypeptide |
-分子 #4: Ammonium transporter Rh type A
| 分子 | 名称: Ammonium transporter Rh type A / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 44.229629 KDa |
| 配列 | 文字列: MRFTFPLMAI VLEIAMIVLF GLFVEYETDQ TVLEQLNITK PTDMGIFFEL YPLFQDVHVM IFVGFGFLMT FLKKYGFSSV GINLLVAAL GLQWGTIVQG ILQSQGQKFN IGIKNMINAD FSAATVLISF GAVLGKTSPT QMLIMTILEI VFFAHNEYLV S EIFKASDI ...文字列: MRFTFPLMAI VLEIAMIVLF GLFVEYETDQ TVLEQLNITK PTDMGIFFEL YPLFQDVHVM IFVGFGFLMT FLKKYGFSSV GINLLVAAL GLQWGTIVQG ILQSQGQKFN IGIKNMINAD FSAATVLISF GAVLGKTSPT QMLIMTILEI VFFAHNEYLV S EIFKASDI GASMTIHAFG AYFGLAVAGI LYRSGLRKGH ENEESAYYSD LFAMIGTLFL WMFWPSFNSA IAEPGDKQCR AI VNTYFSL AACVLTAFAF SSLVEHRGKL NMVHIQNATL AGGVAVGTCA DMAIHPFGSM IIGSIAGMVS VLGYKFLTPL FTT KLRIHD TCGVHNLHGL PGVVGGLAGI VAVAMGASNT SMAMQAAALG SSIGTAVVGG LMTGLILKLP LWGQPSDQNC YDDS VYWKV PKTR UniProtKB: Ammonium transporter Rh type A |
-分子 #5: Protein 4.2
| 分子 | 名称: Protein 4.2 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 77.096914 KDa |
| 配列 | 文字列: MGQALGIKSC DFQAARNNEE HHTKALSSRR LFVRRGQPFT IILYFRAPVR AFLPALKKVA LTAQTGEQPS KINRTQATFP ISSLGDRKW WSAVVEERDA QSWTISVTTP ADAVIGHYSL LLQVSGRKQL LLGQFTLLFN PWNREDAVFL KNEAQRMEYL L NQNGLIYL ...文字列: MGQALGIKSC DFQAARNNEE HHTKALSSRR LFVRRGQPFT IILYFRAPVR AFLPALKKVA LTAQTGEQPS KINRTQATFP ISSLGDRKW WSAVVEERDA QSWTISVTTP ADAVIGHYSL LLQVSGRKQL LLGQFTLLFN PWNREDAVFL KNEAQRMEYL L NQNGLIYL GTADCIQAES WDFGQFEGDV IDLSLRLLSK DKQVEKWSQP VHVARVLGAL LHFLKEQRVL PTPQTQATQE GA LLNKRRG SVPILRQWLT GRGRPVYDGQ AWVLAAVACT VLRCLGIPAR VVTTFASAQG TGGRLLIDEY YNEEGLQNGE GQR GRIWIF QTSTECWMTR PALPQGYDGW QILHPSAPNG GGVLGSCDLV PVRAVKEGTL GLTPAVSDLF AAINASCVVW KCCE DGTLE LTDSNTKYVG NNISTKGVGS DRCEDITQNY KYPEGSLQEK EVLERVEKEK MEREKDNGIR PPSLETASPL YLLLK APSS LPLRGDAQIS VTLVNHSEQE KAVQLAIGVQ AVHYNGVLAA KLWRKKLHLT LSANLEKIIT IGLFFSNFER NPPENT FLR LTAMATHSES NLSCFAQEDI AICRPHLAIK MPEKAEQYQP LTASVSLQNS LDAPMEDCVI SILGRGLIHR ERSYRFR SV WPENTMCAKF QFTPTHVGLQ RLTVEVDCNM FQNLTNYKSV TVVAPELSA UniProtKB: Protein 4.2 |
-分子 #6: Glycophorin-B
| 分子 | 名称: Glycophorin-B / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 9.789674 KDa |
| 配列 | 文字列: MYGKIIFVLL LSEIVSISAL STTEVAMHTS TSSSVTKSYI SSQTNGETGQ LVHRFTVPAP VVIILIILCV MAGIIGTILL ISYSIRRLI KA UniProtKB: Glycophorin-B |
-分子 #7: Glycophorin-A
| 分子 | 名称: Glycophorin-A / タイプ: protein_or_peptide / ID: 7 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 16.348433 KDa |
| 配列 | 文字列: MYGKIIFVLL LSEIVSISAS STTGVAMHTS TSSSVTKSYI SSQTNDTHKR DTYAATPRAH EVSEISVRTV YPPEEETGER VQLAHHFSE PEITLIIFGV MAGVIGTILL ISYGIRRLIK KSPSDVKPLP SPDTDVPLSS VEIENPETSD Q UniProtKB: Glycophorin-A |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 130 mM KCl, 10 mM HEPES, pH 7.4 |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | Ghost membranes (~100 mg) were pelleted (6,000 x g, 10 minutes), washed 5 times in 130 mM KCl, 10 mM HEPES, pH 7.4, and then homogenized with the same buffer. The sample was sonicated using microprobe (amplitude = 30, pulse on = 5 sec, rest time = 10 sec, total pulse on = 50 sec). Large fragments are removed by centrifugation (6,000 x g, 10 minutes) and the supernatant was extruded (Avanti Polar Lipid) 10 times using a 400 nm filter. After extrusion, vesicles were collected by ultra-centrifugation at 35,000 rpm for 30 minutes (S120-AT3, Sorvall). The pellet, which contains vesicles, was resuspended at a final concentration of 5 mg/mL. All steps were performed at room temperature to facilitate vesicle formation. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 3.14 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 3.0 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Erythrocyte ankyrin-1 complex (Class1, 8CS9) was rigid fit as a single body to the map using fitmap in UCSF Chimera, after which waters, ligands and sidechains were removed from the model. |
| 精密化 | プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-8csl: |
 ムービー
ムービー コントローラー
コントローラー












































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)