+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human ankyrin complex (B2P1A1)2 | |||||||||
 マップデータ マップデータ | Cryo-EM structure of Ankyrin complex (B2P1A1)2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Red blood cell / Ankyrin complex / membrane protein / band 3 / protein 4.2 | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報spectrin-associated cytoskeleton / hemoglobin metabolic process / positive regulation of organelle organization / protein-glutamine gamma-glutamyltransferase activity / maintenance of epithelial cell apical/basal polarity / pH elevation / Defective SLC4A1 causes hereditary spherocytosis type 4 (HSP4), distal renal tubular acidosis (dRTA) and dRTA with hemolytic anemia (dRTA-HA) / negative regulation of urine volume / NrCAM interactions / Neurofascin interactions ...spectrin-associated cytoskeleton / hemoglobin metabolic process / positive regulation of organelle organization / protein-glutamine gamma-glutamyltransferase activity / maintenance of epithelial cell apical/basal polarity / pH elevation / Defective SLC4A1 causes hereditary spherocytosis type 4 (HSP4), distal renal tubular acidosis (dRTA) and dRTA with hemolytic anemia (dRTA-HA) / negative regulation of urine volume / NrCAM interactions / Neurofascin interactions / Bicarbonate transporters / intracellular monoatomic ion homeostasis / ankyrin-1 complex / CHL1 interactions / plasma membrane phospholipid scrambling / monoatomic anion transmembrane transporter activity / chloride:bicarbonate antiporter activity / cytoskeletal anchor activity / solute:inorganic anion antiporter activity / bicarbonate transport / monoatomic anion transport / bicarbonate transmembrane transporter activity / M band / Interaction between L1 and Ankyrins / chloride transport / chloride transmembrane transporter activity / ankyrin binding / negative regulation of glycolytic process through fructose-6-phosphate / hemoglobin binding / spectrin binding / cortical cytoskeleton / erythrocyte maturation / exocytosis / axolemma / endoplasmic reticulum to Golgi vesicle-mediated transport / erythrocyte development / COPI-mediated anterograde transport / protein-membrane adaptor activity / spleen development / chloride transmembrane transport / cytoskeleton organization / sarcoplasmic reticulum / protein localization to plasma membrane / regulation of intracellular pH / Erythrocytes take up oxygen and release carbon dioxide / cell morphogenesis / Erythrocytes take up carbon dioxide and release oxygen / sarcolemma / structural constituent of cytoskeleton / transmembrane transport / cytoplasmic side of plasma membrane / Z disc / multicellular organismal-level iron ion homeostasis / blood coagulation / regulation of cell shape / ATPase binding / basolateral plasma membrane / protein phosphatase binding / postsynaptic membrane / transmembrane transporter binding / blood microparticle / cytoskeleton / neuron projection / structural molecule activity / enzyme binding / signal transduction / protein homodimerization activity / extracellular exosome / ATP binding / membrane / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.5 Å | |||||||||
 データ登録者 データ登録者 | Xia X / Liu SH / Zhou ZH | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Structure, dynamics and assembly of the ankyrin complex on human red blood cell membrane. 著者: Xian Xia / Shiheng Liu / Z Hong Zhou /  要旨: The cytoskeleton of a red blood cell (RBC) is anchored to the cell membrane by the ankyrin complex. This complex is assembled during RBC genesis and comprises primarily band 3, protein 4.2 and ...The cytoskeleton of a red blood cell (RBC) is anchored to the cell membrane by the ankyrin complex. This complex is assembled during RBC genesis and comprises primarily band 3, protein 4.2 and ankyrin, whose mutations contribute to numerous human inherited diseases. High-resolution structures of the ankyrin complex have been long sought-after to understand its assembly and disease-causing mutations. Here, we analyzed native complexes on the human RBC membrane by stepwise fractionation. Cryo-electron microscopy structures of nine band-3-associated complexes reveal that protein 4.2 stabilizes the cytoplasmic domain of band 3 dimer. In turn, the superhelix-shaped ankyrin binds to this protein 4.2 via ankyrin repeats (ARs) 6-13 and to another band 3 dimer via ARs 17-20, bridging two band 3 dimers in the ankyrin complex. Integration of these structures with both prior data and our biochemical data supports a model of ankyrin complex assembly during erythropoiesis and identifies interactions essential for the mechanical stability of RBC. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26154.map.gz emd_26154.map.gz | 41.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26154-v30.xml emd-26154-v30.xml emd-26154.xml emd-26154.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26154.png emd_26154.png | 46 KB | ||
| Filedesc metadata |  emd-26154.cif.gz emd-26154.cif.gz | 6.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26154 http://ftp.pdbj.org/pub/emdb/structures/EMD-26154 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26154 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26154 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26154_validation.pdf.gz emd_26154_validation.pdf.gz | 406.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26154_full_validation.pdf.gz emd_26154_full_validation.pdf.gz | 406.3 KB | 表示 | |
| XML形式データ |  emd_26154_validation.xml.gz emd_26154_validation.xml.gz | 6.6 KB | 表示 | |
| CIF形式データ |  emd_26154_validation.cif.gz emd_26154_validation.cif.gz | 7.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26154 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26154 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26154 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26154 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26154.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26154.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of Ankyrin complex (B2P1A1)2 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.2 Å | ||||||||||||||||||||||||||||||||||||
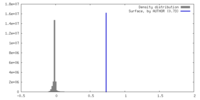
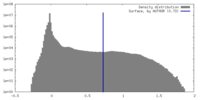
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : High-salt fraction 2 from human red blood cell membrane
| 全体 | 名称: High-salt fraction 2 from human red blood cell membrane |
|---|---|
| 要素 |
|
-超分子 #1: High-salt fraction 2 from human red blood cell membrane
| 超分子 | 名称: High-salt fraction 2 from human red blood cell membrane タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1 MDa |
-分子 #1: band 3
| 分子 | 名称: band 3 / タイプ: protein_or_peptide / ID: 1 / 詳細: four band 3 in the complex / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEELQDDYED MMEENLEQEE YEDPDIPESQ MEEPAAHDTE ATATDYHTTS HPGTHKVYVE LQELVMDEKN QELRWMEAAR WVQLEENLGE NGAWGRPHLS HLTFWSLLEL RRVFTKGTVL LDLQETSLAG VANQLLDRFI FEDQIRPQDR EELLRALLLK HSHAGELEAL ...文字列: MEELQDDYED MMEENLEQEE YEDPDIPESQ MEEPAAHDTE ATATDYHTTS HPGTHKVYVE LQELVMDEKN QELRWMEAAR WVQLEENLGE NGAWGRPHLS HLTFWSLLEL RRVFTKGTVL LDLQETSLAG VANQLLDRFI FEDQIRPQDR EELLRALLLK HSHAGELEAL GGVKPAVLTR SGDPSQPLLP QHSSLETQLF CEQGDGGTEG HSPSGILEKI PPDSEATLVL VGRADFLEQP VLGFVRLQEA AELEAVELPV PIRFLFVLLG PEAPHIDYTQ LGRAAATLMS ERVFRIDAYM AQSRGELLHS LEGFLDCSLV LPPTDAPSEQ ALLSLVPVQR ELLRRRYQSS PAKPDSSFYK GLDLNGGPDD PLQQTGQLFG GLVRDIRRRY PYYLSDITDA FSPQVLAAVI FIYFAALSPA ITFGGLLGEK TRNQMGVSEL LISTAVQGIL FALLGAQPLL VVGFSGPLLV FEEAFFSFCE TNGLEYIVGR VWIGFWLILL VVLVVAFEGS FLVRFISRYT QEIFSFLISL IFIYETFSKL IKIFQDHPLQ KTYNYNVLMV PKPQGPLPNT ALLSLVLMAG TFFFAMMLRK FKNSSYFPGK LRRVIGDFGV PISILIMVLV DFFIQDTYTQ KLSVPDGFKV SNSSARGWVI HPLGLRSEFP IWMMFASALP ALLVFILIFL ESQITTLIVS KPERKMVKGS GFHLDLLLVV GMGGVAALFG MPWLSATTVR SVTHANALTV MGKASTPGAA AQIQEVKEQR ISGLLVAVLV GLSILMEPIL SRIPLAVLFG IFLYMGVTSL SGIQLFDRIL LLFKPPKYHP DVPYVKRVKT WRMHLFTGIQ IICLAVLWVV KSTPASLALP FVLILTVPLR RVLLPLIFRN VELQCLDADD AKATFDEEEG RDEYDEVAMP V UniProtKB: Band 3 anion transport protein |
-分子 #2: protein 4.2
| 分子 | 名称: protein 4.2 / タイプ: protein_or_peptide / ID: 2 / 詳細: two protein 4.2 in the complex / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGQALGIKSC DFQAARNNEE HHTKALSSRR LFVRRGQPFT IILYFRAPVR AFLPALKKVA LTAQTGEQPS KINRTQATFP ISSLGDRKWW SAVVEERDAQ SWTISVTTPA DAVIGHYSLL LQVSGRKQLL LGQFTLLFNP WNREDAVFLK NEAQRMEYLL NQNGLIYLGT ...文字列: MGQALGIKSC DFQAARNNEE HHTKALSSRR LFVRRGQPFT IILYFRAPVR AFLPALKKVA LTAQTGEQPS KINRTQATFP ISSLGDRKWW SAVVEERDAQ SWTISVTTPA DAVIGHYSLL LQVSGRKQLL LGQFTLLFNP WNREDAVFLK NEAQRMEYLL NQNGLIYLGT ADCIQAESWD FGQFEGDVID LSLRLLSKDK QVEKWSQPVH VARVLGALLH FLKEQRVLPT PQTQATQEGA LLNKRRGSVP ILRQWLTGRG RPVYDGQAWV LAAVACTVLR CLGIPARVVT TFASAQGTGG RLLIDEYYNE EGLQNGEGQR GRIWIFQTST ECWMTRPALP QGYDGWQILH PSAPNGGGVL GSCDLVPVRA VKEGTLGLTP AVSDLFAAIN ASCVVWKCCE DGTLELTDSN TKYVGNNIST KGVGSDRCED ITQNYKYPEG SLQEKEVLER VEKEKMEREK DNGIRPPSLE TASPLYLLLK APSSLPLRGD AQISVTLVNH SEQEKAVQLA IGVQAVHYNG VLAAKLWRKK LHLTLSANLE KIITIGLFFS NFERNPPENT FLRLTAMATH SESNLSCFAQ EDIAICRPHL AIKMPEKAEQ YQPLTASVSL QNSLDAPMED CVISILGRGL IHRERSYRFR SVWPENTMCA KFQFTPTHVG LQRLTVEVDC NMFQNLTNYK SVTVVAPELS A UniProtKB: Protein 4.2 |
-分子 #3: ankyrin
| 分子 | 名称: ankyrin / タイプ: protein_or_peptide / ID: 3 / 詳細: two ankyrin in the complex / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MPYSVGFREA DAATSFLRAA RSGNLDKALD HLRNGVDINT CNQNGLNGLH LASKEGHVKM VVELLHKEII LETTTKKGNT ALHIAALAGQ DEVVRELVNY GANVNAQSQK GFTPLYMAAQ ENHLEVVKFL LENGANQNVA TEDGFTPLAV ALQQGHENVV AHLINYGTKG ...文字列: MPYSVGFREA DAATSFLRAA RSGNLDKALD HLRNGVDINT CNQNGLNGLH LASKEGHVKM VVELLHKEII LETTTKKGNT ALHIAALAGQ DEVVRELVNY GANVNAQSQK GFTPLYMAAQ ENHLEVVKFL LENGANQNVA TEDGFTPLAV ALQQGHENVV AHLINYGTKG KVRLPALHIA ARNDDTRTAA VLLQNDPNPD VLSKTGFTPL HIAAHYENLN VAQLLLNRGA SVNFTPQNGI TPLHIASRRG NVIMVRLLLD RGAQIETKTK DELTPLHCAA RNGHVRISEI LLDHGAPIQA KTKNGLSPIH MAAQGDHLDC VRLLLQYDAE IDDITLDHLT PLHVAAHCGH HRVAKVLLDK GAKPNSRALN GFTPLHIACK KNHVRVMELL LKTGASIDAV TESGLTPLHV ASFMGHLPIV KNLLQRGASP NVSNVKVETP LHMAARAGHT EVAKYLLQNK AKVNAKAKDD QTPLHCAARI GHTNMVKLLL ENNANPNLAT TAGHTPLHIA AREGHVETVL ALLEKEASQA CMTKKGFTPL HVAAKYGKVR VAELLLERDA HPNAAGKNGL TPLHVAVHHN NLDIVKLLLP RGGSPHSPAW NGYTPLHIAA KQNQVEVARS LLQYGGSANA ESVQGVTPLH LAAQEGHAEM VALLLSKQAN GNLGNKSGLT PLHLVAQEGH VPVADVLIKH GVMVDATTRM GYTPLHVASH YGNIKLVKFL LQHQADVNAK TKLGYSPLHQ AAQQGHTDIV TLLLKNGASP NEVSSDGTTP LAIAKRLGYI SVTDVLKVVT DETSFVLVSD KHRMSFPETV DEILDVSEDE GEELISFKAE RRDSRDVDEE KELLDFVPKL DQVVESPAIP RIPCAMPETV VIRSEEQEQA SKEYDEDSLI PSSPATETSD NISPVASPVH TGFLVSFMVD ARGGSMRGSR HNGLRVVIPP RTCAAPTRIT CRLVKPQKLS TPPPLAEEEG LASRIIALGP TGAQFLSPVI VEIPHFASHG RGDRELVVLR SENGSVWKEH RSRYGESYLD QILNGMDEEL GSLEELEKKR VCRIITTDFP LYFVIMSRLC QDYDTIGPEG GSLKSKLVPL VQATFPENAV TKRVKLALQA QPVPDELVTK LLGNQATFSP IVTVEPRRRK FHRPIGLRIP LPPSWTDNPR DSGEGDTTSL RLLCSVIGGT DQAQWEDITG TTKLVYANEC ANFTTNVSAR FWLSDCPRTA EAVNFATLLY KELTAVPYMA KFVIFAKMND PREGRLRCYC MTDDKVDKTL EQHENFVEVA RSRDIEVLEG MSLFAELSGN LVPVKKAAQQ RSFHFQSFRE NRLAMPVKVR DSSREPGGSL SFLRKAMKYE DTQHILCHLN ITMPPCAKGS GAEDRRRTPT PLALRYSILS ESTPGSLSGT EQAEMKMAVI SEHLGLSWAE LARELQFSVE DINRIRVENP NSLLEQSVAL LNLWVIREGQ NANMENLYTA LQSIDRGEIV NMLEGSGRQS RNLKPDRRHT DRDYSLSPSQ MNGYSSLQDE LLSPASLGCA LSSPLRADQY WNEVAVLDAI PLAATEHDTM LEMSDMQVWS AGLTPSLVTA EDSSLECSKA EDSDATGHEW KLEGALSEEP RGPELGSLEL VEDDTVDSDA TNGLIDLLEQ EEGQRSEEKL PGSKRQDDAT GAGQDSENEV SLVSGHQRGQ ARITHSPTVS QVTERSQDRL QDWDADGSIV SYLQDAAQGS WQEEVTQGPH SFQGTSTMTE GLEPGGSQEY EKVLVSVSEH TWTEQPEAES SQADRDRRQQ GQEEQVQEAK NTFTQVVQGN EFQNIPGEQV TEEQFTDEQG NIVTKKIIRK VVRQIDLSSA DAAQEHEEVT VEGPLEDPSE LEVDIDYFMK HSKDHTSTPN P UniProtKB: Ankyrin-1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 100.0 K / 最高: 100.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 21187 / 平均露光時間: 2.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|
 ムービー
ムービー コントローラー
コントローラー




































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)