+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Coxsackievirus A21 capsid subdomain in complex with mouse polyclonal antibody pAbC-2 | |||||||||
 マップデータ マップデータ | cryoEM map of the subdomain of CV-A21 viral particle in complex with mouse polyclonal antibody pAbC-2 - Main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | polyclonal antibodies / Immunology / viral particles / VIRUS-IMMUNE SYSTEM complex | |||||||||
| 生物種 |  Coxsackievirus A21 (コクサッキーウイルス) / Coxsackievirus A21 (コクサッキーウイルス) /  | |||||||||
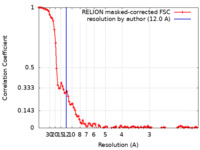
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 12.0 Å | |||||||||
 データ登録者 データ登録者 | Antanasijevic A / Ward AB | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: PNAS Nexus / 年: 2022 ジャーナル: PNAS Nexus / 年: 2022タイトル: High-resolution structural analysis of enterovirus-reactive polyclonal antibodies in complex with whole virions. 著者: Aleksandar Antanasijevic / Autumn J Schulze / Vijay S Reddy / Andrew B Ward /  要旨: Non-polio enteroviruses (NPEVs) cause serious illnesses in young children and neonates, including aseptic meningitis, encephalitis, and inflammatory muscle disease, among others. While over 100 ...Non-polio enteroviruses (NPEVs) cause serious illnesses in young children and neonates, including aseptic meningitis, encephalitis, and inflammatory muscle disease, among others. While over 100 serotypes have been described to date, vaccine only exists for EV-A71. Efforts toward rationally designed pan-NPEV vaccines would greatly benefit from structural biology methods for rapid and comprehensive evaluation of vaccine candidates and elicited antibody responses. Toward this goal, we introduced a cryo-electron-microscopy-based approach for structural analysis of virus- or vaccine-elicited polyclonal antibodies (pAbs) in complex with whole NPEV virions. We demonstrated the feasibility using coxsackievirus A21 and reconstructed five structurally distinct pAbs bound to the virus. The pAbs targeted two immunodominant epitopes, one overlapping with the receptor binding site. These results demonstrate that our method can be applied to map broad-spectrum polyclonal immune responses against intact virions and define potentially cross-reactive epitopes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26069.map.gz emd_26069.map.gz | 76.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26069-v30.xml emd-26069-v30.xml emd-26069.xml emd-26069.xml | 21.3 KB 21.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26069_fsc.xml emd_26069_fsc.xml | 10.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26069.png emd_26069.png | 79.7 KB | ||
| マスクデータ |  emd_26069_msk_1.map emd_26069_msk_1.map | 83.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-26069.cif.gz emd-26069.cif.gz | 6 KB | ||
| その他 |  emd_26069_half_map_1.map.gz emd_26069_half_map_1.map.gz emd_26069_half_map_2.map.gz emd_26069_half_map_2.map.gz | 65.2 MB 65.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26069 http://ftp.pdbj.org/pub/emdb/structures/EMD-26069 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26069 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26069 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26069_validation.pdf.gz emd_26069_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26069_full_validation.pdf.gz emd_26069_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_26069_validation.xml.gz emd_26069_validation.xml.gz | 16.8 KB | 表示 | |
| CIF形式データ |  emd_26069_validation.cif.gz emd_26069_validation.cif.gz | 22.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26069 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26069 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26069 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26069 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26069.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26069.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM map of the subdomain of CV-A21 viral particle in complex with mouse polyclonal antibody pAbC-2 - Main map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.045 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_26069_msk_1.map emd_26069_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
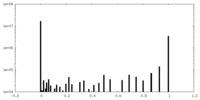
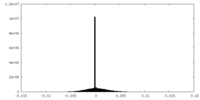
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: cryoEM map of the subdomain of CV-A21 viral...
| ファイル | emd_26069_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM map of the subdomain of CV-A21 viral particle in complex with mouse polyclonal antibody pAbC-2 - Half-map 1 | ||||||||||||
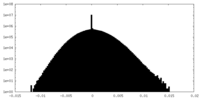
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: cryoEM map of the subdomain of CV-A21 viral...
| ファイル | emd_26069_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoEM map of the subdomain of CV-A21 viral particle in complex with mouse polyclonal antibody pAbC-2 - Half-map 2 | ||||||||||||
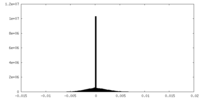
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Immune complex featuring subdomains of coxsackievirus A21 (CV-A21...
| 全体 | 名称: Immune complex featuring subdomains of coxsackievirus A21 (CV-A21) viral particles in complex with mouse polyclonal antibodies |
|---|---|
| 要素 |
|
-超分子 #1: Immune complex featuring subdomains of coxsackievirus A21 (CV-A21...
| 超分子 | 名称: Immune complex featuring subdomains of coxsackievirus A21 (CV-A21) viral particles in complex with mouse polyclonal antibodies タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The complexes were assembled by combining purified CV-A21 particles and polyclonal antibodies isolated from mice (as Fab fragments) |
|---|---|
| 由来(天然) | 生物種:  Coxsackievirus A21 (コクサッキーウイルス) Coxsackievirus A21 (コクサッキーウイルス) |
-分子 #1: Coxsackievirus capsid - Polyprotein
| 分子 | 名称: Coxsackievirus capsid - Polyprotein / タイプ: protein_or_peptide / ID: 1 詳細: CV-A21 capsid polyprotein sequence is provided. The sequence includes VP1-4. 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Coxsackievirus A21 (コクサッキーウイルス) Coxsackievirus A21 (コクサッキーウイルス) |
| 配列 | 文字列: MGAQVSTQKT GAHENQNVAA NGSTINYTTI NYYKDSASNS ATRQDLSQDP SKFTEPVKDL MLKTAPALNS PNVEACGYSD RVRQITLGNS TITTQEAANA IVAYGEWPTY INDSEANPVD APTEPDVSSN RFYTLESVSW KTTSRGWWWK LPDCLKDMGM FGQNMYYHYL ...文字列: MGAQVSTQKT GAHENQNVAA NGSTINYTTI NYYKDSASNS ATRQDLSQDP SKFTEPVKDL MLKTAPALNS PNVEACGYSD RVRQITLGNS TITTQEAANA IVAYGEWPTY INDSEANPVD APTEPDVSSN RFYTLESVSW KTTSRGWWWK LPDCLKDMGM FGQNMYYHYL GRSGYTIHVQ CNASKFHQGA LGVFLIPEFV MACNTESKTS YVSYINANPG ERGGEFTNTY NPSNTDASEG RKFAALDYLL GSGVLAGNAF VYPHQIINLR TNNSATIVVP YVNSLVIDCM AKHNNWGIVI LPLAPLAFAA TSSPQVPITV TIAPMCTEFN GLRNITVPVH QGLPTMNTPG SNQFLTSDDF QSPCALPNFD VTPPIHIPGE VKNMMELAEI DTLIPMNAVD GKVNTMEMYQ IPLNDNLSKA PIFCLSLSPA SDKRLSHTML GEILNYYTHW TGSIRFTFLF CGSMMATGKL LLSYSPPGAK PPTNRKDAML GTHIIWDLGL QSSCSMVAPW ISNTVYRRCA RDDFTEGGFI TCFYQTRIVV PASTPTSMFM LGFVSACPDF SVRLLRDTPH ISQSKLIGRT QGIEDLIDTA IKNALRVSQP PSTQSTEATS GVNSQEVPAL TAVETGASGQ AIPSDVVETR HVVNYKTRSE SCLESFFGRA ACVTILSLTN SSKSGEEKKH FNIWNITYTD TVQLRRKLEF FTYSRFDLEM TFVFTENYPS TASGEVRNQV YQIMYIPPGA PRPSSWDDYT WQSSSNPSIF YMYGNAPPRM SIPYVGIANA YSHFYDGFAR VPLEGENTDA GDTFYGLVSI NDFGVLAVRA VNRSNPHTIH TSVRVYMKPK HIRCWCPRPP RAVLYRGEGV DMISSAILPL AKVDSITTF |
-分子 #2: pAbC-2
| 分子 | 名称: pAbC-2 / タイプ: protein_or_peptide / ID: 2 / 詳細: Polyclonal antibody pAbC-2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: TBS | |||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot time varied: 3-5s. | |||||||||
| 詳細 | Immune complexes were SEC-purified, concentrated and applied onto cryo-grids. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 2 / 実像数: 3862 / 平均露光時間: 9.0 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X