+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | TnsBctd-TnsC-TniQ complex | |||||||||
 マップデータ マップデータ | LocSpiral processed map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CAST / transposase / AAA+ ATPase / AAA+ / CRISPR / Cas / DNA BINDING PROTEIN-DNA complex | |||||||||
| 生物種 |  [Scytonema hofmanni] UTEX 2349 (バクテリア) / synthetic construct (人工物) [Scytonema hofmanni] UTEX 2349 (バクテリア) / synthetic construct (人工物) | |||||||||
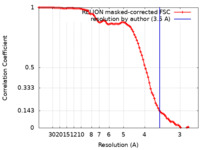
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Park J / Tsai AWT | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Structures of the holo CRISPR RNA-guided transposon integration complex. 著者: Jung-Un Park / Amy Wei-Lun Tsai / Alexandrea N Rizo / Vinh H Truong / Tristan X Wellner / Richard D Schargel / Elizabeth H Kellogg /  要旨: CRISPR-associated transposons (CAST) are programmable mobile genetic elements that insert large DNA cargos using an RNA-guided mechanism. CAST elements contain multiple conserved proteins: a CRISPR ...CRISPR-associated transposons (CAST) are programmable mobile genetic elements that insert large DNA cargos using an RNA-guided mechanism. CAST elements contain multiple conserved proteins: a CRISPR effector (Cas12k or Cascade), a AAA+ regulator (TnsC), a transposase (TnsA-TnsB) and a target-site-associated factor (TniQ). These components are thought to cooperatively integrate DNA via formation of a multisubunit transposition integration complex (transpososome). Here we reconstituted the approximately 1 MDa type V-K CAST transpososome from Scytonema hofmannii (ShCAST) and determined its structure using single-particle cryo-electon microscopy. The architecture of this transpososome reveals modular association between the components. Cas12k forms a complex with ribosomal subunit S15 and TniQ, stabilizing formation of a full R-loop. TnsC has dedicated interaction interfaces with TniQ and TnsB. Of note, we observe TnsC-TnsB interactions at the C-terminal face of TnsC, which contribute to the stimulation of ATPase activity. Although the TnsC oligomeric assembly deviates slightly from the helical configuration found in isolation, the TnsC-bound target DNA conformation differs markedly in the transpososome. As a consequence, TnsC makes new protein-DNA interactions throughout the transpososome that are important for transposition activity. Finally, we identify two distinct transpososome populations that differ in their DNA contacts near TniQ. This suggests that associations with the CRISPR effector can be flexible. This ShCAST transpososome structure enhances our understanding of CAST transposition systems and suggests ways to improve CAST transposition for precision genome-editing applications. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25453.map.gz emd_25453.map.gz | 5.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25453-v30.xml emd-25453-v30.xml emd-25453.xml emd-25453.xml | 26 KB 26 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25453_fsc.xml emd_25453_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25453.png emd_25453.png | 82.7 KB | ||
| マスクデータ |  emd_25453_msk_1.map emd_25453_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-25453.cif.gz emd-25453.cif.gz | 6.5 KB | ||
| その他 |  emd_25453_additional_1.map.gz emd_25453_additional_1.map.gz emd_25453_additional_2.map.gz emd_25453_additional_2.map.gz emd_25453_half_map_1.map.gz emd_25453_half_map_1.map.gz emd_25453_half_map_2.map.gz emd_25453_half_map_2.map.gz | 49.5 MB 5.6 MB 49.7 MB 49.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25453 http://ftp.pdbj.org/pub/emdb/structures/EMD-25453 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25453 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25453 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25453_validation.pdf.gz emd_25453_validation.pdf.gz | 1002.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25453_full_validation.pdf.gz emd_25453_full_validation.pdf.gz | 1001.8 KB | 表示 | |
| XML形式データ |  emd_25453_validation.xml.gz emd_25453_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_25453_validation.cif.gz emd_25453_validation.cif.gz | 21.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25453 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25453 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25453 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25453 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25453.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25453.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LocSpiral processed map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.33 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
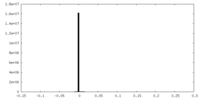
-マスク #1
| ファイル |  emd_25453_msk_1.map emd_25453_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
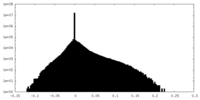
-追加マップ: Full map
| ファイル | emd_25453_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Full map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: uniform B-factor sharpened map
| ファイル | emd_25453_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | uniform B-factor sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
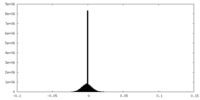
-ハーフマップ: halfmap1
| ファイル | emd_25453_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
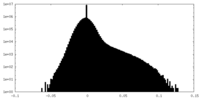
-ハーフマップ: halfmap 2
| ファイル | emd_25453_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmap 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ATP-bound TnsBctd-TnsC-TniQ complex from ShCAST element
| 全体 | 名称: ATP-bound TnsBctd-TnsC-TniQ complex from ShCAST element |
|---|---|
| 要素 |
|
-超分子 #1: ATP-bound TnsBctd-TnsC-TniQ complex from ShCAST element
| 超分子 | 名称: ATP-bound TnsBctd-TnsC-TniQ complex from ShCAST element タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  [Scytonema hofmanni] UTEX 2349 (バクテリア) [Scytonema hofmanni] UTEX 2349 (バクテリア) |
| 分子量 | 理論値: 400 KDa |
-分子 #1: DNA (28-MER)
| 分子 | 名称: DNA (28-MER) / タイプ: dna / ID: 1 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 8.472441 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) |
-分子 #2: DNA (29-MER)
| 分子 | 名称: DNA (29-MER) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 9.03804 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) |
-分子 #3: TnsC
| 分子 | 名称: TnsC / タイプ: protein_or_peptide / ID: 3 / コピー数: 11 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  [Scytonema hofmanni] UTEX 2349 (バクテリア) [Scytonema hofmanni] UTEX 2349 (バクテリア) |
| 分子量 | 理論値: 31.444617 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTEAQAIAKQ LGGVKPDDEW LQAEIARLKG KSIVPLQQVK TLHDWLDGKR KARKSCRVVG ESRTGKTVAC DAYRYRHKPQ QEAGRPPTV PVVYIRPHQK CGPKDLFKKI TEYLKYRVTK GTVSDFRDRT IEVLKGCGVE MLIIDEADRL KPETFADVRD I AEDLGIAV ...文字列: MTEAQAIAKQ LGGVKPDDEW LQAEIARLKG KSIVPLQQVK TLHDWLDGKR KARKSCRVVG ESRTGKTVAC DAYRYRHKPQ QEAGRPPTV PVVYIRPHQK CGPKDLFKKI TEYLKYRVTK GTVSDFRDRT IEVLKGCGVE MLIIDEADRL KPETFADVRD I AEDLGIAV VLVGTDRLDA VIKRDEQVLE RFRAHLRFGK LSGEDFKNTV EMWEQMVLKL PVSSNLKSKE MLRILTSATE GY IGRLDEI LREAAIRSLS RGLKKIDKAV LQEVAKEYK |
-分子 #4: TnsB-CTD
| 分子 | 名称: TnsB-CTD / タイプ: protein_or_peptide / ID: 4 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  [Scytonema hofmanni] UTEX 2349 (バクテリア) [Scytonema hofmanni] UTEX 2349 (バクテリア) |
| 分子量 | 理論値: 1.97711 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: IEVWDYEQLR EEYGF |
-分子 #5: TniQ
| 分子 | 名称: TniQ / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  [Scytonema hofmanni] UTEX 2349 (バクテリア) [Scytonema hofmanni] UTEX 2349 (バクテリア) |
| 分子量 | 理論値: 19.01124 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MIEAPDVKPW LFLIKPYEGE SLSHFLGRFR RANHLSASGL GTLAGIGAIV ARWERFHFNP RPSQQELEAI ASVVEVDAQR LAQMLPPAG VGMQHEPIRL CGACYAESPC HRIEWQYKSV WKCDRHQLKI LAKCPNCQAP FKMPALWEDG CCHRCRMPFA E MAKLQKV |
-分子 #6: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 6 / コピー数: 11 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #7: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 7 / コピー数: 11 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.2 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7svu: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)