+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-25144 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of positive allosteric modulator-free active human calcium-sensing receptor | |||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
| 機能・相同性 | Isoform 1 of Extracellular calcium-sensing receptor 機能・相同性情報 機能・相同性情報 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Park J / Zuo H / Frangaj A / Fu Z / Yen LY / Zhang Z / Mosyak L / Slavkovich VN / Liu J / Ray KM ...Park J / Zuo H / Frangaj A / Fu Z / Yen LY / Zhang Z / Mosyak L / Slavkovich VN / Liu J / Ray KM / Cao B / Vallese F / Geng Y / Chen S / Grassucci R / Dandey VP / Tan YZ / Eng E / Lee Y / Kloss B / Liu Z / Hendrickson WA / Potter CS / Carragher B / Graziano J / Conigrave AD / Frank J / Clarke OB / Fan QR | |||||||||||||||||||||
| 資金援助 |  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: Symmetric activation and modulation of the human calcium-sensing receptor. 著者: Jinseo Park / Hao Zuo / Aurel Frangaj / Ziao Fu / Laura Y Yen / Zhening Zhang / Lidia Mosyak / Vesna N Slavkovich / Jonathan Liu / Kimberly M Ray / Baohua Cao / Francesca Vallese / Yong Geng ...著者: Jinseo Park / Hao Zuo / Aurel Frangaj / Ziao Fu / Laura Y Yen / Zhening Zhang / Lidia Mosyak / Vesna N Slavkovich / Jonathan Liu / Kimberly M Ray / Baohua Cao / Francesca Vallese / Yong Geng / Shaoxia Chen / Robert Grassucci / Venkata P Dandey / Yong Zi Tan / Edward Eng / Yeji Lee / Brian Kloss / Zheng Liu / Wayne A Hendrickson / Clinton S Potter / Bridget Carragher / Joseph Graziano / Arthur D Conigrave / Joachim Frank / Oliver B Clarke / Qing R Fan /     要旨: The human extracellular calcium-sensing (CaS) receptor controls plasma Ca levels and contributes to nutrient-dependent maintenance and metabolism of diverse organs. Allosteric modulation of the CaS ...The human extracellular calcium-sensing (CaS) receptor controls plasma Ca levels and contributes to nutrient-dependent maintenance and metabolism of diverse organs. Allosteric modulation of the CaS receptor corrects disorders of calcium homeostasis. Here, we report the cryogenic-electron microscopy reconstructions of a near-full-length CaS receptor in the absence and presence of allosteric modulators. Activation of the homodimeric CaS receptor requires a break in the transmembrane 6 (TM6) helix of each subunit, which facilitates the formation of a TM6-mediated homodimer interface and expansion of homodimer interactions. This transformation in TM6 occurs without a positive allosteric modulator. Two modulators with opposite functional roles bind to overlapping sites within the transmembrane domain through common interactions, acting to stabilize distinct rotamer conformations of key residues on the TM6 helix. The positive modulator reinforces TM6 distortion and maximizes subunit contact to enhance receptor activity, while the negative modulator strengthens an intact TM6 to dampen receptor function. In both active and inactive states, the receptor displays symmetrical transmembrane conformations that are consistent with its homodimeric assembly. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25144.map.gz emd_25144.map.gz | 9.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25144-v30.xml emd-25144-v30.xml emd-25144.xml emd-25144.xml | 22.7 KB 22.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_25144.png emd_25144.png | 116.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25144 http://ftp.pdbj.org/pub/emdb/structures/EMD-25144 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25144 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25144 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25144_validation.pdf.gz emd_25144_validation.pdf.gz | 354.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25144_full_validation.pdf.gz emd_25144_full_validation.pdf.gz | 354.4 KB | 表示 | |
| XML形式データ |  emd_25144_validation.xml.gz emd_25144_validation.xml.gz | 4.2 KB | 表示 | |
| CIF形式データ |  emd_25144_validation.cif.gz emd_25144_validation.cif.gz | 4.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25144 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25144 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25144 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25144 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7simMC  7silC  7sinC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10834 (タイトル: Structures of positive allosteric modulator-bound and unbound active human calcium-sensing receptor EMPIAR-10834 (タイトル: Structures of positive allosteric modulator-bound and unbound active human calcium-sensing receptorData size: 3.8 TB Data #1: Unaligned multi-frame micrographs of calcium-sensing receptor in both PAM-bound and unbound active states [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25144.map.gz / 形式: CCP4 / 大きさ: 18.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25144.map.gz / 形式: CCP4 / 大きさ: 18.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Homodimer human calcium sensing receptor
| 全体 | 名称: Homodimer human calcium sensing receptor |
|---|---|
| 要素 |
|
-超分子 #1: Homodimer human calcium sensing receptor
| 超分子 | 名称: Homodimer human calcium sensing receptor / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK 293 GnTl- / 組換プラスミド: modified bacMam Homo sapiens (ヒト) / 組換細胞: HEK 293 GnTl- / 組換プラスミド: modified bacMam |
| 分子量 | 理論値: 192 KDa |
-分子 #1: Isoform 1 of Extracellular calcium-sensing receptor
| 分子 | 名称: Isoform 1 of Extracellular calcium-sensing receptor / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 99.299461 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAFYSCCWVL LALTWHTSAY GPDQRAQKKG DIILGGLFPI HFGVAAKDQD LKSRPESVEC IRYNFRGFRW LQAMIFAIEE INSSPALLP NLTLGYRIFD TCNTVSKALE ATLSFVAQNK IDSLNLDEFC NCSEHIPSTI AVVGATGSGV STAVANLLGL F YIPQVSYA ...文字列: MAFYSCCWVL LALTWHTSAY GPDQRAQKKG DIILGGLFPI HFGVAAKDQD LKSRPESVEC IRYNFRGFRW LQAMIFAIEE INSSPALLP NLTLGYRIFD TCNTVSKALE ATLSFVAQNK IDSLNLDEFC NCSEHIPSTI AVVGATGSGV STAVANLLGL F YIPQVSYA SSSRLLSNKN QFKSFLRTIP NDEHQATAMA DIIEYFRWNW VGTIAADDDY GRPGIEKFRE EAEERDICID FS ELISQYS DEEEIQHVVE VIQNSTAKVI VVFSSGPDLE PLIKEIVRRN ITGKIWLASE AWASSSLIAM PQYFHVVGGT IGF ALKAGQ IPGFREFLKK VHPRKSVHNG FAKEFWEETF NCHLQEGAKG PLPVDTFLRG HEESGDRFSN SSTAFRPLCT GDEN ISSVE TPYIDYTHLR ISYNVYLAVY SIAHALQDIY TCLPGRGLFT NGSCADIKKV EAWQVLKHLR HLNFTNNMGE QVTFD ECGD LVGNYSIINW HLSPEDGSIV FKEVGYYNVY AKKGERLFIN EEKILWSGFS REVPFSNCSR DCLAGTRKGI IEGEPT CCF ECVECPDGEY SDETDASACN KCPDDFWSNE NHTSCIAKEI EFLSWTEPFG IALTLFAVLG IFLTAFVLGV FIKFRNT PI VKATNRELSY LLLFSLLCCF SSSLFFIGEP QDWTCRLRQP AFGISFVLCI SCILVKTNRV LLVFEAKIPT SFHRKWWG L NLQFLLVFLC TFMQIVICVI WLYTAPPSSY RNQELEDEII FITCHEGSLM ALGFLIGYTC LLAAICFFFA FKSRKLPEN FNEAKFITFS MLIFFIVWIS FIPAYASTYG KFVSAVEVIA ILAASFGLLA CIFFNKIYII LFKPSRNTIE DYKDDDDK |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 2 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #4: CYCLOMETHYLTRYPTOPHAN
| 分子 | 名称: CYCLOMETHYLTRYPTOPHAN / タイプ: ligand / ID: 4 / コピー数: 2 / 式: TCR |
|---|---|
| 分子量 | 理論値: 216.236 Da |
| Chemical component information | 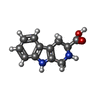 ChemComp-TCR: |
-分子 #5: PHOSPHATE ION
| 分子 | 名称: PHOSPHATE ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: PO4 |
|---|---|
| 分子量 | 理論値: 94.971 Da |
| Chemical component information |  ChemComp-PO4: |
-分子 #6: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 6 / コピー数: 8 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #7: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 7 / コピー数: 8 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.6 mg/mL | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Solution were made fresh from concentrated, and filtered to avoid microbial contamination. | ||||||||||||||||||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | ||||||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 4 seconds before plunging. | ||||||||||||||||||||||||
| 詳細 | This sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最高: 100.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum ER / エネルギーフィルター - スリット幅: 20 eV |
| 詳細 | Preliminary grid screening was performed manually. |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 13246 / 平均露光時間: 2.5 sec. / 平均電子線量: 70.35 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















