+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24303 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ cryoET map of influenza A hemagglutinin | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Influenza A virus (A/Puerto Rico/8/1934(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Puerto Rico/8/1934(H1N1)) (A型インフルエンザウイルス) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 12.9 Å | |||||||||
 データ登録者 データ登録者 | Huang QY / Song K / Schiffer CA / Somasundaran M | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2022 ジャーナル: Structure / 年: 2022タイトル: Quantitative structural analysis of influenza virus by cryo-electron tomography and convolutional neural networks. 著者: Qiuyu J Huang / Kangkang Song / Chen Xu / Daniel N A Bolon / Jennifer P Wang / Robert W Finberg / Celia A Schiffer / Mohan Somasundaran /  要旨: Influenza viruses pose severe public health threats globally. Influenza viruses are extensively pleomorphic, in shape, size, and organization of viral proteins. Analysis of influenza morphology and ...Influenza viruses pose severe public health threats globally. Influenza viruses are extensively pleomorphic, in shape, size, and organization of viral proteins. Analysis of influenza morphology and ultrastructure can help elucidate viral structure-function relationships and aid in therapeutics and vaccine development. While cryo-electron tomography (cryoET) can depict the 3D organization of pleomorphic influenza, the low signal-to-noise ratio inherent to cryoET and viral heterogeneity have precluded detailed characterization of influenza viruses. In this report, we leveraged convolutional neural networks and cryoET to characterize the morphological architecture of the A/Puerto Rico/8/34 (H1N1) influenza strain. Our pipeline improved the throughput of cryoET analysis and accurately identified viral components within tomograms. Using this approach, we successfully characterized influenza morphology, glycoprotein density, and conducted subtomogram averaging of influenza glycoproteins. Application of this processing pipeline can aid in the structural characterization of not only influenza viruses, but other pleomorphic viruses and infected cells. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24303.map.gz emd_24303.map.gz | 24.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24303-v30.xml emd-24303-v30.xml emd-24303.xml emd-24303.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24303_fsc.xml emd_24303_fsc.xml | 9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24303.png emd_24303.png | 25.2 KB | ||
| その他 |  emd_24303_half_map_1.map.gz emd_24303_half_map_1.map.gz emd_24303_half_map_2.map.gz emd_24303_half_map_2.map.gz | 11.4 MB 11.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24303 http://ftp.pdbj.org/pub/emdb/structures/EMD-24303 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24303 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24303 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24303_validation.pdf.gz emd_24303_validation.pdf.gz | 603.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24303_full_validation.pdf.gz emd_24303_full_validation.pdf.gz | 602.8 KB | 表示 | |
| XML形式データ |  emd_24303_validation.xml.gz emd_24303_validation.xml.gz | 13.1 KB | 表示 | |
| CIF形式データ |  emd_24303_validation.cif.gz emd_24303_validation.cif.gz | 16.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24303 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24303 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24303 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24303 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24303.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24303.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.087 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
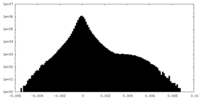
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #2
| ファイル | emd_24303_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
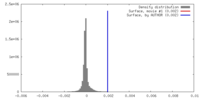
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_24303_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
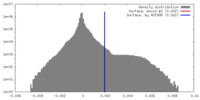
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Influenza A virus (A/Puerto Rico/8/1934(H1N1))
| 全体 | 名称:  Influenza A virus (A/Puerto Rico/8/1934(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Puerto Rico/8/1934(H1N1)) (A型インフルエンザウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Influenza A virus (A/Puerto Rico/8/1934(H1N1))
| 超分子 | 名称: Influenza A virus (A/Puerto Rico/8/1934(H1N1)) / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Purchased from Charles River Laboratories. / NCBI-ID: 211044 / 生物種: Influenza A virus (A/Puerto Rico/8/1934(H1N1)) / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 198 KDa |
-分子 #1: Influenza A H1 hemagglutinin
| 分子 | 名称: Influenza A H1 hemagglutinin / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Influenza A virus (A/Puerto Rico/8/1934(H1N1)) (A型インフルエンザウイルス) Influenza A virus (A/Puerto Rico/8/1934(H1N1)) (A型インフルエンザウイルス) |
| 配列 | 文字列: MKANLLVLLC ALAAADADTI CIGYHANNST DTVDTVLEKN VTVTHSVNLL EDSHNGKLCR LKGIAPLQLG KCNIAGWLLG NPECDPLLPV RSWSYIVETP NSENGICYPG DFIDYEELRE QLSSVSSFER FEIFPKESSW PNHNTTKGVT AACSHAGKSS FYRNLLWLTE ...文字列: MKANLLVLLC ALAAADADTI CIGYHANNST DTVDTVLEKN VTVTHSVNLL EDSHNGKLCR LKGIAPLQLG KCNIAGWLLG NPECDPLLPV RSWSYIVETP NSENGICYPG DFIDYEELRE QLSSVSSFER FEIFPKESSW PNHNTTKGVT AACSHAGKSS FYRNLLWLTE KEGSYPKLKN SYVNKKGKEV LVLWGIHHPS NSKDQQNIYQ NENAYVSVVT SNYNRRFTPE IAERPKVRDQ AGRMNYYWTL LKPGDTIIFE ANGNLIAPRY AFALSRGFGS GIITSNASMH ECNTKCQTPL GAINSSLPFQ NIHPVTIGEC PKYVRSAKLR MVTGLRNIPS IQSRGLFGAI AGFIEGGWTG MIDGWYGYHH QNEQGSGYAA DQKSTQNAIN GITNKVNSVI EKMNIQFTAV GKEFNKLEKR MENLNKKVDD GFLDIWTYNA ELLVLLENER TLDFHDSNVK NLYEKVKSQL KNNAKEIGNG CFEFYHKCDN ECMESVRNGT YDYPKYSEES KLNREKVDGV KLESMGIYQI LAIYSTVASS LVLLVSLGAI SFWMCSNGSL QCRICI |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: EMS-002 RAPID IMMERSION FREEZER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 2.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | プロトコル: FLEXIBLE FIT |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)