+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24195 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Complex structure of HIV superinfection Fab QA013.2 and BG505.SOSIP.664 | |||||||||
 マップデータ マップデータ | Complex structure of QA013.2 Fab and BG505.SOSIP.664 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / apoptotic process / host cell plasma membrane / structural molecule activity / virion membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
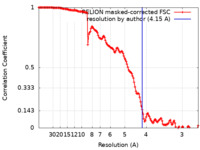
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.15 Å | |||||||||
 データ登録者 データ登録者 | Mangala Prasad V / Shipley MM / Overbaugh JM / Lee KK | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Functional development of a V3/glycan-specific broadly neutralizing antibody isolated from a case of HIV superinfection. 著者: Mackenzie M Shipley / Vidya Mangala Prasad / Laura E Doepker / Adam Dingens / Duncan K Ralph / Elias Harkins / Amrit Dhar / Dana Arenz / Vrasha Chohan / Haidyn Weight / Kishor Mandaliya / ...著者: Mackenzie M Shipley / Vidya Mangala Prasad / Laura E Doepker / Adam Dingens / Duncan K Ralph / Elias Harkins / Amrit Dhar / Dana Arenz / Vrasha Chohan / Haidyn Weight / Kishor Mandaliya / Jesse D Bloom / Frederick A Matsen / Kelly K Lee / Julie M Overbaugh /   要旨: Stimulating broadly neutralizing antibodies (bnAbs) directly from germline remains a barrier for HIV vaccines. HIV superinfection elicits bnAbs more frequently than single infection, providing clues ...Stimulating broadly neutralizing antibodies (bnAbs) directly from germline remains a barrier for HIV vaccines. HIV superinfection elicits bnAbs more frequently than single infection, providing clues of how to elicit such responses. We used longitudinal antibody sequencing and structural studies to characterize bnAb development from a superinfection case. BnAb QA013.2 bound initial and superinfecting viral Env, despite its probable naive progenitor only recognizing the superinfecting strain, suggesting both viruses influenced this lineage. A 4.15 Å cryo-EM structure of QA013.2 bound to native-like trimer showed recognition of V3 signatures (N301/N332 and GDIR). QA013.2 relies less on CDRH3 and more on framework and CDRH1 for affinity and breadth compared to other V3/glycan-specific bnAbs. Antigenic profiling revealed that viral escape was achieved by changes in the structurally-defined epitope and by mutations in V1. These results highlight shared and novel properties of QA013.2 relative to other V3/glycan-specific bnAbs in the setting of sequential, diverse antigens. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24195.map.gz emd_24195.map.gz | 228.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24195-v30.xml emd-24195-v30.xml emd-24195.xml emd-24195.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24195_fsc.xml emd_24195_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24195.png emd_24195.png | 73.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24195 http://ftp.pdbj.org/pub/emdb/structures/EMD-24195 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24195 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24195 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24195_validation.pdf.gz emd_24195_validation.pdf.gz | 526.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24195_full_validation.pdf.gz emd_24195_full_validation.pdf.gz | 526 KB | 表示 | |
| XML形式データ |  emd_24195_validation.xml.gz emd_24195_validation.xml.gz | 13.8 KB | 表示 | |
| CIF形式データ |  emd_24195_validation.cif.gz emd_24195_validation.cif.gz | 18.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24195 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24195 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24195 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24195 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24195.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24195.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Complex structure of QA013.2 Fab and BG505.SOSIP.664 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex of HIV-1 envelope protein with super-infection Fab QA013.2
| 全体 | 名称: Complex of HIV-1 envelope protein with super-infection Fab QA013.2 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of HIV-1 envelope protein with super-infection Fab QA013.2
| 超分子 | 名称: Complex of HIV-1 envelope protein with super-infection Fab QA013.2 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)株: BG505 |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: 293F Homo sapiens (ヒト) / 組換細胞: 293F |
| 分子量 | 理論値: 500 KDa |
-分子 #1: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)株: BG505 |
| 分子量 | 理論値: 56.846613 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARAENLW VTVYYGVPVW KDAETTLFCA SDAKAYETEK HNVWATHACV PTDPNPQEI HLENVTEEFN MWKNNMVEQM HTDIISLWDQ SLKPCVKLTP LCVTLQCTNV TNNITDDMRG ELKNCSFNMT T ELRDKKQK ...文字列: MDAMKRGLCC VLLLCGAVFV SPSQEIHARF RRGARAENLW VTVYYGVPVW KDAETTLFCA SDAKAYETEK HNVWATHACV PTDPNPQEI HLENVTEEFN MWKNNMVEQM HTDIISLWDQ SLKPCVKLTP LCVTLQCTNV TNNITDDMRG ELKNCSFNMT T ELRDKKQK VYSLFYRLDV VQINENQGNR SNNSNKEYRL INCNTSAITQ ACPKVSFEPI PIHYCAPAGF AILKCKDKKF NG TGPCPSV STVQCTHGIK PVVSTQLLLN GSLAEEEVMI RSENITNNAK NILVQFNTPV QINCTRPNNN TRKSIRIGPG QAF YATGDI IGDIRQAHCN VSKATWNETL GKVVKQLRKH FGNNTIIRFA NSSGGDLEVT THSFNCGGEF FYCNTSGLFN STWI SNTSV QGSNSTGSND SITLPCRIKQ IINMWQRIGQ AMYAPPIQGV IRCVSNITGL ILTRDGGSTN STTETFRPGG GDMRD NWRS ELYKYKVVKI EPLGVAPTRC KRRV |
-分子 #2: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)株: BG505 |
| 分子量 | 理論値: 18.24583 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VGRRRRRRAV GIGAVFLGFL GAAGSTMGAA SMTLTVQARN LLSGIVQQQS NLLRAPEAQQ HLLKLTVWGI KQLQARVLAV ERYLRDQQL LGIWGCSGKL ICCTNVPWNS SWSNRNLSEI WDNMTWLQWD KEISNYTQII YGLLEESQNQ QEKNEQDLLA L D |
-分子 #3: Fab QA013.2 Heavy Chain, variable region
| 分子 | 名称: Fab QA013.2 Heavy Chain, variable region / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 14.384085 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIRIAESGGG LVQPGESLRL ACEIIELGFR RAWTTWVRQA PGKGLEWVAD INEDGSEKKY GPSVTGRFTI SRDNGKNLVF LQMNSLRVE DTATYYCARE AYHLVYDDRI PRGNWFDPWG PGTLVTVSS |
-分子 #4: Fab QA013.2 Light Chain, , variable region
| 分子 | 名称: Fab QA013.2 Light Chain, , variable region / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.189263 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSVLTQPPSV SGAPGQRVVI SCTGSRSNIG AGYDVHWYQQ SPGKVPRIII YGSNSRSSGV PARFSGSKSG TSASLAITGL QAEDEADYY CQSYDTTLTA SVFGGGTKV |
-分子 #11: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 11 / コピー数: 9 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #12: alpha-D-mannopyranose
| 分子 | 名称: alpha-D-mannopyranose / タイプ: ligand / ID: 12 / コピー数: 3 / 式: MAN |
|---|---|
| 分子量 | 理論値: 180.156 Da |
| Chemical component information |  ChemComp-MAN: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 名称: Phosphate buffer saline / 詳細: 1X PBS at pH 7.4, made fresh. |
| グリッド | モデル: PELCO Ultrathin Carbon with Lacey Carbon / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 3ul sample volume, blotted for 3-4 seconds. |
| 詳細 | Monodisperse sample in PBS |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | Preliminary grid screening was performed manually |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 6475 / 平均露光時間: 10.0 sec. / 平均電子線量: 44.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.7 µm / 倍率(公称値): 110000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)