+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23942 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bartonella henselae NrnC bound to pGG. C1 reconstruction. | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNase / bacteria / enzyme / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報nucleobase-containing compound metabolic process / 3'-5' exonuclease activity / nucleic acid binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Bartonella henselae (バクテリア) / synthetic construct (人工物) Bartonella henselae (バクテリア) / synthetic construct (人工物) | |||||||||
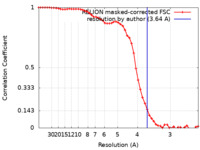
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.64 Å | |||||||||
 データ登録者 データ登録者 | Lormand JD / Brownfield B | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: Structural characterization of NrnC identifies unifying features of dinucleotidases. 著者: Justin D Lormand / Soo-Kyoung Kim / George A Walters-Marrah / Bryce A Brownfield / J Christopher Fromme / Wade C Winkler / Jonathan R Goodson / Vincent T Lee / Holger Sondermann /   要旨: RNA degradation is fundamental for cellular homeostasis. The process is carried out by various classes of endolytic and exolytic enzymes that together degrade an RNA polymer to mono-ribonucleotides. ...RNA degradation is fundamental for cellular homeostasis. The process is carried out by various classes of endolytic and exolytic enzymes that together degrade an RNA polymer to mono-ribonucleotides. Within the exoribonucleases, nano-RNases play a unique role as they act on the smallest breakdown products and hence catalyze the final steps in the process. We recently showed that oligoribonuclease (Orn) acts as a dedicated diribonucleotidase, defining the ultimate step in RNA degradation that is crucial for cellular fitness (Kim et al., 2019). Whether such a specific activity exists in organisms that lack Orn-type exoribonucleases remained unclear. Through quantitative structure-function analyses, we show here that NrnC-type RNases share this narrow substrate length preference with Orn. Although NrnC and Orn employ similar structural features that distinguish these two classes of dinucleotidases from other exonucleases, the key determinants for dinucleotidase activity are realized through distinct structural scaffolds. The structures, together with comparative genomic analyses of the phylogeny of DEDD-type exoribonucleases, indicate convergent evolution as the mechanism of how dinucleotidase activity emerged repeatedly in various organisms. The evolutionary pressure to maintain dinucleotidase activity further underlines the important role these analogous proteins play for cell growth. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23942.map.gz emd_23942.map.gz | 2.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23942-v30.xml emd-23942-v30.xml emd-23942.xml emd-23942.xml | 21.8 KB 21.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23942_fsc.xml emd_23942_fsc.xml | 5.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23942.png emd_23942.png | 109.4 KB | ||
| Filedesc metadata |  emd-23942.cif.gz emd-23942.cif.gz | 6.4 KB | ||
| その他 |  emd_23942_additional_1.map.gz emd_23942_additional_1.map.gz emd_23942_half_map_1.map.gz emd_23942_half_map_1.map.gz emd_23942_half_map_2.map.gz emd_23942_half_map_2.map.gz | 8.6 MB 8.6 MB 8.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23942 http://ftp.pdbj.org/pub/emdb/structures/EMD-23942 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23942 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23942 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7mqcMC  7mplC  7mpmC  7mpnC  7mpoC  7mppC  7mpqC  7mprC  7mpsC  7mptC  7mpuC  7mqbC  7mqdC  7mqeC  7mqfC  7mqgC  7mqhC  7mqiC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23942.map.gz / 形式: CCP4 / 大きさ: 11.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23942.map.gz / 形式: CCP4 / 大きさ: 11.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: #1
| ファイル | emd_23942_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_23942_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_23942_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NrnC octamer bound to pGG
| 全体 | 名称: NrnC octamer bound to pGG |
|---|---|
| 要素 |
|
-超分子 #1: NrnC octamer bound to pGG
| 超分子 | 名称: NrnC octamer bound to pGG / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 186.621 kDa/nm |
-超分子 #2: NrnC octamer
| 超分子 | 名称: NrnC octamer / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Bartonella henselae (バクテリア) Bartonella henselae (バクテリア) |
-超分子 #3: pGG
| 超分子 | 名称: pGG / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Bartonella henselae (バクテリア) / Synthetically produced: Yes Bartonella henselae (バクテリア) / Synthetically produced: Yes |
-分子 #1: NanoRNase C
| 分子 | 名称: NanoRNase C / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Bartonella henselae (バクテリア) Bartonella henselae (バクテリア) |
| 分子量 | 理論値: 23.446725 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SMTEIRVHQG DLPNLDNYRI DAVAVDTETL GLQPHRDRLC VVQLSSGDGT ADVIQIAKGQ KSAPNLVRLL SDRDITKIFH FGRFDLAIL AHTFGVMPDV VFCTKIASKL TRTYTDRHGL KEICGELLNV NISKQQQSSD WAAETLSRAQ IEYAASDVLY L HRLKDIFE ...文字列: SMTEIRVHQG DLPNLDNYRI DAVAVDTETL GLQPHRDRLC VVQLSSGDGT ADVIQIAKGQ KSAPNLVRLL SDRDITKIFH FGRFDLAIL AHTFGVMPDV VFCTKIASKL TRTYTDRHGL KEICGELLNV NISKQQQSSD WAAETLSRAQ IEYAASDVLY L HRLKDIFE ERLKREERES VAKACFQFLP MRANLDLLGW SEIDIFAHS UniProtKB: 3'-5' exonuclease |
-分子 #2: RNA (5'-R(P*GP*G)-3')
| 分子 | 名称: RNA (5'-R(P*GP*G)-3') / タイプ: rna / ID: 2 / コピー数: 8 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 645.454 Da |
| 配列 | 文字列: GG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)