+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23589 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of 2909 Fab in complex with 3BNC117 Fab and CAP256.wk34.c80 SOSIP.RnS2 N160K HIV-1 Env trimer | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / apoptotic process / host cell plasma membrane / structural molecule activity / virion membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.91 Å | |||||||||
 データ登録者 データ登録者 | Gorman J / Kwong PD | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Extended antibody-framework-to-antigen distance observed exclusively with broad HIV-1-neutralizing antibodies recognizing glycan-dense surfaces. 著者: Myungjin Lee / Anita Changela / Jason Gorman / Reda Rawi / Tatsiana Bylund / Cara W Chao / Bob C Lin / Mark K Louder / Adam S Olia / Baoshan Zhang / Nicole A Doria-Rose / Susan Zolla-Pazner / ...著者: Myungjin Lee / Anita Changela / Jason Gorman / Reda Rawi / Tatsiana Bylund / Cara W Chao / Bob C Lin / Mark K Louder / Adam S Olia / Baoshan Zhang / Nicole A Doria-Rose / Susan Zolla-Pazner / Lawrence Shapiro / Gwo-Yu Chuang / Peter D Kwong /  要旨: Antibody-Framework-to-Antigen Distance (AFAD) - the distance between the body of an antibody and a protein antigen - is an important parameter governing antibody recognition. Here, we quantify AFAD ...Antibody-Framework-to-Antigen Distance (AFAD) - the distance between the body of an antibody and a protein antigen - is an important parameter governing antibody recognition. Here, we quantify AFAD for ~2,000 non-redundant antibody-protein-antigen complexes in the Protein Data Bank. AFADs showed a gaussian distribution with mean of 16.3 Å and standard deviation (σ) of 2.4 Å. Notably, antibody-antigen complexes with extended AFADs (>3σ) were exclusively human immunodeficiency virus-type 1 (HIV-1)-neutralizing antibodies. High correlation (R = 0.8110) was observed between AFADs and glycan coverage, as assessed by molecular dynamics simulations of the HIV-1-envelope trimer. Especially long AFADs were observed for antibodies targeting the glycosylated trimer apex, and we tested the impact of introducing an apex-glycan hole (N160K); the cryo-EM structure of the glycan hole-targeting HIV-1-neutralizing antibody 2909 in complex with an N160K-envelope trimer revealed a substantially shorter AFAD. Overall, extended AFADs exclusively recognized densely glycosylated surfaces, with the introduction of a glycan hole enabling closer recognition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23589.map.gz emd_23589.map.gz | 228.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23589-v30.xml emd-23589-v30.xml emd-23589.xml emd-23589.xml | 26.1 KB 26.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23589.png emd_23589.png | 108.2 KB | ||
| マスクデータ |  emd_23589_msk_1.map emd_23589_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_23589_additional_1.map.gz emd_23589_additional_1.map.gz emd_23589_additional_2.map.gz emd_23589_additional_2.map.gz emd_23589_half_map_1.map.gz emd_23589_half_map_1.map.gz emd_23589_half_map_2.map.gz emd_23589_half_map_2.map.gz | 14.6 MB 63.9 MB 226.4 MB 226.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23589 http://ftp.pdbj.org/pub/emdb/structures/EMD-23589 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23589 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23589 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23589_validation.pdf.gz emd_23589_validation.pdf.gz | 909.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23589_full_validation.pdf.gz emd_23589_full_validation.pdf.gz | 909 KB | 表示 | |
| XML形式データ |  emd_23589_validation.xml.gz emd_23589_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_23589_validation.cif.gz emd_23589_validation.cif.gz | 19.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23589 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23589 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23589 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23589 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23589.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23589.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
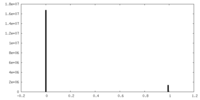
| ボクセルのサイズ | X=Y=Z: 1.0961 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
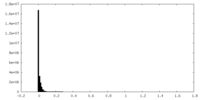
| ファイル |  emd_23589_msk_1.map emd_23589_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
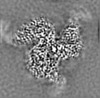
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
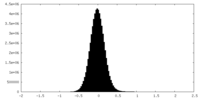
-追加マップ: Density modified map from phenix resolve
| ファイル | emd_23589_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density modified map from phenix resolve | ||||||||||||
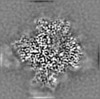
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
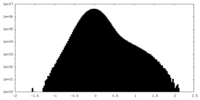
-追加マップ: Unsharpened Map
| ファイル | emd_23589_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened Map | ||||||||||||
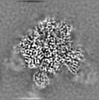
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
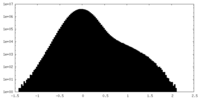
-ハーフマップ: Half map B
| ファイル | emd_23589_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_23589_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 2909 Fab in complex with 3BNC117 Fab and CAP256.wk34.c80 SOSIP.Rn...
| 全体 | 名称: 2909 Fab in complex with 3BNC117 Fab and CAP256.wk34.c80 SOSIP.RnS2 N160K HIV-1 Env trimer |
|---|---|
| 要素 |
|
-超分子 #1: 2909 Fab in complex with 3BNC117 Fab and CAP256.wk34.c80 SOSIP.Rn...
| 超分子 | 名称: 2909 Fab in complex with 3BNC117 Fab and CAP256.wk34.c80 SOSIP.RnS2 N160K HIV-1 Env trimer タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-分子 #1: 2909 Light Chain
| 分子 | 名称: 2909 Light Chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.582885 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: SYVLTQPPSV SVAPGKTARI TCGGNNIANK NVHWYQQKPG QAPVLVIYYD DDRPSGIPDR FSGSNSGNTA TLTISRVEAG DEADYYCQV WDSNSDHVVF GGGTQLTVLG QPKAAPSVTL FPPSSEELQA NKATLVCLIS DFYPGAVTVA WKADSSPVKA G VETTTPSK ...文字列: SYVLTQPPSV SVAPGKTARI TCGGNNIANK NVHWYQQKPG QAPVLVIYYD DDRPSGIPDR FSGSNSGNTA TLTISRVEAG DEADYYCQV WDSNSDHVVF GGGTQLTVLG QPKAAPSVTL FPPSSEELQA NKATLVCLIS DFYPGAVTVA WKADSSPVKA G VETTTPSK QSNNKYAASS YLSLTPEQWK SHRSYSCQVT HEGSTVEKTV APT |
-分子 #2: 2909 Heavy Chain
| 分子 | 名称: 2909 Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.067795 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVQLVESGGN VVQPGGSLRL SCTASGFSFD DSTMHWVRQA PGKGLQWVSL ISWNGGRTYY ADSVKGRFTI SRDNSKNSLY LQMNSLKTE DTAFYFCAKD KGDSD(TYS)D(TYS)NL GYSYFYYMDG WGKGTTVTVS SASTKGPSVF PLAPSSKSTS GGT AALGCL ...文字列: EVQLVESGGN VVQPGGSLRL SCTASGFSFD DSTMHWVRQA PGKGLQWVSL ISWNGGRTYY ADSVKGRFTI SRDNSKNSLY LQMNSLKTE DTAFYFCAKD KGDSD(TYS)D(TYS)NL GYSYFYYMDG WGKGTTVTVS SASTKGPSVF PLAPSSKSTS GGT AALGCL VKDYFPEPVT VSWNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKRV EPK |
-分子 #3: Envelope glycoprotein gp120
| 分子 | 名称: Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 53.337512 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GLWVTVYYGV PVWREAKTTL FCASDAKSYE KEVHNVWATH ACVPTDPNPQ ELVLENVTEN FNMWKNDMVD QMHEDIISLW DQSLKPCVK LTPLCVTLNC SDAKVNATYK GTREEIKNCS FKATTELRDK KRREYALFYR LDIVPLSGEG NNNSEYRLIN C NTSVITQI ...文字列: GLWVTVYYGV PVWREAKTTL FCASDAKSYE KEVHNVWATH ACVPTDPNPQ ELVLENVTEN FNMWKNDMVD QMHEDIISLW DQSLKPCVK LTPLCVTLNC SDAKVNATYK GTREEIKNCS FKATTELRDK KRREYALFYR LDIVPLSGEG NNNSEYRLIN C NTSVITQI CPKVTFDPIP IHYCAPAGYA ILKCNNKTFN GTGPCNNVST VQCTHGIKPV VSTQLLLNGS LAEEEIIIRS EN LTDNVKT IIVHLNESVE ITCTRPNNMT RKSVRIGPGQ TFYALGDIIG DIRQPHCNIS EIKWEKTLQR VSEKLREHFN KTI IFNQSS GGDLEITTHS FNCGGEFFYC NTSDLFFNKT FNETYSTGSN STNSTITLPC RIKQIINMWQ EVGRAMYAPP IAGN ITCKS NITGLLLTRD GGGNNSTKET FRPGGGNMRD NWRSELYKYK VVEVKPLGIA PTECNRTVVQ RRRRRR |
-分子 #4: 3BNC117 Heavy Chain
| 分子 | 名称: 3BNC117 Heavy Chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.656484 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLLQSGAA VTKPGASVRV SCEASGYNIR DYFIHWWRQA PGQGLQWVGW INPKTGQPNN PRQFQGRVSL TRHASWDFDT YSFYMDLKA LRSDDTAVYF CARQRSDYWD FDVWGSGTQV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA ...文字列: QVQLLQSGAA VTKPGASVRV SCEASGYNIR DYFIHWWRQA PGQGLQWVGW INPKTGQPNN PRQFQGRVSL TRHASWDFDT YSFYMDLKA LRSDDTAVYF CARQRSDYWD FDVWGSGTQV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKVEPKSC |
-分子 #5: 3BNC117 Light Chain
| 分子 | 名称: 3BNC117 Light Chain / タイプ: protein_or_peptide / ID: 5 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.022658 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPSS LSASVGDTVT ITCQANGYLN WYQQRRGKAP KLLIYDGSKL ERGVPSRFSG RRWGQEYNLT INNLQPEDIA TYFCQVYEF VVPGTRLDLK RTVAAPSVFI FPPSDEQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG NSQESVTEQD S KDSTYSLS ...文字列: DIQMTQSPSS LSASVGDTVT ITCQANGYLN WYQQRRGKAP KLLIYDGSKL ERGVPSRFSG RRWGQEYNLT INNLQPEDIA TYFCQVYEF VVPGTRLDLK RTVAAPSVFI FPPSDEQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG NSQESVTEQD S KDSTYSLS STLTLSKADY EKHKVYACEV THQGLSSPVT KSFNRGEC |
-分子 #6: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 6 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.355703 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVVGLGAVFL GFLGAAGSTM GAASNTLTVQ ARQLLSGIVQ QQSNLLRAPE AQQHMLQLGV WGFKQLQARV LAIERYLEVQ QLLGMWGCS GKLICCTNVP WNSSWSNKTY NEIWDNMTWM QWDREIGNYT DTIYKLLEVS QFQQEINEKD NLTLD |
-分子 #11: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 11 / コピー数: 54 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 式: PBS |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / 支持フィルム - 材質: CARBON / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | PBS |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 66.88 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.91 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 2.15) / 使用した粒子像数: 35406 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-7ly9: |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X