+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23542 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of full-length GluK1 with L-Glu | |||||||||
 マップデータ マップデータ | GluK1 with L-Glu (full-length | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / negative regulation of synaptic transmission, GABAergic / L-glutamate transmembrane transporter activity / positive regulation of gamma-aminobutyric acid secretion / Activation of Na-permeable kainate receptors / kainate selective glutamate receptor complex / Activation of Ca-permeable Kainate Receptor / negative regulation of synaptic transmission, glutamatergic / regulation of short-term neuronal synaptic plasticity /  inhibitory postsynaptic potential ...: / negative regulation of synaptic transmission, GABAergic / L-glutamate transmembrane transporter activity / positive regulation of gamma-aminobutyric acid secretion / Activation of Na-permeable kainate receptors / kainate selective glutamate receptor complex / Activation of Ca-permeable Kainate Receptor / negative regulation of synaptic transmission, glutamatergic / regulation of short-term neuronal synaptic plasticity / inhibitory postsynaptic potential ...: / negative regulation of synaptic transmission, GABAergic / L-glutamate transmembrane transporter activity / positive regulation of gamma-aminobutyric acid secretion / Activation of Na-permeable kainate receptors / kainate selective glutamate receptor complex / Activation of Ca-permeable Kainate Receptor / negative regulation of synaptic transmission, glutamatergic / regulation of short-term neuronal synaptic plasticity /  inhibitory postsynaptic potential / inhibitory postsynaptic potential /  glutamate receptor activity / glutamate receptor activity /  glutamate binding / glutamate binding /  adult behavior / behavioral response to pain / adult behavior / behavioral response to pain /  脱分極 / ligand-gated monoatomic ion channel activity / kainate selective glutamate receptor activity / 脱分極 / ligand-gated monoatomic ion channel activity / kainate selective glutamate receptor activity /  ionotropic glutamate receptor complex / extracellularly glutamate-gated ion channel activity / glutamate-gated receptor activity / ionotropic glutamate receptor signaling pathway / ionotropic glutamate receptor complex / extracellularly glutamate-gated ion channel activity / glutamate-gated receptor activity / ionotropic glutamate receptor signaling pathway /  excitatory postsynaptic potential / excitatory postsynaptic potential /  synaptic transmission, glutamatergic / synaptic transmission, glutamatergic /  regulation of membrane potential / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / positive regulation of synaptic transmission, GABAergic / regulation of membrane potential / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / positive regulation of synaptic transmission, GABAergic /  regulation of synaptic plasticity / modulation of chemical synaptic transmission / regulation of synaptic plasticity / modulation of chemical synaptic transmission /  terminal bouton / terminal bouton /  presynaptic membrane / presynaptic membrane /  signaling receptor activity / signaling receptor activity /  nervous system development / chemical synaptic transmission / nervous system development / chemical synaptic transmission /  postsynaptic membrane / postsynaptic membrane /  postsynaptic density / postsynaptic density /  receptor complex / neuronal cell body / glutamatergic synapse / receptor complex / neuronal cell body / glutamatergic synapse /  シナプス / シナプス /  樹状突起 / 樹状突起 /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.6 Å クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||
 データ登録者 データ登録者 | Meyerson JR / Selvakumar P | |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2021 ジャーナル: Cell Rep / 年: 2021タイトル: Structural and compositional diversity in the kainate receptor family. 著者: Purushotham Selvakumar / Joon Lee / Nandish Khanra / Changhao He / Hermany Munguba / Lisa Kiese / Johannes Broichhagen / Andreas Reiner / Joshua Levitz / Joel R Meyerson /   要旨: The kainate receptors (KARs) are members of the ionotropic glutamate receptor family and assemble into tetramers from a pool of five subunit types (GluK1-5). Each subunit confers distinct functional ...The kainate receptors (KARs) are members of the ionotropic glutamate receptor family and assemble into tetramers from a pool of five subunit types (GluK1-5). Each subunit confers distinct functional properties to a receptor, but the compositional and stoichiometric diversity of KAR tetramers is not well understood. To address this, we first solve the structure of the GluK1 homomer, which enables a systematic assessment of structural compatibility among KAR subunits. Next, we analyze single-cell RNA sequencing data, which reveal extreme diversity in the combinations of two or more KAR subunits co-expressed within the same cell. We then investigate the composition of individual receptor complexes using single-molecule fluorescence techniques and find that di-heteromers assembled from GluK1, GluK2, or GluK3 can form with all possible stoichiometries, while GluK1/K5, GluK2/K5, and GluK3/K5 can form 3:1 or 2:2 complexes. Finally, using three-color single-molecule imaging, we discover that KARs can form tri- and tetra-heteromers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23542.map.gz emd_23542.map.gz | 117.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23542-v30.xml emd-23542-v30.xml emd-23542.xml emd-23542.xml | 13.8 KB 13.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23542_fsc.xml emd_23542_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23542.png emd_23542.png | 54 KB | ||
| その他 |  emd_23542_additional_1.map.gz emd_23542_additional_1.map.gz emd_23542_additional_2.map.gz emd_23542_additional_2.map.gz | 2.3 MB 98.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23542 http://ftp.pdbj.org/pub/emdb/structures/EMD-23542 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23542 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23542 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23542.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23542.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GluK1 with L-Glu (full-length | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.096 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: GluK1 with L-Glu (ATD)
| ファイル | emd_23542_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GluK1 with L-Glu (ATD) | ||||||||||||
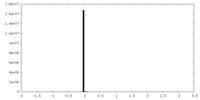
| 投影像・断面図 |
| ||||||||||||
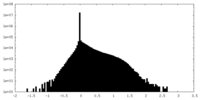
| 密度ヒストグラム |
-追加マップ: GluK1 with L-Glu (LBD-TMD)
| ファイル | emd_23542_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GluK1 with L-Glu (LBD-TMD) | ||||||||||||
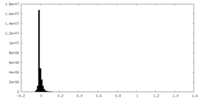
| 投影像・断面図 |
| ||||||||||||
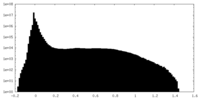
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GluK1 tetrameric complex
| 全体 | 名称: GluK1 tetrameric complex |
|---|---|
| 要素 |
|
-超分子 #1: GluK1 tetrameric complex
| 超分子 | 名称: GluK1 tetrameric complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Isoform Glur5-2 of Glutamate receptor ionotropic, kainate 1
| 分子 | 名称: Isoform Glur5-2 of Glutamate receptor ionotropic, kainate 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
| 分子量 | 理論値: 102.562078 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKIISPVLSN LVFSRSIKVL LCLLWIGYSQ GTTHVLRIGG IFETVENEPV NVEELAFKFA VTSINRNRTL MPNTTLTYDI QRINLFDSF EASRRACDQL ALGVAALFGP SHSSSVSAVQ SICNALEVPH IQTRWKHPSV DSRDLFYINL YPDYAAISRA V LDLVLYYN ...文字列: MKIISPVLSN LVFSRSIKVL LCLLWIGYSQ GTTHVLRIGG IFETVENEPV NVEELAFKFA VTSINRNRTL MPNTTLTYDI QRINLFDSF EASRRACDQL ALGVAALFGP SHSSSVSAVQ SICNALEVPH IQTRWKHPSV DSRDLFYINL YPDYAAISRA V LDLVLYYN WKTVTVVYED STGLIRLQEL IKAPSRYNIK IKIRQLPPAN KDAKPLLKEM KKSKEFYVIF DCSHETAAEI LK QILFMGM MTEYYHYFFT TLDLFALDLE LYRYSGVNMT GFRLLNIDNP HVSSIIEKWS MERLQAPPRP ETGLLDGMMT TEA ALMYDA VYMVAIASHR ASQLTVSSLQ CHRHKPWRLG PRFMNLIKEA RWDGLTGRIT FNKTDGLRKD FDLDIISLKE EGTE KIGIW NSNSGLNMTD GNRDRSNNIT DSLANRTLIV TTILEEPYVM YRKSDKPLYG NDRFEGYCLD LLKELSNILG FLYDV KLVP DGKYGAQNDK GEWNGMVKEL IDHRADLAVA PLTITYVREK VIDFSKPFMT LGISILYRKP NGTNPGVFSF LNPLSP DIW MYVLLACLGV SCVLFVIARF TPYEWYNPHP CNPDSDVVEN NFTLLNSFWF GVGALMQQGS ELMPKALSTR IVGGIWW FF TLIIISSYTA NLAAFLTVER MESPIDSADD LAKQTKIEYG AVRDGSTMTF FKKSKISTYE KMWAFMSSRQ QSALVKNS D EGIQRVLTTD YALLMESTSI EYVTQRNCNL TQIGGLIDSK GYGVGTPIGS PYRDKITIAI LQLQEEGKLH MMKEKWWRG NGCPEEDSKE ASALGVENIG GIFIVLAAGL VLSVFVAIGE FLYKSRKNND VEQCLSFNAI MEELGISLKN QKKLKKKSRT KGKSSFTSI LTCHQRRTQR KETVA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 52.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X