+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2339 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Variable internal flexibility characterizes the helical capsid formed by Agrobacterium VirE2 protein on single-stranded DNA. | |||||||||
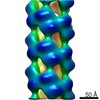
 マップデータ マップデータ | CryoEM reconstruction of the Agrobacterium T-complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | tcomplex / agrobacterium / helical reconstruction | |||||||||
| 機能・相同性 | VirE2 / VirE2 / DNA-mediated transformation / host cell nucleus / DNA binding / extracellular region / identical protein binding / Single-strand DNA-binding protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Agrobacterium fabrum str. C58 (バクテリア) Agrobacterium fabrum str. C58 (バクテリア) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Bharat TAM / Zbaida D / Eisenstein M / Frankenstein Z / Mehlman T / Weiner L / Sorzano COS / Barak Y / Albeck S / Briggs JAG ...Bharat TAM / Zbaida D / Eisenstein M / Frankenstein Z / Mehlman T / Weiner L / Sorzano COS / Barak Y / Albeck S / Briggs JAG / Wolf SG / Elbaum M | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2013 ジャーナル: Structure / 年: 2013タイトル: Variable internal flexibility characterizes the helical capsid formed by agrobacterium VirE2 protein on single-stranded DNA. 著者: Tanmay A M Bharat / David Zbaida / Miriam Eisenstein / Ziv Frankenstein / Tevie Mehlman / Lev Weiner / Carlos Oscar S Sorzano / Yoav Barak / Shira Albeck / John A G Briggs / Sharon G Wolf / Michael Elbaum /  要旨: Agrobacterium is known for gene transfer to plants. In addition to a linear ssDNA oligonucleotide, Agrobacterium tumefaciens secretes an abundant ssDNA-binding effector, VirE2. In many ways VirE2 ...Agrobacterium is known for gene transfer to plants. In addition to a linear ssDNA oligonucleotide, Agrobacterium tumefaciens secretes an abundant ssDNA-binding effector, VirE2. In many ways VirE2 adapts the conjugation mechanism to transform the eukaryotic host. The crystal structure of VirE2 shows two compact domains joined by a flexible linker. Bound to ssDNA, VirE2 forms an ordered solenoidal shell, or capsid known as the T-complex. Here, we present a three-dimensional reconstruction of the VirE2-ssDNA complex using cryo-electron microscopy and iterative helical real-space reconstruction. High-resolution refinement was not possible due to inherent heterogeneity in the protein structure. By a combination of computational modeling, chemical modifications, mass spectroscopy, and electron paramagnetic resonance, we found that the N-terminal domain is tightly constrained by both tangential and longitudinal links, while the C terminus is weakly constrained. The quaternary structure is thus rigidly assembled while remaining locally flexible. This flexibility may be important in accommodating substrates without sequence specificity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2339.map.gz emd_2339.map.gz | 247.1 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2339-v30.xml emd-2339-v30.xml emd-2339.xml emd-2339.xml | 11.3 KB 11.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2339.jpg emd_2339.jpg | 75.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2339 http://ftp.pdbj.org/pub/emdb/structures/EMD-2339 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2339 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2339 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2339_validation.pdf.gz emd_2339_validation.pdf.gz | 220 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2339_full_validation.pdf.gz emd_2339_full_validation.pdf.gz | 219.1 KB | 表示 | |
| XML形式データ |  emd_2339_validation.xml.gz emd_2339_validation.xml.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2339 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2339 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2339 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2339 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2339.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2339.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM reconstruction of the Agrobacterium T-complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.32 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
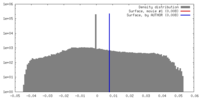
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Agrobacterium T-complex
| 全体 | 名称: Agrobacterium T-complex |
|---|---|
| 要素 |
|
-超分子 #1000: Agrobacterium T-complex
| 超分子 | 名称: Agrobacterium T-complex / タイプ: sample / ID: 1000 / 集合状態: Helical / Number unique components: 2 |
|---|
-分子 #1: VirE2
| 分子 | 名称: VirE2 / タイプ: protein_or_peptide / ID: 1 / 集合状態: Helical / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Agrobacterium fabrum str. C58 (バクテリア) Agrobacterium fabrum str. C58 (バクテリア) |
| 組換発現 | 生物種:  |
-分子 #2: short oligomeric 26mer DNA
| 分子 | 名称: short oligomeric 26mer DNA / タイプ: dna / ID: 2 / 分類: DNA / Structure: SINGLE STRANDED / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種:  Agrobacterium fabrum str. C58 (バクテリア) Agrobacterium fabrum str. C58 (バクテリア) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 50 mM Tris, 500 mM NaCl |
| グリッド | 詳細: Quantifoil holey carbon |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: HOMEMADE PLUNGER |
| 詳細 | Protein was mixed with single-stranded DNA |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at high-magnification (>100,000) |
| 日付 | 2008年6月6日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC TVIPS / 平均電子線量: 20 e/Å2 / 詳細: Image data was collected as focal pairs. |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 3.2 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Particles were picked and preselected using routines of Xmipp, and then reconstruction was carried out using IHRSR. |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 14.67 Å 想定した対称性 - らせんパラメータ - ΔΦ: 110.09 ° 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF ソフトウェア - 名称: Bsoft, EMAN, Xmipp, Spider, IHRSR |
| CTF補正 | 詳細: Phase-flipping |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: fitPDB2EM |
| 詳細 | The N and C terminal domain were fit separately by exhaustive molecular modeling using the fitPDB2EM program. Only the N-terminal domain could be constrained strongly. |
| 精密化 | 空間: RECIPROCAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: Highest cross-correlation |
| 得られたモデル |  PDB-4blf: |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)