+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23351 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Arabidopsis thaliana sulfate transporter AtSULTR4;1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Sulfate transport / SLC26 / MEMBRANE PROTEIN / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / chloroplast membrane / secondary active sulfate transmembrane transporter activity / symporter activity / plasma membrane => GO:0005886 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
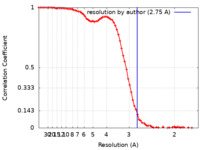
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.75 Å | |||||||||
 データ登録者 データ登録者 | Wang L / Chen K | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
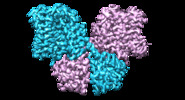
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structure and function of an Arabidopsis thaliana sulfate transporter. 著者: Lie Wang / Kehan Chen / Ming Zhou /  要旨: Plant sulfate transporters (SULTR) mediate absorption and distribution of sulfate (SO) and are essential for plant growth; however, our understanding of their structures and functions remains ...Plant sulfate transporters (SULTR) mediate absorption and distribution of sulfate (SO) and are essential for plant growth; however, our understanding of their structures and functions remains inadequate. Here we present the structure of a SULTR from Arabidopsis thaliana, AtSULTR4;1, in complex with SO at an overall resolution of 2.8 Å. AtSULTR4;1 forms a homodimer and has a structural fold typical of the SLC26 family of anion transporters. The bound SO is coordinated by side-chain hydroxyls and backbone amides, and further stabilized electrostatically by the conserved Arg393 and two helix dipoles. Proton and SO are co-transported by AtSULTR4;1 and a proton gradient significantly enhances SO transport. Glu347, which is ~7 Å from the bound SO, is required for H-driven transport. The cytosolic STAS domain interacts with transmembrane domains, and deletion of the STAS domain or mutations to the interface compromises dimer formation and reduces SO transport, suggesting a regulatory function of the STAS domain. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23351.map.gz emd_23351.map.gz | 28.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23351-v30.xml emd-23351-v30.xml emd-23351.xml emd-23351.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23351_fsc.xml emd_23351_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23351.png emd_23351.png | 295.5 KB | ||
| Filedesc metadata |  emd-23351.cif.gz emd-23351.cif.gz | 5.7 KB | ||
| その他 |  emd_23351_half_map_1.map.gz emd_23351_half_map_1.map.gz emd_23351_half_map_2.map.gz emd_23351_half_map_2.map.gz | 28.2 MB 28.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23351 http://ftp.pdbj.org/pub/emdb/structures/EMD-23351 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23351 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23351 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23351_validation.pdf.gz emd_23351_validation.pdf.gz | 794.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23351_full_validation.pdf.gz emd_23351_full_validation.pdf.gz | 794.5 KB | 表示 | |
| XML形式データ |  emd_23351_validation.xml.gz emd_23351_validation.xml.gz | 14.1 KB | 表示 | |
| CIF形式データ |  emd_23351_validation.cif.gz emd_23351_validation.cif.gz | 17.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23351 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23351 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23351 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23351 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23351.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23351.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #2
| ファイル | emd_23351_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
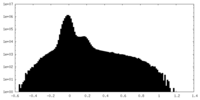
| 投影像・断面図 |
| ||||||||||||
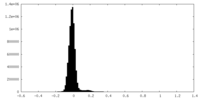
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_23351_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
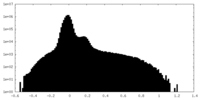
| 投影像・断面図 |
| ||||||||||||
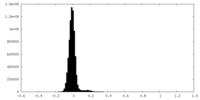
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Sulfate transporter 4;1
| 全体 | 名称: Sulfate transporter 4;1 |
|---|---|
| 要素 |
|
-超分子 #1: Sulfate transporter 4;1
| 超分子 | 名称: Sulfate transporter 4;1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 70 KDa |
-分子 #1: Sulfate transporter 4.1, chloroplastic
| 分子 | 名称: Sulfate transporter 4.1, chloroplastic / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 75.168164 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSYASLSVKD LTSLVSRSGT GSSSSLKPPG QTRPVKVIPL QHPDTSNEAR PPSIPFDDIF SGWTAKIKRM RLVDWIDTLF PCFRWIRTY RWSEYFKLDL MAGITVGIML VPQAMSYAKL AGLPPIYGLY SSFVPVFVYA IFGSSRQLAI GPVALVSLLV S NALGGIAD ...文字列: MSYASLSVKD LTSLVSRSGT GSSSSLKPPG QTRPVKVIPL QHPDTSNEAR PPSIPFDDIF SGWTAKIKRM RLVDWIDTLF PCFRWIRTY RWSEYFKLDL MAGITVGIML VPQAMSYAKL AGLPPIYGLY SSFVPVFVYA IFGSSRQLAI GPVALVSLLV S NALGGIAD TNEELHIELA ILLALLVGIL ECIMGLLRLG WLIRFISHSV ISGFTSASAI VIGLSQIKYF LGYSIARSSK IV PIVESII AGADKFQWPP FVMGSLILVI LQVMKHVGKA KKELQFLRAA APLTGIVLGT TIAKVFHPPS ISLVGEIPQG LPT FSFPRS FDHAKTLLPT SALITGVAIL ESVGIAKALA AKNRYELDSN SELFGLGVAN ILGSLFSAYP ATGSFSRSAV NNES EAKTG LSGLITGIII GCSLLFLTPM FKYIPQCALA AIVISAVSGL VDYDEAIFLW RVDKRDFSLW TITSTITLFF GIEIG VLVG VGFSLAFVIH ESANPHIAVL GRLPGTTVYR NIKQYPEAYT YNGIVIVRID SPIYFANISY IKDRLREYEV AVDKYT NRG LEVDRINFVI LEMSPVTHID SSAVEALKEL YQEYKTRDIQ LAISNPNKDV HLTIARSGMV ELVGKEWFFV RVHDAVQ VC LQYVQSSNLE DKHLSFTRRY GGSNNNSSSS NALLKEPLLS VEK UniProtKB: Sulfate transporter 4.1, chloroplastic |
-分子 #2: (2S,3R,4E)-2-amino-3-hydroxyoctadec-4-en-1-yl dihydrogen phosphate
| 分子 | 名称: (2S,3R,4E)-2-amino-3-hydroxyoctadec-4-en-1-yl dihydrogen phosphate タイプ: ligand / ID: 2 / コピー数: 4 / 式: S1P |
|---|---|
| 分子量 | 理論値: 379.472 Da |
| Chemical component information | 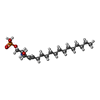 ChemComp-S1P: |
-分子 #3: SULFATE ION
| 分子 | 名称: SULFATE ION / タイプ: ligand / ID: 3 / コピー数: 2 / 式: SO4 |
|---|---|
| 分子量 | 理論値: 96.063 Da |
| Chemical component information |  ChemComp-SO4: |
-分子 #4: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine
| 分子 | 名称: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine / タイプ: ligand / ID: 4 / コピー数: 2 / 式: LBN |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-LBN: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 2 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X