+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23336 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
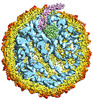
| タイトル | Phage Qbeta virion | |||||||||||||||
 マップデータ マップデータ | Qbeta Virion | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報suppression by virus of host cell wall biogenesis / suppression by virus of host peptidoglycan biosynthetic process / viral release via suppression of host peptidoglycan biosynthetic process / virion attachment to host cell pilus / T=3 icosahedral viral capsid / adhesion receptor-mediated virion attachment to host cell / translation repressor activity / viral release from host cell by cytolysis / virion component / structural molecule activity / RNA binding 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Escherichia virus Qbeta (ウイルス) / Escherichia virus Qbeta (ウイルス) /  Bacteriophage Q-beta (ファージ) Bacteriophage Q-beta (ファージ) | |||||||||||||||
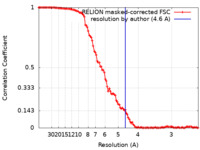
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||||||||
 データ登録者 データ登録者 | Chang JY / Zhang J | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2022 ジャーナル: Viruses / 年: 2022タイトル: Structural Assembly of Qβ Virion and Its Diverse Forms of Virus-like Particles. 著者: Jeng-Yih Chang / Karl V Gorzelnik / Jirapat Thongchol / Junjie Zhang /  要旨: The coat proteins (CPs) of single-stranded RNA bacteriophages (ssRNA phages) directly assemble around the genomic RNA (gRNA) to form a near-icosahedral capsid with a single maturation protein (Mat) ...The coat proteins (CPs) of single-stranded RNA bacteriophages (ssRNA phages) directly assemble around the genomic RNA (gRNA) to form a near-icosahedral capsid with a single maturation protein (Mat) that binds the gRNA and interacts with the retractile pilus during infection of the host. Understanding the assembly of ssRNA phages is essential for their use in biotechnology, such as RNA protection and delivery. Here, we present the complete gRNA model of the ssRNA phage Qβ, revealing that the 3' untranslated region binds to the Mat and the 4127 nucleotides fold domain-by-domain, and is connected through long-range RNA-RNA interactions, such as kissing loops. Thirty-three operator-like RNA stem-loops are located and primarily interact with the asymmetric A/B CP-dimers, suggesting a pathway for the assembly of the virions. Additionally, we have discovered various forms of the virus-like particles (VLPs), including the canonical = 3 icosahedral, larger = 4 icosahedral, prolate, oblate forms, and a small prolate form elongated along the 3-fold axis. These particles are all produced during a normal infection, as well as when overexpressing the CPs. When overexpressing the shorter RNA fragments encoding only the CPs, we observed an increased percentage of the smaller VLPs, which may be sufficient to encapsidate a shorter RNA. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23336.map.gz emd_23336.map.gz | 22.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23336-v30.xml emd-23336-v30.xml emd-23336.xml emd-23336.xml | 18.4 KB 18.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23336_fsc.xml emd_23336_fsc.xml | 10.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23336.png emd_23336.png | 273.7 KB | ||
| その他 |  emd_23336_additional_1.map.gz emd_23336_additional_1.map.gz | 97.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23336 http://ftp.pdbj.org/pub/emdb/structures/EMD-23336 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23336 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23336 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23336_validation.pdf.gz emd_23336_validation.pdf.gz | 464.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23336_full_validation.pdf.gz emd_23336_full_validation.pdf.gz | 464.4 KB | 表示 | |
| XML形式データ |  emd_23336_validation.xml.gz emd_23336_validation.xml.gz | 10.4 KB | 表示 | |
| CIF形式データ |  emd_23336_validation.cif.gz emd_23336_validation.cif.gz | 14.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23336 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23336 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23336 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23336 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23336.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23336.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Qbeta Virion | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.25 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
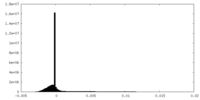
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened Map
| ファイル | emd_23336_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened Map | ||||||||||||
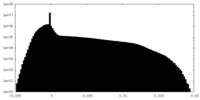
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Escherichia virus Qbeta
| 全体 | 名称:  Escherichia virus Qbeta (ウイルス) Escherichia virus Qbeta (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Escherichia virus Qbeta
| 超分子 | 名称: Escherichia virus Qbeta / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 39803 / 生物種: Escherichia virus Qbeta / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Genomic RNA
| 分子 | 名称: Genomic RNA / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Escherichia virus Qbeta (ウイルス) Escherichia virus Qbeta (ウイルス) |
| 分子量 | 理論値: 1.3520166249999999 MDa |
| 配列 | 文字列: GGGGACCCCC UUUAGGGGGU CACCUCACAC AGCAGUACUU CACUGAGUAU AAGAGGACAU AUGCCUAAAU UACCGCGUGG UCUGCGUUU CGGAGCCGAU AAUGAAAUUC UUAAUGAUUU UCAGGAGCUC UGGUUUCCAG ACCUCUUUAU CGAAUCUUCC G ACACGCAU ...文字列: GGGGACCCCC UUUAGGGGGU CACCUCACAC AGCAGUACUU CACUGAGUAU AAGAGGACAU AUGCCUAAAU UACCGCGUGG UCUGCGUUU CGGAGCCGAU AAUGAAAUUC UUAAUGAUUU UCAGGAGCUC UGGUUUCCAG ACCUCUUUAU CGAAUCUUCC G ACACGCAU CCGUGGUACA CACUGAAGGG UCGUGUGUUG AACGCCCACC UUGAUGAUCG UCUACCUAAU GUAGGCGGUC GC CAGGUAA GGCGCACUCC ACAUCGCGUC ACCGUUCCGA UUGCCUCUUC AGGCCUUCGU CCGGUAACAA CCGUUCAGUA UGA UCCCGC AGCACUAUCG UUCUUAUUGA ACGCUCGUGU UGACUGGGAU UUCGGUAAUG GCGAUAGUGC GAACCUUGUC AUUA AUGAC UUUCUGUUUC GCACCUUUGC ACCUAAGGAG UUUGAUUUUU CGAACUCCUU AGUUCCUCGU UAUACUCAGG CCUUC UCCG CGUUUAAUGC CAAGUAUGGC ACUAUGAUCG GCGAAGGGCU CGAGACUAUA AAAUAUCUCG GGCUUUUACU GCGCAG ACU GCGUGAGGGU UACCGCGCUG UUAAGCGUGG CGAUUUACGU GCUCUUCGUA GGGUUAUCCA GUCCUACCAU AAUGGUA AG UGGAAACCGG CUACUGCUGG UAAUCUCUGG CUUGAAUUUC GUUAUGGCCU UAUGCCUCUC UUUUAUGACA UCAGAGAU G UCAUGUUAGA CUGGCAGAAC CGUCAUGAUA AGAUUCAACG CCUCCUUCGG UUUUCUGUUG GUCACGGCGA GGAUUACGU UGUCGAAUUC GACAAUCUGU ACCCUGCCGU UGCUUACUUU AAACUGAAAG GGGAGAUUAC ACUCGAACGC CGUCAUCGUC AUGGCAUAU CUUACGCUAA CCGCGAAGGA UAUGCUGUUU UCGACAACGG UUCCCUUCGG CCUGUGUCCG AUUGGAAGGA G CUUGCCAC UGCAUUCAUC AAUCCGCAUG AAGUUGCUUG GGAGUUAACU CCCUACAGCU UCGUUGUUGA UUGGUUCUUG AA UGUUGGU GACAUACUUG CUCAACAAGG UCAGCUAUAU CAUAAUAUCG AUAUUGUAGA CGGCUUUGAC AGACGUGACA UCC GGCUCA AAUCUUUCAC CAUAAAAGGU GAACGAAAUG GGCGGCCUGU UAACGUUUCU GCUAGCCUGU CUGCUGUCGA UUUA UUUUA CAGCCGACUC CAUACGAGCA AUCUUCCGUU CGCUACACUA GAUCUUGAUA CCACCUUUAG UUCGUUUAAA CACGU UCUU GAUAGUAUCU UUUUAUUAAC CCAACGCGUA AAGCGUUGAA ACUUUGGGUC AAUUUGAUCA UGGCAAAAUU AGAGAC UGU UACUUUAGGU AACAUCGGGA AAGAUGGAAA ACAAACUCUG GUCCUCAAUC CGCGUGGGGU AAAUCCCACU AACGGCG UU GCCUCGCUUU CACAAGCGGG UGCAGUUCCU GCGCUGGAGA AGCGUGUUAC CGUUUCGGUA UCUCAGCCUU CUCGCAAU C GUAAGAACUA CAAGGUCCAG GUUAAGAUCC AGAACCCGAC CGCUUGCACU GCAAACGGUU CUUGUGACCC AUCCGUUAC UCGCCAGGCA UAUGCUGACG UGACCUUUUC GUUCACGCAG UAUAGUACCG AUGAGGAACG AGCUUUUGUU CGUACAGAGC UUGCUGCUC UGCUCGCUAG UCCUCUGCUG AUCGAUGCUA UUGAUCAGCU GAACCCAGCG UAUUGAACAC UGCUCAUUGC C GGUGGUGG CUCAGGGUCA AAACCCGAUC CGGUUAUUCC GGAUCCACCG AUUGAUCCGC CGCCAGGGAC AGGUAAGUAU AC CUGUCCC UUCGCAAUUU GGUCCCUAGA GGAGGUUUAC GAGCCUCCUA CUAAGAACCG ACCGUGGCCU AUCUAUAAUG CUG UUGAAC UCCAGCCUCG CGAAUUUGAU GUUGCCCUCA AAGAUCUUUU GGGCAAUACA AAGUGGCGUG AUUGGGAUUC UCGG CUUAG UUAUACCACG UUCCGCGGUU GCCGUGGCAA UGGUUAUAUU GACCUUGAUG CGACUUAUCU UGCUACUGAU CAGGC UAUG CGUGAUCAGA AGUAUGAUAU UCGCGAGGGC AAGAAACCUG GUGCUUUCGG UAACAUUGAG CGAUUCAUUU AUCUUA AGU CGAUAAAUGC UUAUUGCUCU CUUAGCGAUA UUGCGGCCUA UCACGCCGAU GGCGUGAUAG UUGGCUUUUG GCGCGAU CC AUCCAGCGGU GGUGCCAUAC CGUUUGACUU CACUAAGUUU GAUAAGACUA AAUGUCCUAU UCAAGCCGUG AUAGUCGU U CCUCGUGCUU AGUAACUAAG GAUGAAAUGC AUGUCUAAGA CAGCAUCUUC GCGUAACUCU CUCAGCGCAC AAUUGCGCC GAGCCGCGAA CACAAGAAUU GAGGUUGAAG GUAACCUCGC ACUUUCCAUU GCCAACGAUU UACUGUUGGC CUAUGGUCAG UCGCCAUUU AACUCUGAGG CUGAGUGUAU UUCAUUCAGC CCGAGAUUCG ACGGGACCCC GGAUGACUUU AGGAUAAAUU A UCUUAAAG CCGAGAUCAU GUCGAAGUAU GACGACUUCA GCCUAGGUAU UGAUACCGAA GCUGUUGCCU GGGAGAAGUU CC UGGCAGC AGAGGCUGAA UGUGCUUUAA CGAACGCUCG UCUCUAUAGG CCUGACUACA GUGAGGAUUU CAAUUUCUCA CUG GGCGAG UCAUGUAUAC ACAUGGCUCG UAGAAAAAUA GCCAAGCUAA UAGGAGAUGU UCCGUCCGUU GAGGGUAUGU UGCG UCACU GCCGAUUUUC UGGCGGUGCU ACAACAACGA AUAACCGUUC GUACGGUCAU CCGUCCUUCA AGUUUGCGCU UCCGC AAGC GUGUACGCCU CGGGCUUUGA AGUAUGUUUU AGCUCUCAGA GCUUCUACAC AUUUCGAUAU CAGAAUUUCU GAUAUU AGC CCUUUUAAUA AAGCAGUUAC UGUACCUAAG AACAGUAAGA CAGAUCGUUG UAUUGCUAUC GAACCUGGUU GGAAUAU GU UUUUCCAACU GGGUAUCGGU GGCAUUCUAC GCGAUCGGUU GCGUUGCUGG GGUAUCGAUC UGAAUGAUCA GACGAUAA A UCAGCGCCGC GCUCACGAAG GCUCCGUUAC UAAUAACUUA GCAACGGUUG AUCUCUCAGC GGCAAGCGAU UCUAUAUCU CUUGCCCUCU GUGAGCUCUU AUUGCCCCCA GGCUGGUUUG AGGUUCUUAU GGACCUCAGA UCACCUAAGG GGCGAUUGCC UGACGGUAG UGUUGUUACC UACGAGAAGA UUUCUUCUAU GGGUAACGGU UACACAUUCG AGCUCGAGUC GCUUAUUUUU G CUUCUCUC GCUCGUUCCG UUUGUGAGAU ACUGGACUUA GACUCGUCUG AGGUCACUGU UUACGGAGAC GAUAUUAUUU UA CCGUCCU GUGCAGUCCC UGCCCUCCGG GAAGUUUUUA AGUAUGUUGG UUUUACGACC AAUACUAAAA AGACUUUUUC CGA GGGGCC GUUCAGAGAG UCGUGCGGCA AGCACUACUA UUCUGGCGUA GAUGUUACUC CCUUUUACAU ACGUCACCGU AUAG UGAGU CCUGCCGAUU UAAUACUGGU UUUGAAUAAC CUAUAUCGGU GGGCCACAAU UGACGGCGUA UGGGAUCCUA GGGCC CAUU CUGUGUACCU CAAGUAUCGU AAGUUGCUGC CUAAACAGCU GCAACGUAAU ACUAUACCUG AUGGUUACGG UGAUGG UGC CCUCGUCGGA UCGGUCCUAA UCAAUCCUUU CGCGAAAAAC CGCGGGUGGA UCCGGUACGU ACCGGUGAUU ACGGACC AU ACAAGGGACC GAGAGCGCGC UGAGUUGGGG UCGUAUCUCU ACGACCUCUU CUCGCGUUGU CUCUCGGAAA GUAACGAU G GGUUGCCUCU UAGGGGUCCA UCGGGUUGCG AUUCUGCGGA UCUAUUUGCC AUCGAUCAGC UUAUCUGUAG GAGUAAUCC UACGAAGAUA AGCAGGUCUA CCGGCAAAUU CGAUAUACAG UAUAUCGCGU GCAGUAGCCG UGUUCUGGCA CCCUACGGGG UCUUCCAGG GCACGAAGGU UGCGUCUCUA CACGAGGCGU AACCUGGGAG GGCGCCAAUA UGGCGCCUAA UUGUGAAUAA A UUAUCACA AUUACUCUUA CGAGUGAGAG GGGGAUCUGC UUUGCCCUCU CUCCUCCCA |
-分子 #2: Maturation protein A2
| 分子 | 名称: Maturation protein A2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Bacteriophage Q-beta (ファージ) Bacteriophage Q-beta (ファージ) |
| 分子量 | 理論値: 48.613078 KDa |
| 配列 | 文字列: MPKLPRGLRF GADNEILNDF QELWFPDLFI ESSDTHPWYT LKGRVLNAHL DDRLPNVGGR QVRRTPHRVT VPIASSGLRP VTTVQYDPA ALSFLLNARV DWDFGNGDSA NLVINDFLFR TFAPKEFDFS NSLVPRYTQA FSAFNAKYGT MIGEGLETIK Y LGLLLRRL ...文字列: MPKLPRGLRF GADNEILNDF QELWFPDLFI ESSDTHPWYT LKGRVLNAHL DDRLPNVGGR QVRRTPHRVT VPIASSGLRP VTTVQYDPA ALSFLLNARV DWDFGNGDSA NLVINDFLFR TFAPKEFDFS NSLVPRYTQA FSAFNAKYGT MIGEGLETIK Y LGLLLRRL REGYRAVKRG DLRALRRVIQ SYHNGKWKPA TAGNLWLEFR YGLMPLFYDI RDVMLDWQNR HDKIQRLLRF SV GHGEDYV VEFDNLYPAV AYFKLKGEIT LERRHRHGIS YANREGYAVF DNGSLRPVSD WKELATAFIN PHEVAWELTP YSF VVDWFL NVGDILAQQG QLYHNIDIVD GFDRRDIRLK SFTIKGERNG RPVNVSASLS AVDLFYSRLH TSNLPFATLD LDTT FSSFK HVLDSIFLLT QRVKR |
-分子 #3: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 180 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Bacteriophage Q-beta (ファージ) Bacteriophage Q-beta (ファージ) |
| 分子量 | 理論値: 14.268071 KDa |
| 配列 | 文字列: MAKLETVTLG NIGKDGKQTL VLNPRGVNPT NGVASLSQAG AVPALEKRVT VSVSQPSRNR KNYKVQVKIQ NPTACTANGS CDPSVTRQA YADVTFSFTQ YSTDEERAFV RTELAALLAS PLLIDAIDQL NPAY |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | JEOL 3200FSC |
| 撮影 | Image recording ID: 1 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
- 電子顕微鏡法 #1~
電子顕微鏡法 #1~
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TECNAI F20 |
| 撮影 | Image recording ID: 2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z
Z Y
Y X
X