+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23243 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | BG505 SOSIP.v5.2 N241/N289 in complex with the polyclonal Fab pAbC-8 from animal Rh.33311 (Wk26 time point) | |||||||||
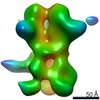
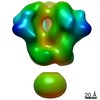
 マップデータ マップデータ | 3D map of BG505 SOSIP v5.2 N241/N289 in complex with the polyclonal Fab pAbC-8 from animal Rh.33311 (Wk26 time point). Main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV / vaccine design / BG505 / VIRAL PROTEIN / Polyclonal antibodies / EMPEM / VIRAL PROTEIN-Immune System complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated perturbation of host defense response / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell ...symbiont-mediated perturbation of host defense response / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||
 データ登録者 データ登録者 | Antanasijevic A / Sewall LM | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Polyclonal antibody responses to HIV Env immunogens resolved using cryoEM. 著者: Aleksandar Antanasijevic / Leigh M Sewall / Christopher A Cottrell / Diane G Carnathan / Luis E Jimenez / Julia T Ngo / Jennifer B Silverman / Bettina Groschel / Erik Georgeson / Jinal Bhiman ...著者: Aleksandar Antanasijevic / Leigh M Sewall / Christopher A Cottrell / Diane G Carnathan / Luis E Jimenez / Julia T Ngo / Jennifer B Silverman / Bettina Groschel / Erik Georgeson / Jinal Bhiman / Raiza Bastidas / Celia LaBranche / Joel D Allen / Jeffrey Copps / Hailee R Perrett / Kimmo Rantalainen / Fabien Cannac / Yuhe R Yang / Alba Torrents de la Peña / Rebeca Froes Rocha / Zachary T Berndsen / David Baker / Neil P King / Rogier W Sanders / John P Moore / Shane Crotty / Max Crispin / David C Montefiori / Dennis R Burton / William R Schief / Guido Silvestri / Andrew B Ward /    要旨: Engineered ectodomain trimer immunogens based on BG505 envelope glycoprotein are widely utilized as components of HIV vaccine development platforms. In this study, we used rhesus macaques to evaluate ...Engineered ectodomain trimer immunogens based on BG505 envelope glycoprotein are widely utilized as components of HIV vaccine development platforms. In this study, we used rhesus macaques to evaluate the immunogenicity of several stabilized BG505 SOSIP constructs both as free trimers and presented on a nanoparticle. We applied a cryoEM-based method for high-resolution mapping of polyclonal antibody responses elicited in immunized animals (cryoEMPEM). Mutational analysis coupled with neutralization assays were used to probe the neutralization potential at each epitope. We demonstrate that cryoEMPEM data can be used for rapid, high-resolution analysis of polyclonal antibody responses without the need for monoclonal antibody isolation. This approach allowed to resolve structurally distinct classes of antibodies that bind overlapping sites. In addition to comprehensive mapping of commonly targeted neutralizing and non-neutralizing epitopes in BG505 SOSIP immunogens, our analysis revealed that epitopes comprising engineered stabilizing mutations and of partially occupied glycosylation sites can be immunogenic. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23243.map.gz emd_23243.map.gz | 164.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23243-v30.xml emd-23243-v30.xml emd-23243.xml emd-23243.xml | 29.2 KB 29.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23243_fsc.xml emd_23243_fsc.xml | 12.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23243.png emd_23243.png | 155 KB | ||
| マスクデータ |  emd_23243_msk_1.map emd_23243_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-23243.cif.gz emd-23243.cif.gz | 7.9 KB | ||
| その他 |  emd_23243_half_map_1.map.gz emd_23243_half_map_1.map.gz emd_23243_half_map_2.map.gz emd_23243_half_map_2.map.gz | 140.2 MB 140.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23243 http://ftp.pdbj.org/pub/emdb/structures/EMD-23243 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23243 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23243 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7l90MC  7l7tC  7l7uC  7l85C  7l86C  7l87C  7l88C  7l89C  7l8aC  7l8bC  7l8cC  7l8dC  7l8eC  7l8fC  7l8gC  7l8sC  7l8tC  7l8uC  7l8wC  7l8xC  7l8yC  7l8zC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23243.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23243.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
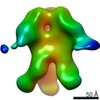
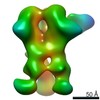
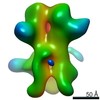
| 注釈 | 3D map of BG505 SOSIP v5.2 N241/N289 in complex with the polyclonal Fab pAbC-8 from animal Rh.33311 (Wk26 time point). Main map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23243_msk_1.map emd_23243_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: 3D map of BG505 SOSIP v5.2 N241/N289 in...
| ファイル | emd_23243_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D map of BG505 SOSIP v5.2 N241/N289 in complex with the polyclonal Fab pAbC-8 from animal Rh.33311 (Wk26 time point). Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: 3D map of BG505 SOSIP v5.2 N241/N289 in...
| ファイル | emd_23243_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D map of BG505 SOSIP v5.2 N241/N289 in complex with the polyclonal Fab pAbC-8 from animal Rh.33311 (Wk26 time point). Half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BG505 SOSIP.v5.2 N241/N289 in complex with the polyclonal Fab pAb...
| 全体 | 名称: BG505 SOSIP.v5.2 N241/N289 in complex with the polyclonal Fab pAbC-8 from animal Rh.33311 (Wk26 time point) |
|---|---|
| 要素 |
|
-超分子 #1: BG505 SOSIP.v5.2 N241/N289 in complex with the polyclonal Fab pAb...
| 超分子 | 名称: BG505 SOSIP.v5.2 N241/N289 in complex with the polyclonal Fab pAbC-8 from animal Rh.33311 (Wk26 time point) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: Polyclonal complexes were generated by incubation of isolated polyclonal Fabs with recombinantly expressed BG505 SOSIP and subsequent SEC purification. Map and model were reconstructed from ...詳細: Polyclonal complexes were generated by incubation of isolated polyclonal Fabs with recombinantly expressed BG505 SOSIP and subsequent SEC purification. Map and model were reconstructed from the immune complex dataset using the focused classification approach. |
|---|
-超分子 #2: BG505 SOSIP.v5.2 N241/N289 gp120/gp41
| 超分子 | 名称: BG505 SOSIP.v5.2 N241/N289 gp120/gp41 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-超分子 #3: polyclonal Fab pAbC-8 from animal Rh.33311
| 超分子 | 名称: polyclonal Fab pAbC-8 from animal Rh.33311 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3-#4 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: BG505 SOSIP.v5.2 N241/N289 - gp120
| 分子 | 名称: BG505 SOSIP.v5.2 N241/N289 - gp120 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 56.417125 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKRGLCCVLL LCGAVFVSPS QEIHARFRRG ARAENLWVTV YYGVPVWKDA ETTLFCASDA KAYETKKHNV WATHCCVPTD PNPQEIHLE NVTEEFNMWK NNMVEQMHTD IISLWDQSLK PCVKLTPLCV TLQCTNVTNN ITDDMRGELK NCSFNMTTEL R DKKQKVYS ...文字列: MKRGLCCVLL LCGAVFVSPS QEIHARFRRG ARAENLWVTV YYGVPVWKDA ETTLFCASDA KAYETKKHNV WATHCCVPTD PNPQEIHLE NVTEEFNMWK NNMVEQMHTD IISLWDQSLK PCVKLTPLCV TLQCTNVTNN ITDDMRGELK NCSFNMTTEL R DKKQKVYS LFYRLDVVQI NENQGNRSNN SNKEYRLINC NTSAITQACP KVSFEPIPIH YCAPAGFAIL KCKDKKFNGT GP CTNVSTV QCTHGIKPVV STQLLLNGSL AEEEVIIRSE NITNNAKNIL VQLNESVQIN CTRPNNNTRK SIRIGPGQWF YAT GDIIGD IRQAHCNVSK ATWNETLGKV VKQLRKHFGN NTIIRFANSS GGDLEVTTHS FNCGGEFFYC NTSGLFNSTW ISNT SVQGS NSTGSNDSIT LPCRIKQIIN MWQRIGQAMY APPIQGVIRC VSNITGLILT RDGGSTNSTT ETFRPGGGDM RDNWR SELY KYKVVKIEPL GVAPTRCKR |
-分子 #2: BG505 SOSIP.v5.2 N241/N289 - gp41
| 分子 | 名称: BG505 SOSIP.v5.2 N241/N289 - gp41 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 16.477719 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: LGFLGAAGST MGAASMTLTV QARNLLSGIV QQQSNLLRAP ECQQHLLKLT VWGIKQLQAR VLAVERYLRD QQLLGIWGCS GKLICCTNV PWNSTWSNRN LSEIWDNMTW LQWDKEISNY TQIIYGLLEE SQNQQEKNEQ DLLALD |
-分子 #3: Rh.33311 pAbC-8 - Heavy Chain
| 分子 | 名称: Rh.33311 pAbC-8 - Heavy Chain / タイプ: protein_or_peptide / ID: 3 詳細: This is a polyclonal antibody and the sequence is inherently unknown. Poly-alanine pseudo-model is used to depict the backbone of this antibody. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 9.209344 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) |
-分子 #4: Rh.33311 pAbC-8 - Light Chain
| 分子 | 名称: Rh.33311 pAbC-8 - Light Chain / タイプ: protein_or_peptide / ID: 4 詳細: This is a polyclonal antibody and the sequence is inherently unknown. Poly-alanine pseudo-model is used to depict the backbone of this antibody. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 7.93277 KDa |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) ...文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 52 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: TBS buffer prepared from a 10X stock | |||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: OTHER | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting time varied between 3 and 7 seconds.. | |||||||||
| 詳細 | Polyclonal complexes were generated by incubation of isolated polyclonal Fabs with recombinantly expressed BG505 SOSIP and subsequent SEC purification. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-39 / 撮影したグリッド数: 4 / 実像数: 6268 / 平均露光時間: 9.75 sec. / 平均電子線量: 44.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)