+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2287 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human TFIID binds to core promoter DNA in a reorganized structural state | |||||||||
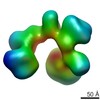
 マップデータ マップデータ | 3D reconstruction of canonical conformation of human TFIID at 35A | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TFIID / transcription / cryo-electron microscopy / TFIIA / core promoter / RNA polymerase II | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 35.0 Å | |||||||||
 データ登録者 データ登録者 | Cianfrocco MA / Kassavetis GA / Grob P / Fang J / Juven-Gershon T / Kadonaga JT / Nogales E | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2013 ジャーナル: Cell / 年: 2013タイトル: Human TFIID binds to core promoter DNA in a reorganized structural state. 著者: Michael A Cianfrocco / George A Kassavetis / Patricia Grob / Jie Fang / Tamar Juven-Gershon / James T Kadonaga / Eva Nogales /  要旨: A mechanistic description of metazoan transcription is essential for understanding the molecular processes that govern cellular decisions. To provide structural insights into the DNA recognition step ...A mechanistic description of metazoan transcription is essential for understanding the molecular processes that govern cellular decisions. To provide structural insights into the DNA recognition step of transcription initiation, we used single-particle electron microscopy (EM) to visualize human TFIID with promoter DNA. This analysis revealed that TFIID coexists in two predominant and distinct structural states that differ by a 100 Å translocation of TFIID's lobe A. The transition between these structural states is modulated by TFIIA, as the presence of TFIIA and promoter DNA facilitates the formation of a rearranged state of TFIID that enables promoter recognition and binding. DNA labeling and footprinting, together with cryo-EM studies, were used to map the locations of TATA, Initiator (Inr), motif ten element (MTE), and downstream core promoter element (DPE) promoter motifs within the TFIID-TFIIA-DNA structure. The existence of two structurally and functionally distinct forms of TFIID suggests that the different conformers may serve as specific targets for the action of regulatory factors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2287.map.gz emd_2287.map.gz | 2.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2287-v30.xml emd-2287-v30.xml emd-2287.xml emd-2287.xml | 9.6 KB 9.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2287.png emd_2287.png | 43.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2287 http://ftp.pdbj.org/pub/emdb/structures/EMD-2287 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2287 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2287 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2287_validation.pdf.gz emd_2287_validation.pdf.gz | 197 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2287_full_validation.pdf.gz emd_2287_full_validation.pdf.gz | 196.1 KB | 表示 | |
| XML形式データ |  emd_2287_validation.xml.gz emd_2287_validation.xml.gz | 5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2287 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2287 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2287 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2287 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2287.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2287.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D reconstruction of canonical conformation of human TFIID at 35A | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.48 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
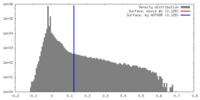
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : holo-TFIID (canonical)
| 全体 | 名称: holo-TFIID (canonical) |
|---|---|
| 要素 |
|
-超分子 #1000: holo-TFIID (canonical)
| 超分子 | 名称: holo-TFIID (canonical) / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 1.3 MDa / 理論値: 1.3 MDa |
-分子 #1: Transcription factor II-D
| 分子 | 名称: Transcription factor II-D / タイプ: protein_or_peptide / ID: 1 / Name.synonym: TFIID / コピー数: 1 / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 株: HeLa / 別称: Human / Organelle: Nucleus / 細胞中の位置: Nucleus Homo sapiens (ヒト) / 株: HeLa / 別称: Human / Organelle: Nucleus / 細胞中の位置: Nucleus |
| 分子量 | 実験値: 1.3 MDa / 理論値: 1.3 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 詳細: 20 mM HEPES pH 7.8, 0.2 mM EDTA, 4 mM MgCl2, ~1% glycerol, 1 mM DTT, 3% trehalose, 50 mM KCl, 0.05% NP-40, |
| グリッド | 詳細: 400 mesh copper grid on C-flat support carbon with a thin carbon support over 4 um holes space 2 um apart. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK II 手法: Sample was incubated on grid for 30 seconds before blotting 6.5s at -1 offset. The grid was washed in 4 ul buffer to blot again for 6.5 s at -1 offset prior to plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective and condenser lens astigmatism was corrected at 80,000 times magnification |
| 日付 | 2012年1月31日 |
| 撮影 | カテゴリ: FILM フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - スキャナー: OTHER / 実像数: 494 / 平均電子線量: 25 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 10500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 80000 |
| 試料ステージ | 試料ホルダー: Gatan 626 liquid nitrogen cooled / 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Particles were prepared within the APPION processing environment. The particles were automatically selected using Signature and extracted from phase flipped micrographs using values calculated by CTFFIND3. After projection matching within EMAN2/Sparx, the final map was further refined in FREALIGN |
|---|---|
| CTF補正 | 詳細: Per micrograph for phase flipping, per particles for refinement in FREALIGN |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 35.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: Sparx, EMAN2, FREALIGN / 使用した粒子像数: 11898 |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)