+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22341 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Streptococcus thermophilus SHP pheromone receptor Rgg3 in complex with SHP3 | |||||||||
 マップデータ マップデータ | Sharpened map generated by cryoSPARC | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA binding transcription factor / quorum sensing / RRNPP / pheromone binding / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 | Transcription activator MutR, C-terminal / HTH-type transcriptional regulator Rgg, C-terminal domain / : / Cro/C1-type HTH domain profile. / Cro/C1-type helix-turn-helix domain / Lambda repressor-like, DNA-binding domain superfamily / Tetratricopeptide-like helical domain superfamily / DNA binding / Positive transcriptional regulator MutR family 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) / Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) /  Streptococcus thermophilus (strain ATCC BAA-250 / LMG 18311) (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) / Streptococcus thermophilus (strain ATCC BAA-250 / LMG 18311) (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) /  Streptococcus thermophilus CNRZ1066 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus CNRZ1066 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) | |||||||||
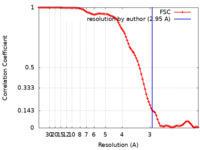
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.95 Å | |||||||||
 データ登録者 データ登録者 | Petrou VI / Capodagli GC | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2020 ジャーナル: Proc Natl Acad Sci U S A / 年: 2020タイトル: Structure-function studies of Rgg binding to pheromones and target promoters reveal a model of transcription factor interplay. 著者: Glenn C Capodagli / Kaitlyn M Tylor / Jason T Kaelber / Vasileios I Petrou / Michael J Federle / Matthew B Neiditch /  要旨: Regulator gene of glucosyltransferase (Rgg) family proteins, such as Rgg2 and Rgg3, have emerged as primary quorum-sensing regulated transcription factors in species, controlling virulence, ...Regulator gene of glucosyltransferase (Rgg) family proteins, such as Rgg2 and Rgg3, have emerged as primary quorum-sensing regulated transcription factors in species, controlling virulence, antimicrobial resistance, and biofilm formation. Rgg2 and Rgg3 function is regulated by their interaction with oligopeptide quorum-sensing signals called short hydrophobic peptides (SHPs). The molecular basis of Rgg-SHP and Rgg-target DNA promoter specificity was unknown. To close this gap, we determined the cryoelectron microscopy (cryo-EM) structure of Rgg3 bound to its quorum-sensing signal, SHP3, and the X-ray crystal structure of Rgg3 alone. Comparison of these structures with that of an Rgg in complex with cyclosporin A (CsA), an inhibitor of SHP-induced Rgg activity, reveals the molecular basis of CsA function. Furthermore, to determine how Rgg proteins recognize DNA promoters, we determined X-ray crystal structures of both Rgg2 and Rgg3 in complex with their target DNA promoters. The physiological importance of observed Rgg-DNA interactions was dissected using in vivo genetic experiments and in vitro biochemical assays. Based on these structure-function studies, we present a revised unifying model of Rgg regulatory interplay. In contrast to existing models, where Rgg2 proteins are transcriptional activators and Rgg3 proteins are transcriptional repressors, we propose that both are capable of transcriptional activation. However, when Rgg proteins with different activation requirements compete for the same DNA promoters, those with more stringent activation requirements function as repressors by blocking promoter access of SHP-bound conformationally active Rgg proteins. While a similar gene expression regulatory scenario has not been previously described, in all likelihood it is not unique to streptococci. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22341.map.gz emd_22341.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22341-v30.xml emd-22341-v30.xml emd-22341.xml emd-22341.xml | 22.2 KB 22.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22341_fsc.xml emd_22341_fsc.xml | 10 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22341.png emd_22341.png | 80.9 KB | ||
| マスクデータ |  emd_22341_msk_1.map emd_22341_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-22341.cif.gz emd-22341.cif.gz | 6.4 KB | ||
| その他 |  emd_22341_additional_1.map.gz emd_22341_additional_1.map.gz emd_22341_half_map_1.map.gz emd_22341_half_map_1.map.gz emd_22341_half_map_2.map.gz emd_22341_half_map_2.map.gz | 32.2 MB 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22341 http://ftp.pdbj.org/pub/emdb/structures/EMD-22341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22341 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ji0MC  6w1aC  6w1eC  6w1fC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10535 (タイトル: Single particle cryoEM of Streptococcus thermophilus SHP pheromone receptor Rgg3 in complex with SHP3 EMPIAR-10535 (タイトル: Single particle cryoEM of Streptococcus thermophilus SHP pheromone receptor Rgg3 in complex with SHP3Data size: 3.0 TB Data #1: Unaligned multi-frame micrographs of Rgg3-SHP3 complex [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22341.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22341.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map generated by cryoSPARC | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.038 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_22341_msk_1.map emd_22341_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Unsharpened map generated by cryoSPARC
| ファイル | emd_22341_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map generated by cryoSPARC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 2 generated by cryoSPARC
| ファイル | emd_22341_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 generated by cryoSPARC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half-map 1 generated by cryoSPARC
| ファイル | emd_22341_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 generated by cryoSPARC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Streptococcus thermophilus SHP pheromone receptor Rgg3 in complex...
| 全体 | 名称: Streptococcus thermophilus SHP pheromone receptor Rgg3 in complex with SHP3 |
|---|---|
| 要素 |
|
-超分子 #1: Streptococcus thermophilus SHP pheromone receptor Rgg3 in complex...
| 超分子 | 名称: Streptococcus thermophilus SHP pheromone receptor Rgg3 in complex with SHP3 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス)器官: CNRZ1066 |
-分子 #1: Positive transcriptional regulator MutR family
| 分子 | 名称: Positive transcriptional regulator MutR family / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (strain ATCC BAA-250 / LMG 18311) (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (strain ATCC BAA-250 / LMG 18311) (ストレプトコッカス・サリバリウス 亜種 サーモフィラス)株: ATCC BAA-250 / LMG 18311 |
| 分子量 | 理論値: 33.242078 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKSKLGSTLR KVRNGKQISI CSVADEHLSK SQISRFERGE SEISCIRLIN ILDKLHITLD EFLILHDEDY TKTESFANLV QYIRKQYSL QNINNIQSLL SDSSNYTLDP FEKTMVKSIL HTMDSSIIPS DDELLQLADY LFKVEKWGYY EIILLGNCVR T IDYNSVFL ...文字列: MKSKLGSTLR KVRNGKQISI CSVADEHLSK SQISRFERGE SEISCIRLIN ILDKLHITLD EFLILHDEDY TKTESFANLV QYIRKQYSL QNINNIQSLL SDSSNYTLDP FEKTMVKSIL HTMDSSIIPS DDELLQLADY LFKVEKWGYY EIILLGNCVR T IDYNSVFL LTKEMLNNYI YSSLNKTNKR IVTQLAINCL ILSIDMEEFT NCFYLIDEIK ALLDNELNFY EQTVFLYATG YF EFKRWQS TSGIEKMKQA IQVLDILGED NLKLHYTIHF DKLINNK UniProtKB: Positive transcriptional regulator MutR family |
-分子 #2: SHP3
| 分子 | 名称: SHP3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus CNRZ1066 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus CNRZ1066 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 798.969 Da |
| 組換発現 | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: DIIIIVGG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 3 sec. / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV 詳細: 2.5 microliters Rgg3St-SHP3 was applied to glow-discharged UltraAuFoil (1.2/1.3) 300-mesh grids (Quantifoil), blotted with filter paper for 3-3.5 s, and flash-frozen by plunging in liquid ...詳細: 2.5 microliters Rgg3St-SHP3 was applied to glow-discharged UltraAuFoil (1.2/1.3) 300-mesh grids (Quantifoil), blotted with filter paper for 3-3.5 s, and flash-frozen by plunging in liquid ethane cooled with liquid nitrogen. Grids were stored in liquid nitrogen.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 2 / 実像数: 1464 / 平均露光時間: 8.0 sec. / 平均電子線量: 47.13 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 0.5 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)