+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22236 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of EcmrR-RNAP-promoter initial transcribing complex with 3-nt RNA transcript (EcmrR-RPitc-3nt) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Transcriptional factor / TRANSCRIPTION TRANSFERASE-DNA complex / promoter / multidrug recognition / TRANSCRIPTION / TRANSFERASE-DNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sigma factor antagonist complex / regulation of DNA-templated transcription initiation / sigma factor activity / cytosolic DNA-directed RNA polymerase complex / DNA-directed RNA polymerase complex / DNA-templated transcription initiation / ribonucleoside binding / DNA-directed RNA polymerase / DNA-directed RNA polymerase activity / response to heat ...sigma factor antagonist complex / regulation of DNA-templated transcription initiation / sigma factor activity / cytosolic DNA-directed RNA polymerase complex / DNA-directed RNA polymerase complex / DNA-templated transcription initiation / ribonucleoside binding / DNA-directed RNA polymerase / DNA-directed RNA polymerase activity / response to heat / protein dimerization activity / transcription cis-regulatory region binding / negative regulation of DNA-templated transcription / DNA-templated transcription / regulation of DNA-templated transcription / magnesium ion binding / DNA binding / zinc ion binding / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.5 Å | |||||||||
 データ登録者 データ登録者 | Yang Y / Liu C | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structural visualization of transcription activated by a multidrug-sensing MerR family regulator. 著者: Yang Yang / Chang Liu / Wei Zhou / Wei Shi / Ming Chen / Baoyue Zhang / David G Schatz / Yangbo Hu / Bin Liu /   要旨: Bacterial RNA polymerase (RNAP) holoenzyme initiates transcription by recognizing the conserved -35 and -10 promoter elements that are optimally separated by a 17-bp spacer. The MerR family of ...Bacterial RNA polymerase (RNAP) holoenzyme initiates transcription by recognizing the conserved -35 and -10 promoter elements that are optimally separated by a 17-bp spacer. The MerR family of transcriptional regulators activate suboptimal 19-20 bp spacer promoters in response to myriad cellular signals, ranging from heavy metals to drug-like compounds. The regulation of transcription by MerR family regulators is not fully understood. Here we report one crystal structure of a multidrug-sensing MerR family regulator EcmrR and nine cryo-electron microscopy structures that capture the EcmrR-dependent transcription process from promoter opening to initial transcription to RNA elongation. These structures reveal that EcmrR is a dual ligand-binding factor that reshapes the suboptimal 19-bp spacer DNA to enable optimal promoter recognition, sustains promoter remodeling to stabilize initial transcribing complexes, and finally dissociates from the promoter to reverse DNA remodeling and facilitate the transition to elongation. Our findings yield a comprehensive model for transcription regulation by MerR family factors and provide insights into the transition from transcription initiation to elongation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22236.map.gz emd_22236.map.gz | 204.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22236-v30.xml emd-22236-v30.xml emd-22236.xml emd-22236.xml | 25.7 KB 25.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_22236.png emd_22236.png | 185.1 KB | ||
| Filedesc metadata |  emd-22236.cif.gz emd-22236.cif.gz | 9.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22236 http://ftp.pdbj.org/pub/emdb/structures/EMD-22236 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22236 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22236 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6xl9MC  6wl5C  6xl5C  6xl6C  6xlaC  6xljC  6xlkC  6xllC  6xlmC  6xlnC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22236.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22236.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.895 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
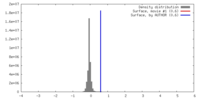
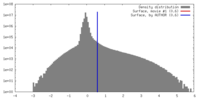
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : EcmrR-RNAP-promoter initial transcribing complex with 3-nt RNA tr...
+超分子 #1: EcmrR-RNAP-promoter initial transcribing complex with 3-nt RNA tr...
+分子 #1: DNA-directed RNA polymerase subunit alpha
+分子 #2: DNA-directed RNA polymerase subunit beta
+分子 #3: DNA-directed RNA polymerase subunit beta'
+分子 #4: RNA polymerase sigma factor RpoD
+分子 #8: MerR family transcriptional regulator EcmrR
+分子 #5: synthetic non-template strand DNA (54-MER)
+分子 #7: synthetic template strand DNA (54-MER)
+分子 #6: RNA (5'-D(*(GTP))-R(P*AP*G)-3')
+分子 #9: ZINC ION
+分子 #10: MAGNESIUM ION
+分子 #11: TETRAPHENYLANTIMONIUM ION
+分子 #12: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-40 / 平均露光時間: 30.0 sec. / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)