+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22078 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic Analysis | |||||||||
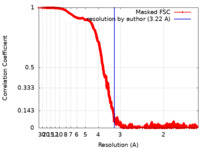
 マップデータ マップデータ | SARS-CoV2 SPIKE protein reconstruction at 3.22 Angstrom | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane ...Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / entry receptor-mediated virion attachment to host cell / receptor-mediated endocytosis of virus by host cell / Attachment and Entry / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / receptor ligand activity / host cell surface receptor binding / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.22 Å | |||||||||
 データ登録者 データ登録者 | Herrera NG / Morano NC / Celikgil A / Georgiev GI / Malonis R / Lee JH / Tong K / Vergnolle O / Massimi A / Yen LY ...Herrera NG / Morano NC / Celikgil A / Georgiev GI / Malonis R / Lee JH / Tong K / Vergnolle O / Massimi A / Yen LY / Noble AJ / Kopylov M / Bonanno JB / Garrett-Thompson SC / Hayes DB / Brenowitz M / Garforth SJ / Eng ET / Lai JR / Almo SC | |||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2020 ジャーナル: bioRxiv / 年: 2020タイトル: Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic Analysis. 著者: Natalia G Herrera / Nicholas C Morano / Alev Celikgil / George I Georgiev / Ryan J Malonis / James H Lee / Karen Tong / Olivia Vergnolle / Aldo B Massimi / Laura Y Yen / Alex J Noble / ...著者: Natalia G Herrera / Nicholas C Morano / Alev Celikgil / George I Georgiev / Ryan J Malonis / James H Lee / Karen Tong / Olivia Vergnolle / Aldo B Massimi / Laura Y Yen / Alex J Noble / Mykhailo Kopylov / Jeffrey B Bonanno / Sarah C Garrett-Thomson / David B Hayes / Robert H Bortz / Ariel S Wirchnianski / Catalina Florez / Ethan Laudermilch / Denise Haslwanter / J Maximilian Fels / M Eugenia Dieterle / Rohit K Jangra / Jason Barnhill / Amanda Mengotto / Duncan Kimmel / Johanna P Daily / Liise-Anne Pirofski / Kartik Chandran / Michael Brenowitz / Scott J Garforth / Edward T Eng / Jonathan R Lai / Steven C Almo /  要旨: Coronavirus disease 2019 ( ) is a global health crisis caused by the novel severe acute respiratory syndrome coronavirus 2 ( ), and there is a critical need to produce large quantities of high- ...Coronavirus disease 2019 ( ) is a global health crisis caused by the novel severe acute respiratory syndrome coronavirus 2 ( ), and there is a critical need to produce large quantities of high-quality SARS-CoV-2 Spike ( ) protein for use in both clinical and basic science settings. To address this need, we have evaluated the expression and purification of two previously reported S protein constructs in Expi293F and ExpiCHO-S cells, two different cell lines selected for increased expression of secreted glycoproteins. We show that ExpiCHO-S cells produce enhanced yields of both SARS-CoV-2 S proteins. Biochemical, biophysical, and structural ( ) characterization of the SARS-CoV-2 S proteins produced in both cell lines demonstrate that the reported purification strategy yields high quality S protein (non-aggregated, uniform material with appropriate biochemical and biophysical properties). Importantly, we show that multiple preparations of these two recombinant S proteins from either cell line exhibit identical behavior in two different serology assays. We also evaluate the specificity of S protein-mediated host cell binding by examining interactions with proposed binding partners in the human secretome. In addition, the antigenicity of these proteins is demonstrated by standard ELISAs, and in a flexible protein microarray format. Collectively, we establish an array of metrics for ensuring the production of high-quality S protein to support clinical, biological, biochemical, structural and mechanistic studies to combat the global pandemic caused by SARS-CoV-2. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22078.map.gz emd_22078.map.gz | 204 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22078-v30.xml emd-22078-v30.xml emd-22078.xml emd-22078.xml | 16.3 KB 16.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22078_fsc.xml emd_22078_fsc.xml | 1 MB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22078.png emd_22078.png | 142.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22078 http://ftp.pdbj.org/pub/emdb/structures/EMD-22078 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22078 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22078 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22078_validation.pdf.gz emd_22078_validation.pdf.gz | 477 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22078_full_validation.pdf.gz emd_22078_full_validation.pdf.gz | 476.6 KB | 表示 | |
| XML形式データ |  emd_22078_validation.xml.gz emd_22078_validation.xml.gz | 121.8 KB | 表示 | |
| CIF形式データ |  emd_22078_validation.cif.gz emd_22078_validation.cif.gz | 332.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22078 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22078 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22078 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22078 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6x6pMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10433 (タイトル: Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic Analysis EMPIAR-10433 (タイトル: Characterization of the SARS-CoV-2 S Protein: Biophysical, Biochemical, Structural, and Antigenic AnalysisData size: 484.3 Data #1: Unaligned multi frame micrographs of SARS CoV-2 SPIKE protein produced in CHO cells [micrographs - multiframe] Data #2: Processed data - aligned frame sum with dose-weighting [micrographs - single frame]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22078.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22078.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | SARS-CoV2 SPIKE protein reconstruction at 3.22 Angstrom | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.058 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Severe acute respiratory syndrome coronavirus 2
| 全体 | 名称:  |
|---|---|
| 要素 |
|
-超分子 #1: Severe acute respiratory syndrome coronavirus 2
| 超分子 | 名称: Severe acute respiratory syndrome coronavirus 2 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1 / NCBI-ID: 2697049 / 生物種: Severe acute respiratory syndrome coronavirus 2 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: Yes / ウイルス・中空状態: Yes |
|---|---|
| Host system | 生物種:  |
-分子 #1: Spike glycoprotein
| 分子 | 名称: Spike glycoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 140.824406 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: CVNLTTRTQL PPAYTNSFTR GVYYPDKVFR SSVLHSTQDL FLPFFSNVTW FHAIHVSGTN GTKRFDNPVL PFNDGVYFAS TEKSNIIRG WIFGTTLDSK TQSLLIVNNA TNVVIKVCEF QFCNDPFLGV YYHKNNKSWM ESEFRVYSSA NNCTFEYVSQ P FLMDLEGK ...文字列: CVNLTTRTQL PPAYTNSFTR GVYYPDKVFR SSVLHSTQDL FLPFFSNVTW FHAIHVSGTN GTKRFDNPVL PFNDGVYFAS TEKSNIIRG WIFGTTLDSK TQSLLIVNNA TNVVIKVCEF QFCNDPFLGV YYHKNNKSWM ESEFRVYSSA NNCTFEYVSQ P FLMDLEGK QGNFKNLREF VFKNIDGYFK IYSKHTPINL VRDLPQGFSA LEPLVDLPIG INITRFQTLL ALHRSYLTPG DS SSGWTAG AAAYYVGYLQ PRTFLLKYNE NGTITDAVDC ALDPLSETKC TLKSFTVEKG IYQTSNFRVQ PTESIVRFPN ITN LCPFGE VFNATRFASV YAWNRKRISN CVADYSVLYN SASFSTFKCY GVSPTKLNDL CFTNVYADSF VIRGDEVRQI APGQ TGKIA DYNYKLPDDF TGCVIAWNSN NLDSKVGGNY NYLYRLFRKS NLKPFERDIS TEIYQAGSTP CNGVEGFNCY FPLQS YGFQ PTNGVGYQPY RVVVLSFELL HAPATVCGPK KSTNLVKNKC VNFNFNGLTG TGVLTESNKK FLPFQQFGRD IADTTD AVR DPQTLEILDI TPCSFGGVSV ITPGTNTSNQ VAVLYQDVNC TEVPVAIHAD QLTPTWRVYS TGSNVFQTRA GCLIGAE HV NNSYECDIPI GAGICASYQT QTNSPGSASS VASQSIIAYT MSLGAENSVA YSNNSIAIPT NFTISVTTEI LPVSMTKT S VDCTMYICGD STECSNLLLQ YGSFCTQLNR ALTGIAVEQD KNTQEVFAQV KQIYKTPPIK DFGGFNFSQI LPDPSKPSK RSFIEDLLFN KVTLADAGFI KQYGDCLGDI AARDLICAQK FNGLTVLPPL LTDEMIAQYT SALLAGTITS GWTFGAGAAL QIPFAMQMA YRFNGIGVTQ NVLYENQKLI ANQFNSAIGK IQDSLSSTAS ALGKLQDVVN QNAQALNTLV KQLSSNFGAI S SVLNDILS RLDPPEAEVQ IDRLITGRLQ SLQTYVTQQL IRAAEIRASA NLAATKMSEC VLGQSKRVDF CGKGYHLMSF PQ SAPHGVV FLHVTYVPAQ EKNFTTAPAI CHDGKAHFPR EGVFVSNGTH WFVTQRNFYE PQIITTDNTF VSGNCDVVIG IVN NTVYDP LQPELDSFKE ELDKYFKNHT SPDVDLGDIS GINASVVNIQ KEIDRLNEVA KNLNESLIDL QELGKYEQGS GYIP EAPRD GQAYVRKDGE WVLLSTFLGR SLEVLFQGPG HHHHHHHHSA WSHPQFEKGG GSGGGGSGGS AWSHPQFEK |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 33 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: PLASMA CLEANING | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 1131 / 平均露光時間: 2.5 sec. / 平均電子線量: 66.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー