+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22067 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bovine Cardiac Myosin in Complex with Chicken Skeletal Actin and Human Cardiac Tropomyosin in the Rigor State | ||||||||||||||||||||||||
 マップデータ マップデータ | Cryo-EM map of the cardiac actin-myosin-tropomyosin complex. | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード | myosin / tropomyosin / actin / cardiac / CONTRACTILE PROTEIN | ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of heart rate by epinephrine / muscle thin filament tropomyosin / bleb / Striated Muscle Contraction / negative regulation of vascular associated smooth muscle cell migration / regulation of muscle contraction / muscle filament sliding / myosin filament / adult heart development / ruffle organization ...positive regulation of heart rate by epinephrine / muscle thin filament tropomyosin / bleb / Striated Muscle Contraction / negative regulation of vascular associated smooth muscle cell migration / regulation of muscle contraction / muscle filament sliding / myosin filament / adult heart development / ruffle organization / negative regulation of vascular associated smooth muscle cell proliferation / positive regulation of ATP-dependent activity / Striated Muscle Contraction / myosin II complex / structural constituent of muscle / ventricular cardiac muscle tissue morphogenesis / regulation of heart contraction / sarcomere organization / microfilament motor activity / myofibril / skeletal muscle thin filament assembly / striated muscle thin filament / positive regulation of cell adhesion / Smooth Muscle Contraction / skeletal muscle fiber development / cardiac muscle contraction / stress fiber / positive regulation of stress fiber assembly / cytoskeleton organization / cytoskeletal protein binding / sarcomere / negative regulation of cell migration / actin filament organization / actin filament / ruffle membrane / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / wound healing / cellular response to reactive oxygen species / structural constituent of cytoskeleton / actin filament binding / actin cytoskeleton / actin binding / regulation of cell shape / calmodulin binding / cytoskeleton / hydrolase activity / protein heterodimerization activity / protein homodimerization activity / ATP binding / identical protein binding / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | ||||||||||||||||||||||||
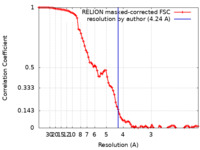
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.24 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Doran MH / Lehman W / Bullitt E | ||||||||||||||||||||||||
| 資金援助 |  米国, European Union, 7件 米国, European Union, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Biophys J / 年: 2020 ジャーナル: Biophys J / 年: 2020タイトル: Cryo-EM and Molecular Docking Shows Myosin Loop 4 Contacts Actin and Tropomyosin on Thin Filaments. 著者: Matthew H Doran / Elumalai Pavadai / Michael J Rynkiewicz / Jonathan Walklate / Esther Bullitt / Jeffrey R Moore / Michael Regnier / Michael A Geeves / William Lehman /   要旨: The motor protein myosin drives muscle and nonmuscle motility by binding to and moving along actin of thin filaments. Myosin binding to actin also modulates interactions of the regulatory protein, ...The motor protein myosin drives muscle and nonmuscle motility by binding to and moving along actin of thin filaments. Myosin binding to actin also modulates interactions of the regulatory protein, tropomyosin, on thin filaments, and conversely tropomyosin affects myosin binding to actin. Insight into this reciprocity will facilitate a molecular level elucidation of tropomyosin regulation of myosin interaction with actin in muscle contraction, and in turn, promote better understanding of nonmuscle cell motility. Indeed, experimental approaches such as fiber diffraction, cryoelectron microscopy, and three-dimensional reconstruction have long been used to define regulatory interaction of tropomyosin and myosin on actin at a structural level. However, their limited resolution has not proven sufficient to determine tropomyosin and myosin contacts at an atomic-level and thus to fully substantiate possible functional contributions. To overcome this deficiency, we have followed a hybrid approach by performing new cryogenic electron microscopy reconstruction of myosin-S1-decorated F-actin-tropomyosin together with atomic scale protein-protein docking of tropomyosin to the EM models. Here, cryo-EM data were derived from filaments reconstituted with α1-actin, cardiac αα-tropomyosin, and masseter muscle β-myosin complexes; masseter myosin, which shares sequence identity with β-cardiac myosin-heavy chain, was used because of its stability in vitro. The data were used to build an atomic model of the tropomyosin cable that fits onto the actin filament between the tip of the myosin head and a cleft on the innermost edge of actin subunits. The docking and atomic scale fitting showed multiple discrete interactions of myosin loop 4 and acidic residues on successive 39-42 residue-long tropomyosin pseudorepeats. The contacts between S1 and tropomyosin on actin appear to compete with and displace ones normally found between actin and tropomyosin on myosin-free thin filaments in relaxed muscle, thus restructuring the filament during myosin-induced activation. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22067.map.gz emd_22067.map.gz | 9.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22067-v30.xml emd-22067-v30.xml emd-22067.xml emd-22067.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22067_fsc.xml emd_22067_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22067.png emd_22067.png | 222.1 KB | ||
| Filedesc metadata |  emd-22067.cif.gz emd-22067.cif.gz | 8.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22067 http://ftp.pdbj.org/pub/emdb/structures/EMD-22067 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22067 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22067 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22067_validation.pdf.gz emd_22067_validation.pdf.gz | 488.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22067_full_validation.pdf.gz emd_22067_full_validation.pdf.gz | 488.4 KB | 表示 | |
| XML形式データ |  emd_22067_validation.xml.gz emd_22067_validation.xml.gz | 10.8 KB | 表示 | |
| CIF形式データ |  emd_22067_validation.cif.gz emd_22067_validation.cif.gz | 14.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22067 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22067 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22067 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22067 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22067.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22067.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the cardiac actin-myosin-tropomyosin complex. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Helical complex of bovine S1 cardiac myosin and human cardiac tro...
| 全体 | 名称: Helical complex of bovine S1 cardiac myosin and human cardiac tropomyosin-decorated chicken skeletal actin filaments |
|---|---|
| 要素 |
|
-超分子 #1: Helical complex of bovine S1 cardiac myosin and human cardiac tro...
| 超分子 | 名称: Helical complex of bovine S1 cardiac myosin and human cardiac tropomyosin-decorated chicken skeletal actin filaments タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Bovine myosin was isolated from the masseter muscle and the S1 fragment was generated through proteolytic cleavage. |
|---|---|
| 分子量 | 理論値: 403.22 KDa |
-超分子 #3: S1 cardiac myosin
| 超分子 | 名称: S1 cardiac myosin / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #4: cardiac tropomyosin
| 超分子 | 名称: cardiac tropomyosin / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #2: skeletal actin filaments
| 超分子 | 名称: skeletal actin filaments / タイプ: organelle_or_cellular_component / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Actin, alpha skeletal muscle
| 分子 | 名称: Actin, alpha skeletal muscle / タイプ: protein_or_peptide / ID: 1 詳細: Residues 1-9 and 377 were disordered and were not modeled. コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 42.096953 KDa |
| 配列 | 文字列: MCDEDETTAL VCDNGSGLVK AGFAGDDAPR AVFPSIVGRP RHQGVMVGMG QKDSYVGDEA QSKRGILTLK YPIEHGIITN WDDMEKIWH HTFYNELRVA PEEHPTLLTE APLNPKANRE KMTQIMFETF NVPAMYVAIQ AVLSLYASGR TTGIVLDSGD G VTHNVPIY ...文字列: MCDEDETTAL VCDNGSGLVK AGFAGDDAPR AVFPSIVGRP RHQGVMVGMG QKDSYVGDEA QSKRGILTLK YPIEHGIITN WDDMEKIWH HTFYNELRVA PEEHPTLLTE APLNPKANRE KMTQIMFETF NVPAMYVAIQ AVLSLYASGR TTGIVLDSGD G VTHNVPIY EGYALPHAIM RLDLAGRDLT DYLMKILTER GYSFVTTAER EIVRDIKEKL CYVALDFENE MATAASSSSL EK SYELPDG QVITIGNERF RCPETLFQPS FIGMESAGIH ETTYNSIMKC DIDIRKDLYA NNVMSGGTTM YPGIADRMQK EIT ALAPST MKIKIIAPPE RKYSVWIGGS ILASLSTFQQ MWITKQEYDE AGPSIVHRKC F UniProtKB: Actin, alpha skeletal muscle |
-分子 #2: Tropomyosin alpha-1 chain
| 分子 | 名称: Tropomyosin alpha-1 chain / タイプ: protein_or_peptide / ID: 2 詳細: Only the central section (residues 45-210) was modeled. コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 32.763621 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDAIKKKMQM LKLDKENALD RAEQAEADKK AAEDRSKQLE DELVSLQKKL KGTEDELDKY SEALKDAQEK LELAEKKATD AEADVASLN RRIQLVEEEL DRAQERLATA LQKLEEAEKA ADESERGMKV IESRAQKDEE KMEIQEIQLK EAKHIAEDAD R KYEEVARK ...文字列: MDAIKKKMQM LKLDKENALD RAEQAEADKK AAEDRSKQLE DELVSLQKKL KGTEDELDKY SEALKDAQEK LELAEKKATD AEADVASLN RRIQLVEEEL DRAQERLATA LQKLEEAEKA ADESERGMKV IESRAQKDEE KMEIQEIQLK EAKHIAEDAD R KYEEVARK LVIIESDLER AEERAELSEG KCAELEEELK TVTNNLKSLE AQAEKYSQKE DRYEEEIKVL SDKLKEAETR AE FAERSVT KLEKSIDDLE DELYAQKLKY KAISEELDHA LNDMTSI UniProtKB: Tropomyosin alpha-1 chain |
-分子 #3: Myosin-7
| 分子 | 名称: Myosin-7 / タイプ: protein_or_peptide / ID: 3 詳細: Residues 1-35, 200-216, 624-641, and 777-850 were disordered and not modeled. コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 97.446898 KDa |
| 配列 | 文字列: MVDAEMAAFG EAAPYLRKSE KERLEAQTRP FDLKKDVFVP DDKEEFVKAT ILSREGGKVT AETEHGKTVT VKEDQVLQQN PPKFDKIED MAMLTFLHEP AVLYNLKERY ASWMIYTYSG LFCVTINPYK WLPVYNAEVV AAYRGKKRSE APPHIFSISD N AYQYMLTD ...文字列: MVDAEMAAFG EAAPYLRKSE KERLEAQTRP FDLKKDVFVP DDKEEFVKAT ILSREGGKVT AETEHGKTVT VKEDQVLQQN PPKFDKIED MAMLTFLHEP AVLYNLKERY ASWMIYTYSG LFCVTINPYK WLPVYNAEVV AAYRGKKRSE APPHIFSISD N AYQYMLTD RENQSILITG ESGAGKTVNT KRVIQYFAVI AAIGDRSKKE QATGKGTLED QIIQANPALE AFGNAKTVRN DN SSRFGKF IRIHFGATGK LASADIETYL LEKSRVIFQL KAERDYHIFY QILSNKKPEL LDMLLITNNP YDYAFISQGE TTV ASIDDA EELMATDNAF DVLGFTTEEK NSMYKLTGAI MHFGNMKFKL KQREEQAEPD GTEEADKSAY LMGLNSADLL KGLC HPRVK VGNEYVTKGQ NVQQVVYAKG ALAKAVYERM FNWMVTRINA TLETKQPRQY FIGVLDIAGF EIFDFNSFEQ LCINF TNEK LQQFFNHHMF VLEQEEYKKE GIEWEFIDFG MDLQACIDLI EKPMGIMSIL EEECMFPKAT DMTFKAKLFD NHLGKS SNF QKPRNIKGKP EAHFSLIHYA GTVDYNIIGW LQKNKDPLNE TVVDLYKKSS LKMLSSLFAN YAGFDTPIEK GKGKAKK GS SFQTVSALHR ENLNKLMTNL RSTHPHFVRC IIPNETKSPG VIDNPLVMHQ LRCNGVLEGI RICRKGFPNR ILYGDFRQ R YRILNPAAIP EGQFIDSRKG AEKLLGSLDI DHNQYKFGHT KVFFKAGLLG LLEEMRDERL SRIITRIQAQ SRGVLSRME FKKLLERRDS LLIIQWNIRA FMGVKNWPWM KLYFKIKPLL KSAETEKEIA UniProtKB: Myosin-7 |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 3 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 3 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | .525 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: AIR / 詳細: Glow discharged using a PELCO easiGlow station | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK III 詳細: Thin filaments were reconstituted by first mixing F-actin and tropomyosin to final concentrations of 10 uM actin and 7 uM tropomyosin. Just before applying 1.5 uL actin-tropomyosin to a ...詳細: Thin filaments were reconstituted by first mixing F-actin and tropomyosin to final concentrations of 10 uM actin and 7 uM tropomyosin. Just before applying 1.5 uL actin-tropomyosin to a freshly glow discharged holey-carbon grid, the surfactant octyl B-D-glucopyranoside was added to the protein solution to a concentration of 12 nM. The grid sample was manually blotted for 1 second and a 1.5 uL drop of 7.5 uM myosin-S1 sub-fragment was then applied to the blotted grid sample. The sample was immediately blotted for 4 seconds and plunge-frozen in liquid ethane.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-35 / 撮影したグリッド数: 1 / 実像数: 2496 / 平均露光時間: 7.0 sec. / 平均電子線量: 53.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 130000 / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 詳細 | version 1.9.3; initial flexible fitting to align secondary structure using MDFF | |||||||||
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT | |||||||||
| 得られたモデル |  PDB-6x5z: |
-原子モデル構築 2
| 詳細 | version 1.18-3861 |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 70.88 |
| 得られたモデル |  PDB-6x5z: |
-原子モデル構築 3
| 詳細 | manual refinement of residues to match backbone and sidechain density and to limit clashes, sidechain outliers, and Ramachandran outliers |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER |
| 得られたモデル |  PDB-6x5z: |
 ムービー
ムービー コントローラー
コントローラー