+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21921 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Bacillus subtilis RNA Polymerase in complex with HelD | |||||||||
 マップデータ マップデータ | RNA Polymerase in complex with HelD | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA POLYMERASE / TRANSFERASE-HELD COMPLEX / TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA helicase complex / nucleoid / recombinational repair / 3'-5' DNA helicase activity / DNA-directed RNA polymerase complex / ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity / DNA-directed RNA polymerase / DNA helicase / protein dimerization activity ...DNA helicase complex / nucleoid / recombinational repair / 3'-5' DNA helicase activity / DNA-directed RNA polymerase complex / ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity / DNA-directed RNA polymerase / DNA helicase / protein dimerization activity / response to antibiotic / DNA-templated transcription / magnesium ion binding / ATP hydrolysis activity / DNA binding / zinc ion binding / ATP binding / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
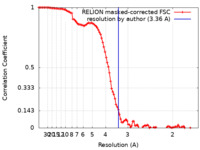
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.36 Å | |||||||||
 データ登録者 データ登録者 | Newing T / Tolun G | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Molecular basis for RNA polymerase-dependent transcription complex recycling by the helicase-like motor protein HelD. 著者: Timothy P Newing / Aaron J Oakley / Michael Miller / Catherine J Dawson / Simon H J Brown / James C Bouwer / Gökhan Tolun / Peter J Lewis /  要旨: In bacteria, transcription complexes stalled on DNA represent a major source of roadblocks for the DNA replication machinery that must be removed in order to prevent damaging collisions. Gram- ...In bacteria, transcription complexes stalled on DNA represent a major source of roadblocks for the DNA replication machinery that must be removed in order to prevent damaging collisions. Gram-positive bacteria contain a transcription factor HelD that is able to remove and recycle stalled complexes, but it was not known how it performed this function. Here, using single particle cryo-electron microscopy, we have determined the structures of Bacillus subtilis RNA polymerase (RNAP) elongation and HelD complexes, enabling analysis of the conformational changes that occur in RNAP driven by HelD interaction. HelD has a 2-armed structure which penetrates deep into the primary and secondary channels of RNA polymerase. One arm removes nucleic acids from the active site, and the other induces a large conformational change in the primary channel leading to removal and recycling of the stalled polymerase, representing a novel mechanism for recycling transcription complexes in bacteria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21921.map.gz emd_21921.map.gz | 19.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21921-v30.xml emd-21921-v30.xml emd-21921.xml emd-21921.xml | 23 KB 23 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_21921_fsc.xml emd_21921_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21921.png emd_21921.png | 141 KB | ||
| Filedesc metadata |  emd-21921.cif.gz emd-21921.cif.gz | 8.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21921 http://ftp.pdbj.org/pub/emdb/structures/EMD-21921 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21921 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21921 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21921_validation.pdf.gz emd_21921_validation.pdf.gz | 562.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21921_full_validation.pdf.gz emd_21921_full_validation.pdf.gz | 562.3 KB | 表示 | |
| XML形式データ |  emd_21921_validation.xml.gz emd_21921_validation.xml.gz | 9.8 KB | 表示 | |
| CIF形式データ |  emd_21921_validation.cif.gz emd_21921_validation.cif.gz | 13.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21921 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21921 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21921 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21921 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6wvkMC  6wvjC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-11052 (タイトル: Cryo-EM structure of Bacillus subtilis RNA Polymerase in complex with HelD EMPIAR-11052 (タイトル: Cryo-EM structure of Bacillus subtilis RNA Polymerase in complex with HelDData size: 2.8 TB Data #1: Unaligned multi-frame micrographs for Bacillus subtilis RNA Polymerase in complex with HelD [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21921.map.gz / 形式: CCP4 / 大きさ: 20.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21921.map.gz / 形式: CCP4 / 大きさ: 20.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RNA Polymerase in complex with HelD | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.84 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : DNA-directed RNA polymerase complex with helD
| 全体 | 名称: DNA-directed RNA polymerase complex with helD |
|---|---|
| 要素 |
|
-超分子 #1: DNA-directed RNA polymerase complex with helD
| 超分子 | 名称: DNA-directed RNA polymerase complex with helD / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 442.06 KDa |
-分子 #1: DNA-directed RNA polymerase subunit alpha
| 分子 | 名称: DNA-directed RNA polymerase subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 34.842387 KDa |
| 配列 | 文字列: MIEIEKPKIE TVEISDDAKF GKFVVEPLER GYGTTLGNSL RRILLSSLPG AAVTSIQIDG VLHEFSTIEG VVEDVTTIIL HIKKLALKI YSDEEKTLEI DVQGEGTVTA ADITHDSDVE ILNPDLHIAT LGENASFRVR LTAQRGRGYT PADANKRDDQ P IGVIPIDS ...文字列: MIEIEKPKIE TVEISDDAKF GKFVVEPLER GYGTTLGNSL RRILLSSLPG AAVTSIQIDG VLHEFSTIEG VVEDVTTIIL HIKKLALKI YSDEEKTLEI DVQGEGTVTA ADITHDSDVE ILNPDLHIAT LGENASFRVR LTAQRGRGYT PADANKRDDQ P IGVIPIDS IYTPVSRVSY QVENTRVGQV ANYDKLTLDV WTDGSTGPKE AIALGSKILT EHLNIFVGLT DEAQHAEIMV EK EEDQKEK VLEMTIEELD LSVRSYNCLK RAGINTVQEL ANKTEEDMMK VRNLGRKSLE EVKAKLEELG LGLRKDD UniProtKB: DNA-directed RNA polymerase subunit alpha |
-分子 #2: DNA-directed RNA polymerase subunit beta
| 分子 | 名称: DNA-directed RNA polymerase subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 133.847938 KDa |
| 配列 | 文字列: MTGQLVQYGR HRQRRSYARI SEVLELPNLI EIQTSSYQWF LDEGLREMFQ DISPIEDFTG NLSLEFIDYS LGEPKYPVEE SKERDVTYS APLRVKVRLI NKETGEVKDQ DVFMGDFPIM TDTGTFIING AERVIVSQLV RSPSVYFSGK VDKNGKKGFT A TVIPNRGA ...文字列: MTGQLVQYGR HRQRRSYARI SEVLELPNLI EIQTSSYQWF LDEGLREMFQ DISPIEDFTG NLSLEFIDYS LGEPKYPVEE SKERDVTYS APLRVKVRLI NKETGEVKDQ DVFMGDFPIM TDTGTFIING AERVIVSQLV RSPSVYFSGK VDKNGKKGFT A TVIPNRGA WLEYETDAKD VVYVRIDRTR KLPVTVLLRA LGFGSDQEIL DLIGENEYLR NTLDKDNTEN SDKALLEIYE RL RPGEPPT VENAKSLLDS RFFDPKRYDL ANVGRYKINK KLHIKNRLFN QRLAETLVDP ETGEILAEKG QILDRRTLDK VLP YLENGI GFRKLYPNGG VVEDEVTLQS IKIFAPTDQE GEQVINVIGN AYIEEEIKNI TPADIISSIS YFFNLLHGVG DTDD IDHLG NRRLRSVGEL LQNQFRIGLS RMERVVRERM SIQDTNTITP QQLINIRPVI ASIKEFFGSS QLSQFMDQTN PLAEL THKR RLSALGPGGL TRERAGMEVR DVHYSHYGRM CPIETPEGPN IGLINSLSSY AKVNRFGFIE TPYRRVDPET GKVTGR IDY LTADEEDNYV VAQANARLDD EGAFIDDSIV ARFRGENTVV SRNRVDYMDV SPKQVVSAAT ACIPFLENDD SNRALMG AN MQRQAVPLMQ PEAPFVGTGM EYVSGKDSGA AVICKHPGIV ERVEAKNVWV RRYEEVDGQK VKGNLDKYSL LKFVRSNQ G TCYNQRPIVS VGDEVVKGEI LADGPSMELG ELALGRNVMV GFMTWDGYNY EDAIIMSERL VKDDVYTSIH IEEYESEAR DTKLGPEEIT RDIPNVGEDA LRNLDDRGII RIGAEVKDGD LLVGKVTPKG VTELTAEERL LHAIFGEKAR EVRDTSLRVP HGGGGIIHD VKVFNREDGD ELPPGVNQLV RVYIVQKRKI SEGDKMAGRH GNKGVISKIL PEEDMPYLPD GTPIDIMLNP L GVPSRMNI GQVLELHMGM AARYLGIHIA SPVFDGAREE DVWETLEEAG MSRDAKTVLY DGRTGEPFDN RVSVGIMYMI KL AHMVDDK LHARSTGPYS LVTQQPLGGK AQFGGQRFGE MEVWALEAYG AAYTLQEILT VKSDDVVGRV KTYEAIVKGD NVP EPGVPE SFKVLIKELQ SLGMDVKILS GDEEEIEMRD LEDEEDAKQA DGLALSGDEE PEETASADVE RDVVTKE UniProtKB: DNA-directed RNA polymerase subunit beta |
-分子 #3: DNA-directed RNA polymerase subunit beta'
| 分子 | 名称: DNA-directed RNA polymerase subunit beta' / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 134.444953 KDa |
| 配列 | 文字列: MLDVNNFEYM NIGLASPDKI RSWSFGEVKK PETINYRTLK PEKDGLFCER IFGPTKDWEC HCGKYKRVRY KGVVCDRCGV EVTRAKVRR ERMGHIELAA PVSHIWYFKG IPSRMGLVLD MSPRALEEVI YFASYVVTDP ANTPLEKKQL LSEKEYRAYL D KYGNKFQA ...文字列: MLDVNNFEYM NIGLASPDKI RSWSFGEVKK PETINYRTLK PEKDGLFCER IFGPTKDWEC HCGKYKRVRY KGVVCDRCGV EVTRAKVRR ERMGHIELAA PVSHIWYFKG IPSRMGLVLD MSPRALEEVI YFASYVVTDP ANTPLEKKQL LSEKEYRAYL D KYGNKFQA SMGAEAIHKL LQDIDLVKEV DMLKEELKTS QGQRRTRAIK RLEVLEAFRN SGNKPSWMIL DVLPVIPPEL RP MVQLDGG RFATSDLNDL YRRVINRNNR LKRLLDLGAP SIIVQNEKRM LQEAVDALID NGRRGRPVTG PGNRPLKSLS HML KGKQGR FRQNLLGKRV DYSGRSVIVV GPHLKMYQCG LPKEMALELF KPFVMKELVE KGLAHNIKSA KRKIERVQPE VWDV LESVI KEHPVLLNRA PTLHRLGIQA FEPTLVEGRA IRLHPLVCTA YNADFDGDQM AVHVPLSAEA QAEARILMLA AQNIL NPKD GKPVVTPSQD MVLGNYYLTL ERAGAVGEGM VFKNTDEALL AYQNGYVHLH TRVAVAANSL KNVTFTEEQR SKLLIT TVG KLVFNEILPE SFPYMNEPTK SNIEEKTPDR FFLEKGADVK AVIAQQPINA PFKKGILGKI IAEIFKRFHI TETSKML DR MKNLGFKYST KAGITVGVSD IVVLDDKQEI LEEAQSKVDN VMKQFRRGLI TEEERYERVI SIWSAAKDVI QGKLMKSL D ELNPIYMMSD SGARGNASNF TQLAGMRGLM ANPAGRIIEL PIKSSFREGL TVLEYFISTH GARKGLADTA LKTADSGYL TRRLVDVAQD VIIRETDCGT DRGILAKPLK EGTETIERLE ERLIGRFARK QVKHPETGEV LVNENELIDE DKALEIVEAG IEEVWIRSA FTCNTPHGVC KRCYGRNLAT GSDVEVGEAV GIIAAQSIGE PGTQLTMRTF HTGGVAGDDI TQGLPRIQEL F EARNPKGQ ATITEIDGTV VEINEVRDKQ QEIVVQGAVE TRSYTAPYNS RLKVAEGDKI TRGQVLTEGS IDPKELLKVT DL TTVQEYL LHEVQKVYRM QGVEIGDKHV EVMVRQMLRK VRVIDAGDTD VLPGTLLDIH QFTEANKKVL LEGNRPATGR PVL LGITKA SLETDSFLSA ASFQETTRVL TDAAIKGKRD ELLGLKENVI IGKLVPAGTG MMKYRKVKPV SNVQPTDDMV PVE UniProtKB: DNA-directed RNA polymerase subunit beta' |
-分子 #4: UPF0356 protein YkzG
| 分子 | 名称: UPF0356 protein YkzG / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 8.263358 KDa |
| 配列 | 文字列: MIYKVFYQEK ADEVPVREKT DSLYIEGVSE RDVRTKLKEK KFNIEFITPV DGAFLEYEQQ SENFKVLEL UniProtKB: DNA-directed RNA polymerase subunit epsilon |
-分子 #5: DNA-directed RNA polymerase subunit omega
| 分子 | 名称: DNA-directed RNA polymerase subunit omega / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 7.766921 KDa |
| 配列 | 文字列: MLDPSIDSLM NKLDSKYTLV TVSARRAREM QIKKDQMIEH TISHKYVGKA LEEIDAGLLS FEKEDRE UniProtKB: DNA-directed RNA polymerase subunit omega |
-分子 #6: DNA helicase IV
| 分子 | 名称: DNA helicase IV / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 90.052516 KDa |
| 配列 | 文字列: MNQQDKEWKE EQSRIDEVLK ELEKKERFLE TSAGGLKHDI IGLRKSFWED VKVNFDDAHE AIETMASIKQ QAELLSDREH NHRRMDQQL KRIHQLKKSP YFGRIDFIEN GEEQAERIYI GLASCLDEKE EHFLIYDWRA PISSLYYNYS PGKAEYEVPG E TIEGEMVL ...文字列: MNQQDKEWKE EQSRIDEVLK ELEKKERFLE TSAGGLKHDI IGLRKSFWED VKVNFDDAHE AIETMASIKQ QAELLSDREH NHRRMDQQL KRIHQLKKSP YFGRIDFIEN GEEQAERIYI GLASCLDEKE EHFLIYDWRA PISSLYYNYS PGKAEYEVPG E TIEGEMVL KRQFMIKNGT LKAMFNTDMT IGDEMLQEVL SHHSDTQMKN IVSTIQKEQN QIIRNEKSKI LIVQGAAGSG KT SAALQRV AYLLYRHRGV IDAGQIVLFS PNFLFNSYVS SVLPELGEEN MEQATFQEYI EHRLGRKFKC ESPFDQLEYC LTE TKGGDF PTRLAGITWK AGLSFQQFIN EYVTRLSSEG MIFKNIIFRG QKLITKEQIQ SYFYSLDQNH SIPNRMEQTA KWLL SELNK LEKKERRKDW VVHEAELLDK EDYLDVYKKL QERKRFSEST FNDYQREQQL LAAIIVKKAF KPLKQAVRLL AFLDV TQLY LQLFSGWGGK FQHEKMDAIG ELTRSAFTDN KLLYEDAAPF LYMQDLIEGR KKNTKIKHLF IDEAQDYSPF QMAYMR SIF PAASMTVLGD INQSIYAHTI NGDQRMDACF EDEPAEYVRL KRTYRSTRQI VEFTKAMLQD GADIEPFNRS GEMPLVV KT EGHESLCQKL AQEIGRLKKK GHETIAVICK TAHQCIQAHA HMSEYTDVRL IHKENQPFQK GVCVIPVYLA KGIEFDAV L VYDASEEHYH TEHDRRLLYT ACTRAMHMLA VFYTGEASPF VTAVPPHLYQ IAE UniProtKB: DNA helicase IV |
-分子 #7: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 7 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #8: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 8 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.57 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 50 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 32 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.02 kPa | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV 詳細: Sample loading volume ranged between 2 and 3 microlitres. Samples were blotted for 5 seconds prior to vitrification.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-60 / 撮影したグリッド数: 1 / 実像数: 4331 / 平均露光時間: 9.0 sec. / 平均電子線量: 63.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 59500 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.6 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-6wvk: |
 ムービー
ムービー コントローラー
コントローラー