+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20651 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Human Metapneumovirus Polymerase bound to the phosphoprotein tetramer | ||||||||||||
 マップデータ マップデータ | None | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | human metapneumovirus / HMPV / Polymerase / Phosphoprotein / Respiratory syncytial virus / RSV / pneumoviridae / Mononegavirales / VIRAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報NNS virus cap methyltransferase / GDP polyribonucleotidyltransferase / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / virion component / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / host cell cytoplasm / RNA-directed RNA polymerase / RNA-dependent RNA polymerase activity / GTPase activity / ATP binding / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Human metapneumovirus CAN97-83 (ウイルス) / Human metapneumovirus CAN97-83 (ウイルス) /  Human metapneumovirus (strain CAN97-83) (ウイルス) Human metapneumovirus (strain CAN97-83) (ウイルス) | ||||||||||||
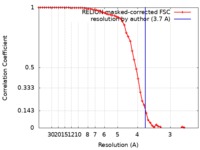
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | ||||||||||||
 データ登録者 データ登録者 | Pan J / Qian X | ||||||||||||
| 資金援助 |  シンガポール, シンガポール,  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Structure of the human metapneumovirus polymerase phosphoprotein complex. 著者: Junhua Pan / Xinlei Qian / Simon Lattmann / Abbas El Sahili / Tiong Han Yeo / Huan Jia / Tessa Cressey / Barbara Ludeke / Sarah Noton / Marian Kalocsay / Rachel Fearns / Julien Lescar /   要旨: Respiratory syncytial virus (RSV) and human metapneumovirus (HMPV) cause severe respiratory diseases in infants and elderly adults. No vaccine or effective antiviral therapy currently exists to ...Respiratory syncytial virus (RSV) and human metapneumovirus (HMPV) cause severe respiratory diseases in infants and elderly adults. No vaccine or effective antiviral therapy currently exists to control RSV or HMPV infections. During viral genome replication and transcription, the tetrameric phosphoprotein P serves as a crucial adaptor between the ribonucleoprotein template and the L protein, which has RNA-dependent RNA polymerase (RdRp), GDP polyribonucleotidyltransferase and cap-specific methyltransferase activities. How P interacts with L and mediates the association with the free form of N and with the ribonucleoprotein is not clear for HMPV or other major human pathogens, including the viruses that cause measles, Ebola and rabies. Here we report a cryo-electron microscopy reconstruction that shows the ring-shaped structure of the polymerase and capping domains of HMPV-L bound to a tetramer of P. The connector and methyltransferase domains of L are mobile with respect to the core. The putative priming loop that is important for the initiation of RNA synthesis is fully retracted, which leaves space in the active-site cavity for RNA elongation. P interacts extensively with the N-terminal region of L, burying more than 4,016 Å of the molecular surface area in the interface. Two of the four helices that form the coiled-coil tetramerization domain of P, and long C-terminal extensions projecting from these two helices, wrap around the L protein in a manner similar to tentacles. The structural versatility of the four P protomers-which are largely disordered in their free state-demonstrates an example of a 'folding-upon-partner-binding' mechanism for carrying out P adaptor functions. The structure shows that P has the potential to modulate multiple functions of L and these results should accelerate the design of specific antiviral drugs. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20651.map.gz emd_20651.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20651-v30.xml emd-20651-v30.xml emd-20651.xml emd-20651.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20651_fsc.xml emd_20651_fsc.xml | 5.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20651.png emd_20651.png | 110 KB | ||
| Filedesc metadata |  emd-20651.cif.gz emd-20651.cif.gz | 8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20651 http://ftp.pdbj.org/pub/emdb/structures/EMD-20651 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20651 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20651 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20651_validation.pdf.gz emd_20651_validation.pdf.gz | 577.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20651_full_validation.pdf.gz emd_20651_full_validation.pdf.gz | 577.1 KB | 表示 | |
| XML形式データ |  emd_20651_validation.xml.gz emd_20651_validation.xml.gz | 8.3 KB | 表示 | |
| CIF形式データ |  emd_20651_validation.cif.gz emd_20651_validation.cif.gz | 10.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20651 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20651 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20651 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20651 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20651.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20651.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
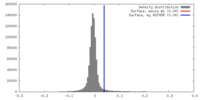
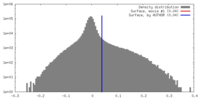
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human metapneumovirus CAN97-83
| 全体 | 名称:  Human metapneumovirus CAN97-83 (ウイルス) Human metapneumovirus CAN97-83 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human metapneumovirus CAN97-83
| 超分子 | 名称: Human metapneumovirus CAN97-83 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Human metapneumovirus L and P genes were co-expressed in sf9 insect cells using recombinant baculovirus expression system. The protein complex were affinity purified using nickel-NTA resin, ...詳細: Human metapneumovirus L and P genes were co-expressed in sf9 insect cells using recombinant baculovirus expression system. The protein complex were affinity purified using nickel-NTA resin, followed by purification on a HiTrap Heparin column and a superpose 6 size exclusion column. |
|---|---|
| 由来(天然) | 生物種:  Human metapneumovirus CAN97-83 (ウイルス) Human metapneumovirus CAN97-83 (ウイルス) |
| 分子量 | 理論値: 360 KDa |
-分子 #1: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 1 詳細: amino acid residues 1-25 is the N-terminal expression/purification tag containing a Strep II tag, a linker and a TEV protease cleavage site. コピー数: 1 / 光学異性体: LEVO / EC番号: RNA-directed RNA polymerase |
|---|---|
| 由来(天然) | 生物種:  Human metapneumovirus (strain CAN97-83) (ウイルス) Human metapneumovirus (strain CAN97-83) (ウイルス)株: CAN97-83 |
| 分子量 | 理論値: 233.889562 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGWSHPQFEK SSGVDLGTEN LYFQSMDPLN ESTVNVYLPD SYLKGVISFS ETNAIGSCLL KRPYLKNDNT AKVAIENPVI EHVRLKNAV NSKMKISDYK VVEPVNMQHE IMKNVHSCEL TLLKQFLTRS KNISTLKLNM ICDWLQLKST SDDTSILSFI D VEFIPSWV ...文字列: MGWSHPQFEK SSGVDLGTEN LYFQSMDPLN ESTVNVYLPD SYLKGVISFS ETNAIGSCLL KRPYLKNDNT AKVAIENPVI EHVRLKNAV NSKMKISDYK VVEPVNMQHE IMKNVHSCEL TLLKQFLTRS KNISTLKLNM ICDWLQLKST SDDTSILSFI D VEFIPSWV SNWFSNWYNL NKLILEFRRE EVIRTGSILC RSLGKLVFIV SSYGCIVKSN KSKRVSFFTY NQLLTWKDVM LS RFNANFC IWVSNSLNEN QEGLGLRSNL QGMLTNKLYE TVDYMLSLCC NEGFSLVKEF EGFIMSEILR ITEHAQFSTR FRN TLLNGL TDQLTKLKNK NRLRVHSTVL ENNDYPMYEV VLKLLGDTLR CIKLLINKNL ENAAELYYIF RIFGHPMVDE RDAM DAVKL NNEITKILRL ESLTELRGAF ILRIIKGFVD NNKRWPKIKN LKVLSKRWTM YFKAKNYPSQ LELSEQDFLE LAAIQ FEQE FSVPEKTNLE MVLNDKAISP PKRLIWSVYP KNYLPETIKN RYLEETFNAS DSLKTRRVLE YYLKDNKFDQ KELKSY VVR QEYLNDKEHI VSLTGKEREL SVGRMFAMQP GKQRQIQILA EKLLADNIVP FFPETLTKYG DLDLQRIMEI KSELSSI KT RRNDSYNNYI ARASIVTDLS KFNQAFRYET TAICADVADE LHGTQSLFCW LHLIVPMTTM ICAYRHAPPE TKGEYDID K IEEQSGLYRY HMGGIEGWCQ KLWTMEAISL LDVVSVKTRC QMTSLLNGDN QSIDVSKPVK LSEGLDEVKA DYRLAVKML KEIRDAYRNI GHKLKEGETY ISRDLQFISK VIQSEGVMHP TPIKKVLRVG PWINTILDDI KTSAESIGSL CQELEFRGES IIVSLILRN FWLYNLYMHE SKQHPLAGKQ LFKQLNKTLT SVQRFFEIKR ENEVVDLWMN IPMQFGGGDP VVFYRSFYRR T PDFLTEAI SHVDILLKIS ANIKNETKVS FFKALLSIEK NERATLTTLM RDPQAVGSER QAKVTSDINR TAVTSILSLS PN QLFSDSA IHYSRNEEEV GIIAENITPV YPHGLRVLYE SLPFHKAEKV VNMISGTKSI TNLLQRTSAI NGEDIDRAVS MML ENLGLL SRILSVVVDS IEIPIKSNGR LICCQISRTL RETSWNNMEI VGVTSPSITT CMDVIYATSS HLKGIIIEKF STDR TTRGQ RGPKSPWVGS STQEKKLVPV YNRQILSKQQ REQLEAIGKM RWVYKGTPGL RRLLNKICLG SLGISYKCVK PLLPR FMSV NFLHRLSVSS RPMEFPASVP AYRTTNYHFD TSPINQALSE RFGNEDINLV FQNAISCGIS IMSVVEQLTG RSPKQL VLI PQLEEIDIMP PPVFQGKFNY KLVDKITSDQ HIFSPDKIDM LTLGKMLMPT IKGQKTDQFL NKRENYFHGN NLIESLS AA LACHWCGILT EQCIENNIFK KDWGDGFISD HAFMDFKIFL CVFKTKLLCS WGSQGKNIKD EDIVDESIDK LLRIDNTF W RMFSKVMFEP KVKKRIMLYD VKFLSLVGYI GFKNWFIEQL RSAELHEIPW IVNAEGDLVE IKSIKIYLQL IEQSLFLRI TVLNYTDMAH ALTRLIRKKL MCDNALLTPI SSPMVNLTQV IDPTTQLDYF PKITFERLKN YDTSSNYAKG KLTRNYMILL PWQHVNRYN FVFSSTGCKV SLKTCIGKLM KDLNPKVLYF IGEGAGNWMA RTACEYPDIK FVYRSLKDDL DHHYPLEYQR V IGELSRII DSGEGLSMET TDATQKTHWD LIHRVSKDAL LITLCDAEFK DRDDFFKMVI LWRKHVLSCR ICTTYGTDLY LF AKYHAKD CNVKLPFFVR SVATFIMQGS KLSGSECYIL LTLGHHNSLP CHGEIQNSKM KIAVCNDFYA AKKLDNKSIE ANC KSLLSG LRIPINKKEL DRQRRLLTLQ SNHSSVATVG GSKIIESKWL TNKASTIIDW LEHILNSPKG ELNYDFFEAL ENTY PNMIK LIDNLGNAEI KKLIKVTGYM LVSKK UniProtKB: RNA-directed RNA polymerase L |
-分子 #2: Phosphoprotein
| 分子 | 名称: Phosphoprotein / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human metapneumovirus (strain CAN97-83) (ウイルス) Human metapneumovirus (strain CAN97-83) (ウイルス)株: CAN97-83 |
| 分子量 | 理論値: 35.685066 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGHHHHHHHH SSGVDLGTEN LYFQSMSFPE GKDILFMGNE AAKLAEAFQK SLRKPSHKRS QSIIGEKVNT VSETLELPTI SRPTKPTIL SEPKLAWTDK GGAIKTEAKQ TIKVMDPIEE EEFTEKRVLP SSDGKTPAEK KLKPSTNTKK KVSFTPNEPG K YTKLEKDA ...文字列: MGHHHHHHHH SSGVDLGTEN LYFQSMSFPE GKDILFMGNE AAKLAEAFQK SLRKPSHKRS QSIIGEKVNT VSETLELPTI SRPTKPTIL SEPKLAWTDK GGAIKTEAKQ TIKVMDPIEE EEFTEKRVLP SSDGKTPAEK KLKPSTNTKK KVSFTPNEPG K YTKLEKDA LDLLSDNEEE DAESSILTFE ERDTSSLSIE ARLESIEEKL SMILGLLRTL NIATAGPTAA RDGIRDAMIG IR EELIADI IKEAKGKAAE MMEEEMNQRT KIGNGSVKLT EKAKELNKIV EDESTSGESE EEEELKDTQE NNQEDDIYQL IM UniProtKB: Phosphoprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: sample of protein complex was in buffer containing 20 mM Na Hepes, pH 7.4, 300 mM NaCl, 1 mM MgCl2 and 0.5 mM TCEP (components as listed). | |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR / 詳細: 20 mA | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV 詳細: 3.5 uL sample (0.8 mg/mL)/grid, 4-second blotting (double-sided) at offset -2 before plunging.. | |||||||||||||||
| 詳細 | purified human metapneumovirus L:P complex in a 1:4 stoichiometry. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 90.0 K |
| 詳細 | preliminary grid screening was performed manually on a FEI Tecnai F20 microscope equipped with a Gatan K2 summit camera. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7676 pixel / デジタル化 - サイズ - 縦: 7420 pixel / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 1 / 実像数: 7438 / 平均露光時間: 0.2 sec. / 平均電子線量: 1.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.5 µm / 倍率(補正後): 40323 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 31000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)