+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20647 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM Structure of Pyocin R2 - postcontracted - collar | |||||||||||||||
 マップデータ マップデータ | Structure of Pyocin R2 - postcontracted - collar | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | bacteriocin / pyocin / ANTIMICROBIAL PROTEIN | |||||||||||||||
| 機能・相同性 | Tail sheath protein Gp18 domain III N-terminal region / Tail sheath protein, subtilisin-like domain / Phage tail sheath protein subtilisin-like domain / : / Tail sheath protein, C-terminal domain / Phage tail sheath C-terminal domain / Probable bacteriophage protein / Phage protein 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Pseudomonas aeruginosa PAO1 (緑膿菌) / Pseudomonas aeruginosa PAO1 (緑膿菌) /  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) | |||||||||||||||
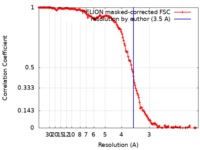
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||||||||
 データ登録者 データ登録者 | Ge P / Avaylon J | |||||||||||||||
| 資金援助 |  米国, 米国,  スイス, 4件 スイス, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Action of a minimal contractile bactericidal nanomachine. 著者: Peng Ge / Dean Scholl / Nikolai S Prokhorov / Jaycob Avaylon / Mikhail M Shneider / Christopher Browning / Sergey A Buth / Michel Plattner / Urmi Chakraborty / Ke Ding / Petr G Leiman / Jeff ...著者: Peng Ge / Dean Scholl / Nikolai S Prokhorov / Jaycob Avaylon / Mikhail M Shneider / Christopher Browning / Sergey A Buth / Michel Plattner / Urmi Chakraborty / Ke Ding / Petr G Leiman / Jeff F Miller / Z Hong Zhou /    要旨: R-type bacteriocins are minimal contractile nanomachines that hold promise as precision antibiotics. Each bactericidal complex uses a collar to bridge a hollow tube with a contractile sheath loaded ...R-type bacteriocins are minimal contractile nanomachines that hold promise as precision antibiotics. Each bactericidal complex uses a collar to bridge a hollow tube with a contractile sheath loaded in a metastable state by a baseplate scaffold. Fine-tuning of such nucleic acid-free protein machines for precision medicine calls for an atomic description of the entire complex and contraction mechanism, which is not available from baseplate structures of the (DNA-containing) T4 bacteriophage. Here we report the atomic model of the complete R2 pyocin in its pre-contraction and post-contraction states, each containing 384 subunits of 11 unique atomic models of 10 gene products. Comparison of these structures suggests the following sequence of events during pyocin contraction: tail fibres trigger lateral dissociation of baseplate triplexes; the dissociation then initiates a cascade of events leading to sheath contraction; and this contraction converts chemical energy into mechanical force to drive the iron-tipped tube across the bacterial cell surface, killing the bacterium. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20647.map.gz emd_20647.map.gz | 261.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20647-v30.xml emd-20647-v30.xml emd-20647.xml emd-20647.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20647_fsc.xml emd_20647_fsc.xml | 14.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20647.png emd_20647.png | 226.1 KB | ||
| Filedesc metadata |  emd-20647.cif.gz emd-20647.cif.gz | 6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20647 http://ftp.pdbj.org/pub/emdb/structures/EMD-20647 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20647 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20647 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20647_validation.pdf.gz emd_20647_validation.pdf.gz | 749.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20647_full_validation.pdf.gz emd_20647_full_validation.pdf.gz | 748.8 KB | 表示 | |
| XML形式データ |  emd_20647_validation.xml.gz emd_20647_validation.xml.gz | 14.5 KB | 表示 | |
| CIF形式データ |  emd_20647_validation.cif.gz emd_20647_validation.cif.gz | 19.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20647 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20647 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20647 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20647 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20647.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20647.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Pyocin R2 - postcontracted - collar | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.041 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Pyocin R2 - Postcontracted
| 全体 | 名称: Pyocin R2 - Postcontracted |
|---|---|
| 要素 |
|
-超分子 #1: Pyocin R2 - Postcontracted
| 超分子 | 名称: Pyocin R2 - Postcontracted / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
-分子 #1: Collar PA0615
| 分子 | 名称: Collar PA0615 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 18.965461 KDa |
| 配列 | 文字列: MPEQAVTLEA LYAAIEQVLR ERLPEAQLIG FWPGVPENTP AVSLEIAELL PERDPGTGES ALLCRLQARI MVPPGADRQA VSIACGIVR TLREQTWNLS LQPARFVRSA VDGSREELKS LRVWLVEWTQ SLRLGDPEWA WEDQPPGSLM LGFDPQTGPG H EPDYFAPE ALA UniProtKB: Phage protein |
-分子 #2: Sheath PA0622
| 分子 | 名称: Sheath PA0622 / タイプ: protein_or_peptide / ID: 2 / コピー数: 18 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 41.247332 KDa |
| 配列 | 文字列: MSFFHGVTVT NVDIGARTIA LPASSVIGLC DVFTPGAQAS AKPNVPVLLT SKKDAAAAFG IGSSIYLACE AIYNRAQAVI VAVGVETAE TPEAQASAVI GGISAAGERT GLQALLDGKS RFNAQPRLLV APGHSAQQAV ATAMDGLAEK LRAIAILDGP N STDEAAVA ...文字列: MSFFHGVTVT NVDIGARTIA LPASSVIGLC DVFTPGAQAS AKPNVPVLLT SKKDAAAAFG IGSSIYLACE AIYNRAQAVI VAVGVETAE TPEAQASAVI GGISAAGERT GLQALLDGKS RFNAQPRLLV APGHSAQQAV ATAMDGLAEK LRAIAILDGP N STDEAAVA YAKNFGSKRL FMVDPGVQVW DSATNAARNA PASAYAAGLF AWTDAEYGFW SSPSNKEIKG VTGTSRPVEF LD GDETCRA NLLNNANIAT IIRDDGYRLW GNRTLSSDSK WAFVTRVRTM DLVMDAILAG HKWAVDRGIT KTYVKDVTEG LRA FMRDLK NQGAVINFEV YADPDLNSAS QLAQGKVYWN IRFTDVPPAE NPNFRVEVTD QWLTEVLDVA UniProtKB: Probable bacteriophage protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 構成要素:
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 詳細: unspecified | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 81.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS エネルギーフィルター - エネルギー下限: -10 eV エネルギーフィルター - エネルギー上限: 10 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 3-20 / 撮影したグリッド数: 1 / 実像数: 7331 / 平均露光時間: 10.0 sec. / 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 最大 デフォーカス(補正後): 3.4 µm / 最小 デフォーカス(補正後): 1.1 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.16 µm / 最小 デフォーカス(公称値): 2.16 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-6u5j: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)