+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20200 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the yeast Ctf3 complex | |||||||||
 マップデータ マップデータ | Final volume | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | kinetochore / chromosome / cryo-em / CELL CYCLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報attachment of spindle microtubules to kinetochore / establishment of mitotic sister chromatid cohesion / mitotic spindle assembly checkpoint signaling / DNA replication initiation / meiotic cell cycle / chromosome segregation / kinetochore / cell division / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
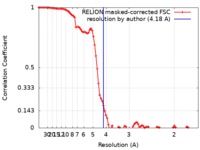
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.18 Å | |||||||||
 データ登録者 データ登録者 | Hinshaw SM / Harrison SC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2019 ジャーナル: Elife / 年: 2019タイトル: The structure of the yeast Ctf3 complex. 著者: Stephen M Hinshaw / Andrew N Dates / Stephen C Harrison /  要旨: Kinetochores are the chromosomal attachment points for spindle microtubules. They are also signaling hubs that control major cell cycle transitions and coordinate chromosome folding. Most well- ...Kinetochores are the chromosomal attachment points for spindle microtubules. They are also signaling hubs that control major cell cycle transitions and coordinate chromosome folding. Most well-studied eukaryotes rely on a conserved set of factors, which are divided among two loosely-defined groups, for these functions. Outer kinetochore proteins contact microtubules or regulate this contact directly. Inner kinetochore proteins designate the kinetochore assembly site by recognizing a specialized nucleosome containing the H3 variant Cse4/CENP-A. We previously determined the structure, resolved by cryo-electron microscopy (cryo-EM), of the yeast Ctf19 complex (Ctf19c, homologous to the vertebrate CCAN), providing a high-resolution view of inner kinetochore architecture (Hinshaw and Harrison, 2019). We now extend these observations by reporting a near-atomic model of the Ctf3 complex, the outermost Ctf19c sub-assembly seen in our original cryo-EM density. The model is sufficiently well-determined by the new data to enable molecular interpretation of Ctf3 recruitment and function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20200.map.gz emd_20200.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20200-v30.xml emd-20200-v30.xml emd-20200.xml emd-20200.xml | 13 KB 13 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20200_fsc.xml emd_20200_fsc.xml | 9.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20200.png emd_20200.png | 102.7 KB | ||
| マスクデータ |  emd_20200_msk_1.map emd_20200_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-20200.cif.gz emd-20200.cif.gz | 5.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20200 http://ftp.pdbj.org/pub/emdb/structures/EMD-20200 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20200 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20200 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20200_validation.pdf.gz emd_20200_validation.pdf.gz | 553.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20200_full_validation.pdf.gz emd_20200_full_validation.pdf.gz | 552.8 KB | 表示 | |
| XML形式データ |  emd_20200_validation.xml.gz emd_20200_validation.xml.gz | 10.8 KB | 表示 | |
| CIF形式データ |  emd_20200_validation.cif.gz emd_20200_validation.cif.gz | 14.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20200 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20200 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20200 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20200 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20200.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20200.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final volume | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_20200_msk_1.map emd_20200_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ctf3 complex
| 全体 | 名称: Ctf3 complex |
|---|---|
| 要素 |
|
-超分子 #1: Ctf3 complex
| 超分子 | 名称: Ctf3 complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Inner kinetochore subunit CTF3
| 分子 | 名称: Inner kinetochore subunit CTF3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 84.617891 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAMSLILDD IILSLTNANE RTPPQALKTT LSLLYEKSKQ YGLSSPQLQA LVRLLCETSI IDTVTKVYIV ENCFLPDGYL TKELLLEII NHLGTPTVFS RYRIQTPPVL QSALCKWLVH VYFLFPVHSE REHNISSSIW LHLWQFSFLQ KWITPLVIWQ A TTPVDVKP ...文字列: SNAMSLILDD IILSLTNANE RTPPQALKTT LSLLYEKSKQ YGLSSPQLQA LVRLLCETSI IDTVTKVYIV ENCFLPDGYL TKELLLEII NHLGTPTVFS RYRIQTPPVL QSALCKWLVH VYFLFPVHSE REHNISSSIW LHLWQFSFLQ KWITPLVIWQ A TTPVDVKP WKLSIIKRCA MHPGYRDAPG SATLILQRFQ CLVGASSQIT ESIITINCNR KTLKSHRNLK LDAHFLSILK RI LSRAHPA NFPADTVQNT IDMYLSEIHQ LGADSIYPLR LQSLPEYVPS DSTVSLWDVT SLEQLAQNWP QLHIPNDVDY MMK PSLNSN VLLPRKVMSR DSLKHLYSSI ILIKNSRDES SSPYEWCIWQ LKRCFAHQIE TPQEVIPIII SVSSMDNKLS SRII QTFCN LKYLKLDELT LKKVCGGILP LWKPELISGT REFFVKFMAS IFMWSTRDGH DNNCTFSETC FYVLQMITNW VLDDK LIAL GLTLLHDMQS LLTLDKIFNN ATSNRFSTMA FISSLDILTQ LSKQTKSDYA IQYLIVGPDI MNKVFSSDDP LLLSAA CRY LVATKNKLMQ YPSTNKFVRM QNQYIMDLTN YLYRNKVLSS KSLFGVSPDF FKQILENLYI PTADFKNAKF FTITGIP AL SYICIIILRR LETAENTKIK FTSGIINEET FNNFFRVHHD EIGQHGWIKG VNNIHDLRVK ILMHLSNTAN PYRDIAAF L FTYLKSLSKY SVQNS UniProtKB: Inner kinetochore subunit CTF3 |
-分子 #2: Inner kinetochore subunit MCM16
| 分子 | 名称: Inner kinetochore subunit MCM16 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 21.438359 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAMTNSSEK QWERIQQLEK EHVEVYRELL ITLDRLYLIR KHNHAVILSH TQQRLLEIRH QLQINLEKTA LLIRLLEKPD NTNVLFTKL QNLLEESNSL DYELLQSLGA QSSLHKQLIE SRAERDELMS KLIELSSKFP KPTIPPDDSD TAGKQVEVEK E NETIQELM IALQIHSGYT NISYTI UniProtKB: Inner kinetochore subunit MCM16 |
-分子 #3: Inner kinetochore subunit MCM22
| 分子 | 名称: Inner kinetochore subunit MCM22 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 27.874799 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SNAMDVEKDV LDVYIKNLEN QIGNKRYFLK QAQGAIDEIT KRSLDTEGKP VNSEVFTELL RKPMFFSERA DPIGFSLTSN FLSLRAQSS SEWLSLMNDQ SVDQKAMLLL QNNINSDLKE LLRKLQHQMT IMDSKKQDHA HIRTRKARNK ELWDSLADFL K GYLVPNLD ...文字列: SNAMDVEKDV LDVYIKNLEN QIGNKRYFLK QAQGAIDEIT KRSLDTEGKP VNSEVFTELL RKPMFFSERA DPIGFSLTSN FLSLRAQSS SEWLSLMNDQ SVDQKAMLLL QNNINSDLKE LLRKLQHQMT IMDSKKQDHA HIRTRKARNK ELWDSLADFL K GYLVPNLD DNDESIDSLT NEVMLLMKRL IEHDLNLTLN DFSSKTIPIY RLLLRANIIT VIEGSTNPGT KYIKLIDFNE TS LT UniProtKB: Inner kinetochore subunit MCM22 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8.5 |
|---|---|
| グリッド | モデル: C-flat-2/2 / 材質: COPPER |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | Recombinant protein frozen in vitreous ice |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均電子線量: 50.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-6oua: |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X