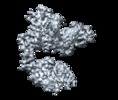
登録情報 データベース : EMDB / ID : EMD-20055タイトル Cryo-EM structure of Her2 extracellular domain-Trastuzumab Fab-Pertuzumab Fab complex Sharpened map with phenix.auto_sharpen 複合体 : Her2 extracellular domain-Trastuzumab Fab-Pertuzumab Fab complex複合体 : Human HER2 extracellular domainタンパク質・ペプチド : Receptor tyrosine-protein kinase erbB-2複合体 : Pertuzumab Fabタンパク質・ペプチド : Pertuzumab FAB LIGHT CHAINタンパク質・ペプチド : Pertuzumab FAB HEAVY CHAIN複合体 : Trastuzumab Fabタンパク質・ペプチド : Trastuzumab FAB LIGHT CHAINタンパク質・ペプチド : Trastuzumab FAB HEAVY CHAINリガンド : 2-acetamido-2-deoxy-beta-D-glucopyranose / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 4.36 Å Hao Y / Yu X ジャーナル : PLoS One / 年 : 2019タイトル : Cryo-EM Structure of HER2-trastuzumab-pertuzumab complex.著者 : Yue Hao / Xinchao Yu / Yonghong Bai / Helen J McBride / Xin Huang / 要旨 : Trastuzumab and pertuzumab are monoclonal antibodies that bind to distinct subdomains of the extracellular domain of human epidermal growth factor receptor 2 (HER2). Adding these monoclonal ... Trastuzumab and pertuzumab are monoclonal antibodies that bind to distinct subdomains of the extracellular domain of human epidermal growth factor receptor 2 (HER2). Adding these monoclonal antibodies to the treatment regimen of HER2-positive breast cancer has changed the paradigm for treatment in that form of cancer. Synergistic activity has been observed with the combination of these two antibodies leading to hypotheses regarding the mechanism(s) and to the development of bispecific antibodies to maximize the clinical effect further. Although the individual crystal structures of HER2-trastuzumab and HER2-pertuzumab revealed the distinct binding sites and provided the structural basis for their anti-tumor activities, detailed structural information on the HER2-trastuzumab-pertuzumab complex has been elusive. Here we present the cryo-EM structure of HER2-trastuzumab-pertuzumab at 4.36 Å resolution. Comparison with the binary complexes reveals no cooperative interaction between trastuzumab and pertuzumab, and provides key insights into the design of novel, high-avidity bispecific molecules with potentially greater clinical efficacy. 履歴 登録 2019年4月2日 - ヘッダ(付随情報) 公開 2019年5月1日 - マップ公開 2019年5月15日 - 更新 2024年11月20日 - 現状 2024年11月20日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: PLoS One / 年: 2019
ジャーナル: PLoS One / 年: 2019
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_20055.map.gz
emd_20055.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-20055-v30.xml
emd-20055-v30.xml emd-20055.xml
emd-20055.xml EMDBヘッダ
EMDBヘッダ emd_20055.png
emd_20055.png emd-20055.cif.gz
emd-20055.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-20055
http://ftp.pdbj.org/pub/emdb/structures/EMD-20055 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20055
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20055 emd_20055_validation.pdf.gz
emd_20055_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_20055_full_validation.pdf.gz
emd_20055_full_validation.pdf.gz emd_20055_validation.xml.gz
emd_20055_validation.xml.gz emd_20055_validation.cif.gz
emd_20055_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20055
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20055 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20055
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20055 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_20055.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_20055.map.gz / 形式: CCP4 / 大きさ: 34.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー







































 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)