+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the mu opioid receptor bound to the antagonist nanobody NbE | ||||||||||||
 マップデータ マップデータ | DeepEMhancer sharpened map of the MOR_NbE_NabFab_anti-Fab complex. | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | opioid receptor / nanobody / antagonist / NabFab / MOR / MEMBRANE PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Opioid Signalling / spine apparatus / beta-endorphin receptor activity / morphine receptor activity / negative regulation of Wnt protein secretion / Peptide ligand-binding receptors / adenylate cyclase-inhibiting opioid receptor signaling pathway / positive regulation of appetite / G-protein activation / G protein-coupled opioid receptor activity ...Opioid Signalling / spine apparatus / beta-endorphin receptor activity / morphine receptor activity / negative regulation of Wnt protein secretion / Peptide ligand-binding receptors / adenylate cyclase-inhibiting opioid receptor signaling pathway / positive regulation of appetite / G-protein activation / G protein-coupled opioid receptor activity / negative regulation of luteinizing hormone secretion / G protein-coupled opioid receptor signaling pathway / sperm ejaculation / G alpha (i) signalling events / adenylate cyclase-inhibiting G protein-coupled acetylcholine receptor signaling pathway / negative regulation of nitric oxide biosynthetic process / negative regulation of adenylate cyclase-activating G protein-coupled receptor signaling pathway / neuropeptide binding / negative regulation of cAMP-mediated signaling / regulation of NMDA receptor activity / positive regulation of neurogenesis / eating behavior / negative regulation of cytosolic calcium ion concentration / transmission of nerve impulse / social behavior / G-protein alpha-subunit binding / voltage-gated calcium channel activity / GABA-ergic synapse / neuropeptide signaling pathway / positive regulation of gluconeogenesis / sensory perception of pain / dendrite membrane / presynaptic modulation of chemical synaptic transmission / adenylate cyclase-inhibiting G protein-coupled receptor signaling pathway / dendrite cytoplasm / excitatory postsynaptic potential / locomotory behavior / G protein-coupled receptor activity / adenylate cyclase-activating dopamine receptor signaling pathway / G-protein beta-subunit binding / presynaptic membrane / phospholipase C-activating G protein-coupled receptor signaling pathway / postsynaptic membrane / perikaryon / response to ethanol / positive regulation of ERK1 and ERK2 cascade / endosome / neuron projection / G protein-coupled receptor signaling pathway / protein domain specific binding / axon / focal adhesion / dendrite / membrane / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | ||||||||||||
 データ登録者 データ登録者 | Yu J / Kumar A / Zhang X / Martin C / Raia P / Manglik A / Ballet S / Boland A / Stoeber M | ||||||||||||
| 資金援助 |  スイス, 3件 スイス, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: bioRxiv / 年: 2023 ジャーナル: bioRxiv / 年: 2023タイトル: Structural Basis of μ-Opioid Receptor-Targeting by a Nanobody Antagonist. 著者: Jun Yu / Amit Kumar / Xuefeng Zhang / Charlotte Martin / Pierre Raia / Antoine Koehl / Toon Laeremans / Jan Steyaert / Aashish Manglik / Steven Ballet / Andreas Boland / Miriam Stoeber /    要旨: The μ-opioid receptor (μOR), a prototypical member of the G protein-coupled receptor (GPCR) family, is the molecular target of opioid analgesics such as morphine and fentanyl. Due to the ...The μ-opioid receptor (μOR), a prototypical member of the G protein-coupled receptor (GPCR) family, is the molecular target of opioid analgesics such as morphine and fentanyl. Due to the limitations and severe side effects of currently available opioid drugs, there is considerable interest in developing novel modulators of μOR function. Most GPCR ligands today are small molecules, however biologics, including antibodies and nanobodies, are emerging as alternative therapeutics with clear advantages such as affinity and target selectivity. Here, we describe the nanobody NbE, which selectively binds to the μOR and acts as an antagonist. We functionally characterize NbE as an extracellular and genetically encoded μOR ligand and uncover the molecular basis for μOR antagonism by solving the cryo-EM structure of the NbE-μOR complex. NbE displays a unique ligand binding mode and achieves μOR selectivity by interactions with the orthosteric pocket and extracellular receptor loops. Based on a β-hairpin loop formed by NbE that deeply inserts into the μOR and centers most binding contacts, we design short peptide analogues that retain μOR antagonism. The work illustrates the potential of nanobodies to uniquely engage with GPCRs and describes novel μOR ligands that can serve as a basis for therapeutic developments. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18541.map.gz emd_18541.map.gz | 108.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18541-v30.xml emd-18541-v30.xml emd-18541.xml emd-18541.xml | 36.6 KB 36.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18541.png emd_18541.png | 29.5 KB | ||
| マスクデータ |  emd_18541_msk_1.map emd_18541_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18541.cif.gz emd-18541.cif.gz | 7.6 KB | ||
| その他 |  emd_18541_additional_1.map.gz emd_18541_additional_1.map.gz emd_18541_additional_2.map.gz emd_18541_additional_2.map.gz emd_18541_additional_3.map.gz emd_18541_additional_3.map.gz emd_18541_additional_4.map.gz emd_18541_additional_4.map.gz emd_18541_additional_5.map.gz emd_18541_additional_5.map.gz emd_18541_half_map_1.map.gz emd_18541_half_map_1.map.gz emd_18541_half_map_2.map.gz emd_18541_half_map_2.map.gz | 62.8 MB 62.9 MB 109.4 MB 115.9 MB 115.9 MB 116.1 MB 116.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18541 http://ftp.pdbj.org/pub/emdb/structures/EMD-18541 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18541 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18541 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18541_validation.pdf.gz emd_18541_validation.pdf.gz | 685.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18541_full_validation.pdf.gz emd_18541_full_validation.pdf.gz | 685.4 KB | 表示 | |
| XML形式データ |  emd_18541_validation.xml.gz emd_18541_validation.xml.gz | 13.7 KB | 表示 | |
| CIF形式データ |  emd_18541_validation.cif.gz emd_18541_validation.cif.gz | 16.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18541 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18541 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18541 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18541 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qotMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18541.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18541.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer sharpened map of the MOR_NbE_NabFab_anti-Fab complex. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.9759 Å | ||||||||||||||||||||
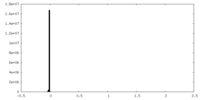
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18541_msk_1.map emd_18541_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
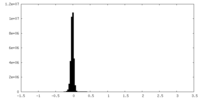
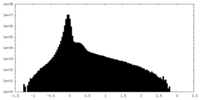
| 投影像・断面図 |
| ||||||||||||
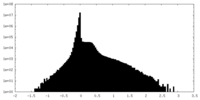
| 密度ヒストグラム |
-追加マップ: Unsharpened map of the MOR NbE NabFab anti-Fab complex.
| ファイル | emd_18541_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map of the MOR_NbE_NabFab_anti-Fab complex. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
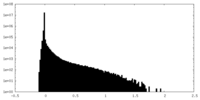
| 密度ヒストグラム |
-追加マップ: Unsharpened MOR NbE map (focused refinement).
| ファイル | emd_18541_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened MOR_NbE map (focused refinement). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
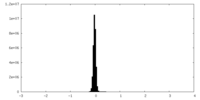
| 密度ヒストグラム |
-追加マップ: DeepEMhancer sharpened MOR NbE map (focused refinement).
| ファイル | emd_18541_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer sharpened MOR_NbE map (focused refinement). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Half map A of the MOR NbE map (focused refinement).
| ファイル | emd_18541_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A of the MOR_NbE map (focused refinement). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Half map B of the MOR NbE map (focused refinement).
| ファイル | emd_18541_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B of the MOR_NbE map (focused refinement). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A of the MOR NbE NabFab anti-Fab complex.
| ファイル | emd_18541_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A of the MOR_NbE_NabFab_anti-Fab complex. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B of the MOR NbE NabFab anti-Fab complex.
| ファイル | emd_18541_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B of the MOR_NbE_NabFab_anti-Fab complex. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MOR-NbE-NabFab-AntiFab complex
| 全体 | 名称: MOR-NbE-NabFab-AntiFab complex |
|---|---|
| 要素 |
|
-超分子 #1: MOR-NbE-NabFab-AntiFab complex
| 超分子 | 名称: MOR-NbE-NabFab-AntiFab complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Mu-opioid receptor bound to a nanobody antagonist (NbE), and NabFab module used as fiducial marker. |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 120 KDa |
-分子 #1: Mu-type opioid receptor
| 分子 | 名称: Mu-type opioid receptor / タイプ: protein_or_peptide / ID: 1 / 詳細: MOR-2xSTREP-8xHIS / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 53.994215 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKTIIALSYI FCLVFADYKD DDDAMGPGNI SDCSDPLAPA SCSPAPGSWL NLSHVDGNQS DPCGPNRTGL GENLYFQGSH SLCPQTGSP SMVTAITIMA LYSIVCVVGL FGNFLVMYVI VRYTKMKTAT NIYIFNLALA DALATSTLPF QSVNYLMGTW P FGNILCKI ...文字列: MKTIIALSYI FCLVFADYKD DDDAMGPGNI SDCSDPLAPA SCSPAPGSWL NLSHVDGNQS DPCGPNRTGL GENLYFQGSH SLCPQTGSP SMVTAITIMA LYSIVCVVGL FGNFLVMYVI VRYTKMKTAT NIYIFNLALA DALATSTLPF QSVNYLMGTW P FGNILCKI VISIDYYNMF TSIFTLCTMS VDRYIAVCHP VKALDFRTPR NAKIVNVCNW ILSSAIGLPV MFMATTKYRQ GS IDCTLTF SHPTWYWENL LKICVFIFAF IMPVLIITVC YGLMILRLKS VRMLSGSKEK DRNLRRITRM VLVVVAVFIV CWT PIHIYV IIKALITIPE TTFQTVSWHF CIALGYTNSC LNPVLYAFLD ENFKRCFREF CIPTSSTILE VLFQGPEQQN SARI RQNTR EHPSTANTVD RTNHQLENLE AETAPLPSSL AEENLYFQSW SHPQFEKGGG SGGGSGGGSW SHPQFEKGAH HHHHH HH UniProtKB: Mu-type opioid receptor |
-分子 #2: Nanobody E (NbE)
| 分子 | 名称: Nanobody E (NbE) / タイプ: protein_or_peptide / ID: 2 / 詳細: synthesized nanobody NbE-6xHis / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 18.458662 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: VKKLLFAIPL VVPFYAAQPA MAQVQLVESG GGLVQAGGSL RLSCAASGSI SSISTMGWYR QAPGKERELV AAITSGGSTN YADSVKGRF TISRDNAKNT VYLQMNSLKP EDTAVYYCNF KYYSGSYFYK SEYDYWGKGT PVTVSSAAAH HHHHHGAAEQ K LISEEDLN GAA |
-分子 #3: NabFab HC
| 分子 | 名称: NabFab HC / タイプ: protein_or_peptide / ID: 3 / 詳細: Signal sequence-NabFab HC / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 28.220525 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKKNIAFLLA SMFVFSIATN AYAEISEVQL VESGGGLVQP GGSLRLSCAA SGFNFSYYSI HWVRQAPGKG LEWVAYISSS SSYTSYADS VKGRFTISAD TSKNTAYLQM NSLRAEDTAV YYCARGYQYW QYHASWYWNG GLDYWGQGTL VTVSSASTKG P SVFPLAPS ...文字列: MKKNIAFLLA SMFVFSIATN AYAEISEVQL VESGGGLVQP GGSLRLSCAA SGFNFSYYSI HWVRQAPGKG LEWVAYISSS SSYTSYADS VKGRFTISAD TSKNTAYLQM NSLRAEDTAV YYCARGYQYW QYHASWYWNG GLDYWGQGTL VTVSSASTKG P SVFPLAPS SKSTSGGTAA LGCLVKDYFP EPVTVSWNSG ALTSGVHTFP AVLQSSGLYS LSSVVTVPSS SLGTQTYICN VN HKPSNTK VDKKVEPKSC DKTHT |
-分子 #4: NabFab LC
| 分子 | 名称: NabFab LC / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 25.794859 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKKNIAFLLA SMFVFSIATN AYASDIQMTQ SPSSLSASVG DRVTITCRAS QSVSSAVAWY QQKPGKAPKL LIYSASSLYS GVPSRFSGS RSGTDFTLTI SSLQPEDFAT YYCQQSSSSL ITFGQGTKVE IKRTVAAPSV FIFPPSDSQL KSGTASVVCL L NNFYPREA ...文字列: MKKNIAFLLA SMFVFSIATN AYASDIQMTQ SPSSLSASVG DRVTITCRAS QSVSSAVAWY QQKPGKAPKL LIYSASSLYS GVPSRFSGS RSGTDFTLTI SSLQPEDFAT YYCQQSSSSL ITFGQGTKVE IKRTVAAPSV FIFPPSDSQL KSGTASVVCL L NNFYPREA KVQWKVDNAL QSGNSQESVT EQDSKDSTYS LSSTLTLSKA DYEKHKVYAC EVTHQGLSSP VTKSFNRGEC |
-分子 #5: Anti-Fab Nanobody
| 分子 | 名称: Anti-Fab Nanobody / タイプ: protein_or_peptide / ID: 5 / 詳細: Signal sequence-6xHis Anti-Fab nanobody / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 17.414383 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKYLLPTAAA GLLLLAAQPA MAMHHHHHHG ENLYFQGSQV QLQESGGGLV QPGGSLRLSC AASGRTISRY AMSWFRQAPG KEREFVAVA RRSGDGAFYA DSVQGRFTVS RDDAKNTVYL QMNSLKPEDT AVYYCAIDSD TFYSGSYDYW GQGTQVTVSS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.8 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 288 K / 装置: LEICA EM GP | |||||||||||||||
| 詳細 | MOR receptor was expressed and purified in insect cells in isolation. NbE was expressed and purified in E. coli. NabFab module was expressed and purified in E. coli. All purified components were incubated and formed readily a stable complex. Final complex was purified over size exclusion chromatography. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 温度 | 最低: 90.0 K / 最高: 95.0 K |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 4 / 実像数: 5938 / 平均露光時間: 52.0 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 150000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 68 | ||||||||||
| 得られたモデル |  PDB-8qot: |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X