+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Asymmetric structure of the Borrelia bacteriophage BB1 procapsid, 3D class 2 | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Portal / capsid / procapsid / scaffold / VIRAL PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Protein of unknown function DUF228 / Lyme disease proteins of unknown function / Uncharacterised conserved protein UCP020357 / Protein of unknown function DUF1073 / Anti-CBASS protein Acb1-like / Protein of unknown function DUF1357 / Protein of unknown function (DUF1357) 類似検索 - ドメイン・相同性 | ||||||||||||
| 生物種 |  Borreliella burgdorferi B31 (バクテリア) Borreliella burgdorferi B31 (バクテリア) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 10.62 Å | ||||||||||||
 データ登録者 データ登録者 | Rumnieks J / Fuzik T / Tars K | ||||||||||||
| 資金援助 | European Union, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2023 ジャーナル: J Mol Biol / 年: 2023タイトル: Structure of the Borrelia Bacteriophage φBB1 Procapsid. 著者: Jānis Rūmnieks / Tibor Füzik / Kaspars Tārs /   要旨: Bacteriophages of Borrelia burgdorferi are a biologically important but under-investigated feature of the Lyme disease-causing spirochete. No virulent borrelial viruses have been identified, but all ...Bacteriophages of Borrelia burgdorferi are a biologically important but under-investigated feature of the Lyme disease-causing spirochete. No virulent borrelial viruses have been identified, but all B. burgdorferi isolates carry a prophage φBB1 as resident circular plasmids. Like its host, the φBB1 phage is quite distinctive and shares little sequence similarity with other known bacteriophages. We expressed φBB1 head morphogenesis proteins in Escherichia coli which resulted in assembly of homogeneous prolate procapsid structures and used cryo-electron microscopy to determine the three-dimensional structure of these particles. The φBB1 procapsids consist of 415 copies of the major capsid protein and an equal combined number of three homologous capsid decoration proteins that form trimeric knobs on the outside of the particle. One of the end vertices of the particle is occupied by a portal assembled from twelve copies of the portal protein. The φBB1 scaffolding protein is entirely α-helical and has an elongated shape with a small globular domain in the middle. Within the tubular section of the procapsid, the internal scaffold is built of stacked rings, each composed of 32 scaffolding protein molecules, which run in opposite directions from both caps with a heterogeneous part in the middle. Inside the portal-containing cap, the scaffold is organized asymmetrically with ten scaffolding protein molecules bound to the portal. The φBB1 procapsid structure provides better insight into the vast structural diversity of bacteriophages and presents clues of how elongated bacteriophage particles might be assembled. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18518.map.gz emd_18518.map.gz | 122.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18518-v30.xml emd-18518-v30.xml emd-18518.xml emd-18518.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18518_fsc.xml emd_18518_fsc.xml | 25.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18518.png emd_18518.png | 136.1 KB | ||
| マスクデータ |  emd_18518_msk_1.map emd_18518_msk_1.map | 1.4 GB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18518.cif.gz emd-18518.cif.gz | 7.2 KB | ||
| その他 |  emd_18518_half_map_1.map.gz emd_18518_half_map_1.map.gz emd_18518_half_map_2.map.gz emd_18518_half_map_2.map.gz | 1.1 GB 1.1 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18518 http://ftp.pdbj.org/pub/emdb/structures/EMD-18518 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18518 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18518 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qo0MC  8phoC  8phpC  8phqC  8phrC  8phsC  8phtC  8phuC  8pkhC  8qo1C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18518.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18518.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.6672 Å | ||||||||||||||||||||||||||||||||||||
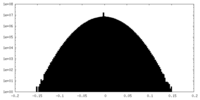
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18518_msk_1.map emd_18518_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
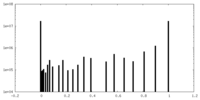
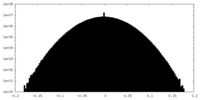
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18518_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
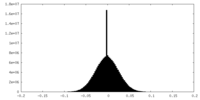
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18518_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
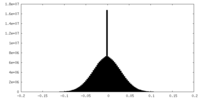
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Procapsid of Borrelia bacteriophage BB1
| 全体 | 名称: Procapsid of Borrelia bacteriophage BB1 |
|---|---|
| 要素 |
|
-超分子 #1: Procapsid of Borrelia bacteriophage BB1
| 超分子 | 名称: Procapsid of Borrelia bacteriophage BB1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Borreliella burgdorferi B31 (バクテリア) Borreliella burgdorferi B31 (バクテリア) |
-分子 #1: Cytosolic protein
| 分子 | 名称: Cytosolic protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 415 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Borreliella burgdorferi B31 (バクテリア) Borreliella burgdorferi B31 (バクテリア) |
| 分子量 | 理論値: 36.255551 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MELFDENYYA KAVANIIGEV KDPIMYKWFS PDQIEDVDLQ MGYQKTVKWD AFLNANPTTI ANEVNTISTI GFSSEVVRLN YLKLQYKFR HLKQTSEKFY TSDSYIGDIN NNLLPFAQAY KLASSEIIKL INHFVLTGTV SIQKDGKNQK RLLPNMYGLL N MPEQIKEE ...文字列: MELFDENYYA KAVANIIGEV KDPIMYKWFS PDQIEDVDLQ MGYQKTVKWD AFLNANPTTI ANEVNTISTI GFSSEVVRLN YLKLQYKFR HLKQTSEKFY TSDSYIGDIN NNLLPFAQAY KLASSEIIKL INHFVLTGTV SIQKDGKNQK RLLPNMYGLL N MPEQIKEE VASGDKDKMD KIFEKIEAGL SKLELGDEFS TPMMVIVDPA TSLKLVKPYA AAQGAASSCE KWEDVLIQTI KA INNREDV YIETSNLLKH KILIYPLNSE LIKFKPSKYM LPTPNEQVDK DSTDVAHSYI DFVLGGLLAT RKTILQVNIK QS UniProtKB: Cytosolic protein |
-分子 #2: Decoration protein P03
| 分子 | 名称: Decoration protein P03 / タイプ: protein_or_peptide / ID: 2 / コピー数: 280 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Borreliella burgdorferi B31 (バクテリア) Borreliella burgdorferi B31 (バクテリア) |
| 分子量 | 理論値: 20.026479 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSDITKIKQE FDKKVAEIQA LMKNPQQDSG LLSNSIDFRD QNLIFSNSGG VCTSSKDKIE NYPAKGYPYK RGVKLSFGDG TTELEVEAG GGDDLYGVCS DIDEFSGMAT VIPITNNFTG YLTLKKDGQN GVNPGDKLNF NQHGELEKVT GAQKSVNAIA L SKAHKLTE DLFIVLASVF GNRAIKG UniProtKB: Uncharacterized protein |
-分子 #3: Decoration protein P04
| 分子 | 名称: Decoration protein P04 / タイプ: protein_or_peptide / ID: 3 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Borreliella burgdorferi B31 (バクテリア) Borreliella burgdorferi B31 (バクテリア) |
| 分子量 | 理論値: 28.067344 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MALKGNMQVE NLEAVEDPQV DLGAQVSAAP RAKRQARQAE DVQGEDPYLE SISELDDVLL KFKKYSKSMS SIENKVFSSS SGCFKSKNE RVDAYSFACS SYTDKIEEYL YDPANSFPYK RGVKLVPKEN SIYVEVGADT DMYGICVDVC EFSCTAYVLP I TNNFEGYL ...文字列: MALKGNMQVE NLEAVEDPQV DLGAQVSAAP RAKRQARQAE DVQGEDPYLE SISELDDVLL KFKKYSKSMS SIENKVFSSS SGCFKSKNE RVDAYSFACS SYTDKIEEYL YDPANSFPYK RGVKLVPKEN SIYVEVGADT DMYGICVDVC EFSCTAYVLP I TNNFEGYL VTRNPSIKIG EILDINNNGV IIKAGGGPPT AINIYALSDS FTINFAPEDG NQDQNRYPRQ EYSINLIKVA IF GNRGLEK TVNPDGG UniProtKB: Uncharacterized protein |
-分子 #4: DUF228 domain-containing protein
| 分子 | 名称: DUF228 domain-containing protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 130 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Borreliella burgdorferi B31 (バクテリア) Borreliella burgdorferi B31 (バクテリア) |
| 分子量 | 理論値: 21.274934 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGDTTQLVKE YQEKRSKLEK FMKNPQHDAS LLSNSNEFRD KNVEFFASGG TRTSKFDKLE NHPFLGYPYK RGVKRVIQEA QDNQSHYEP HVEAGGGEDL YGICIDIDEF SKTATIVPIT NNFEGYLVAK DSTVKVKDKL IFNKDGALEK VTGAPNKATI N ATALTDAK ...文字列: MGDTTQLVKE YQEKRSKLEK FMKNPQHDAS LLSNSNEFRD KNVEFFASGG TRTSKFDKLE NHPFLGYPYK RGVKRVIQEA QDNQSHYEP HVEAGGGEDL YGICIDIDEF SKTATIVPIT NNFEGYLVAK DSTVKVKDKL IFNKDGALEK VTGAPNKATI N ATALTDAK QISNEVYLVK VAVFGNKAMS RN UniProtKB: DUF228 domain-containing protein |
-分子 #5: Scaffold protein
| 分子 | 名称: Scaffold protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 826 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Borreliella burgdorferi B31 (バクテリア) Borreliella burgdorferi B31 (バクテリア) |
| 分子量 | 理論値: 26.710363 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTEKEEKEDL QAQDKEEQQI KADTKVISVQ EFEEYMRFKE QANSKSKETS RDLSINERIT KELAEVEERE RIEKQLLLEA ERINEIDTL AKAHLSNHFN KEVLLAKGYT LKDIMQAQRR ELVRKFVPIE QIKAIAKVSD ISHIDGEILE QLVSLAKVNI K LRKNASSS ...文字列: MTEKEEKEDL QAQDKEEQQI KADTKVISVQ EFEEYMRFKE QANSKSKETS RDLSINERIT KELAEVEERE RIEKQLLLEA ERINEIDTL AKAHLSNHFN KEVLLAKGYT LKDIMQAQRR ELVRKFVPIE QIKAIAKVSD ISHIDGEILE QLVSLAKVNI K LRKNASSS SSSVDSIKGN IAIKSEERAS LLDSNFVPIN FTEFVQAISN TYKQRRIQFY ENLKRHKRTS IA UniProtKB: Uncharacterized protein |
-分子 #6: Putative phage portal protein
| 分子 | 名称: Putative phage portal protein / タイプ: protein_or_peptide / ID: 6 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Borreliella burgdorferi B31 (バクテリア) Borreliella burgdorferi B31 (バクテリア) |
| 分子量 | 理論値: 47.095508 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MCDLRKTKLI DKISSLELYK YSIFFRNYIE NVAEDCLKNG LILESAAHNV SEVELARLKV QLKNALLNCI ISYRFHGIGY VLVKTKDTL IDLEQPVNIE LPIGFEYLDY EYVRDLGVDF DHITYKVKSN NKNNSLDAVK IHKSRLIIYE NFDYILKRYV P CYTESFLL ...文字列: MCDLRKTKLI DKISSLELYK YSIFFRNYIE NVAEDCLKNG LILESAAHNV SEVELARLKV QLKNALLNCI ISYRFHGIGY VLVKTKDTL IDLEQPVNIE LPIGFEYLDY EYVRDLGVDF DHITYKVKSN NKNNSLDAVK IHKSRLIIYE NFDYILKRYV P CYTESFLL DIYLFEKIYV EIERRIENHN FLFYKDESLV QLQDALSSAT TSLSALTQSN NDRGSGILSS FLRKQNSNNH SK DISNLRN LNDSLSQELA RLKSNLNNEG MFYTATPSAS LEVIKYDLSY LKEALALIKA KIGADTKEPL TRSFNEQAKG LGN DGKGDR SNYYDFLKGV QEQVENSCNL KLTKYFGLDM KFNSLIMLSE EQKVERDIKL IELYSKYNQL IQSSSFNNEE LAML KEKLF SF UniProtKB: Phage portal protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / 支持フィルム - #0 - Film type ID: 1 / 支持フィルム - #0 - 材質: CARBON / 支持フィルム - #0 - トポロジー: HOLEY / 支持フィルム - #1 - Film type ID: 2 / 支持フィルム - #1 - 材質: GRAPHENE / 支持フィルム - #1 - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 44.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: experimental model / 詳細: previous reconstruction |
|---|---|
| 得られたモデル |  PDB-8qo0: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)