+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of immature HTLV-1 CA-NTD from in vitro assembled MA126-CANC tubes: axis angle 20 degrees | |||||||||||||||
 マップデータ マップデータ | HTLV-1 MA126CANC tubes, axis angle 20, B-factor sharpened map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Retrovirus / HTLV / immature capsid / CA / CA-NTD / VIRAL PROTEIN | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Human T-cell leukemia virus type I (ウイルス) Human T-cell leukemia virus type I (ウイルス) | |||||||||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 6.1 Å | |||||||||||||||
 データ登録者 データ登録者 | Obr M / Percipalle M / Chernikova D / Yang H / Thader A / Pinke G / Porley D / Mansky LM / Dick RA / Schur FKM | |||||||||||||||
| 資金援助 |  オーストリア, オーストリア,  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Distinct stabilization of the human T cell leukemia virus type 1 immature Gag lattice. 著者: Martin Obr / Mathias Percipalle / Darya Chernikova / Huixin Yang / Andreas Thader / Gergely Pinke / Dario Porley / Louis M Mansky / Robert A Dick / Florian K M Schur /    要旨: Human T cell leukemia virus type 1 (HTLV-1) immature particles differ in morphology from other retroviruses, suggesting a distinct way of assembly. Here we report the results of cryo-electron ...Human T cell leukemia virus type 1 (HTLV-1) immature particles differ in morphology from other retroviruses, suggesting a distinct way of assembly. Here we report the results of cryo-electron tomography studies of HTLV-1 virus-like particles assembled in vitro, as well as derived from cells. This work shows that HTLV-1 uses a distinct mechanism of Gag-Gag interactions to form the immature viral lattice. Analysis of high-resolution structural information from immature capsid (CA) tubular arrays reveals that the primary stabilizing component in HTLV-1 is the N-terminal domain of CA. Mutagenesis analysis supports this observation. This distinguishes HTLV-1 from other retroviruses, in which the stabilization is provided primarily by the C-terminal domain of CA. These results provide structural details of the quaternary arrangement of Gag for an immature deltaretrovirus and this helps explain why HTLV-1 particles are morphologically distinct. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17940.map.gz emd_17940.map.gz | 51.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17940-v30.xml emd-17940-v30.xml emd-17940.xml emd-17940.xml | 26.5 KB 26.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_17940.png emd_17940.png | 164.9 KB | ||
| Filedesc metadata |  emd-17940.cif.gz emd-17940.cif.gz | 6.6 KB | ||
| その他 |  emd_17940_half_map_1.map.gz emd_17940_half_map_1.map.gz emd_17940_half_map_2.map.gz emd_17940_half_map_2.map.gz | 28.3 MB 28.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17940 http://ftp.pdbj.org/pub/emdb/structures/EMD-17940 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17940 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17940 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17940_validation.pdf.gz emd_17940_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17940_full_validation.pdf.gz emd_17940_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_17940_validation.xml.gz emd_17940_validation.xml.gz | 11.9 KB | 表示 | |
| CIF形式データ |  emd_17940_validation.cif.gz emd_17940_validation.cif.gz | 13.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17940 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17940 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17940 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17940 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pufMC  8pu6C  8pu7C  8pu8C  8pu9C  8puaC  8pubC  8pucC  8pudC  8pueC  8pugC  8puhC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17940.map.gz / 形式: CCP4 / 大きさ: 55.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17940.map.gz / 形式: CCP4 / 大きさ: 55.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HTLV-1 MA126CANC tubes, axis angle 20, B-factor sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.327 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: HTLV-1 MA126CANC tubes, axis angle 20, halfmap 2
| ファイル | emd_17940_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HTLV-1 MA126CANC tubes, axis angle 20, halfmap 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
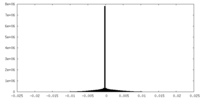
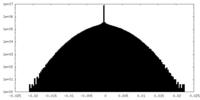
| 密度ヒストグラム |
-ハーフマップ: HTLV-1 MA126CANC tubes, axis angle 20, halfmap 1
| ファイル | emd_17940_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HTLV-1 MA126CANC tubes, axis angle 20, halfmap 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
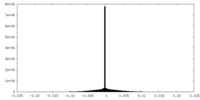
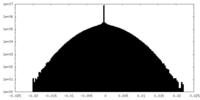
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human T-cell leukemia virus type I
| 全体 | 名称:  Human T-cell leukemia virus type I (ウイルス) Human T-cell leukemia virus type I (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human T-cell leukemia virus type I
| 超分子 | 名称: Human T-cell leukemia virus type I / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 11908 / 生物種: Human T-cell leukemia virus type I / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|
-分子 #1: Gag protein (Fragment)
| 分子 | 名称: Gag protein (Fragment) / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human T-cell leukemia virus type I (ウイルス) Human T-cell leukemia virus type I (ウイルス) |
| 分子量 | 理論値: 13.969809 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PVMHPHGAPP NHRPWQMKDL QAIKQEVSQA APGSPQFMQT IRLAVQQFDP TAKDLQDLLQ YLCSSLVASL HHQQLDSLIS EAETRGITG YNPLAGPLRV QANNPQQQGL RREYQQLWLA AFAALP UniProtKB: Gag protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: C-flat-2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AMYLAMINE | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 283 K / 装置: LEICA EM GP / 詳細: Grids coated with 2nm continuous carbon layer. |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | Image recording ID: 1 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 平均露光時間: 0.3675 sec. / 平均電子線量: 3.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 80000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #1~
電子顕微鏡法 #1~
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | Image recording ID: 2 フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3708 pixel / デジタル化 - サイズ - 縦: 3838 pixel / 平均露光時間: 1.05 sec. / 平均電子線量: 3.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Residue range: 13-125 / Chain - Source name: Other / Chain - Initial model type: other 詳細: rigid body fit derived from refined model deposited in D_1292131146 |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-8puf: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)