+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CUL2-RBX1-ELOB/C-FEM1C-SIL1 conformation 2 | |||||||||
 マップデータ マップデータ | DeepEMhancer sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CUL2 / FEM1C / ELOB/C / SIL1 / Ubiquitin / Ubiquitin Ligase / LIGASE | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
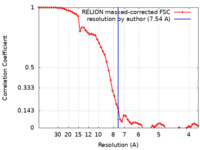
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.54 Å | |||||||||
 データ登録者 データ登録者 | Liwocha J / Prabu JR / Kleiger G / Schulman BA | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Mechanism of millisecond Lys48-linked poly-ubiquitin chain formation by cullin-RING ligases. 著者: Joanna Liwocha / Jerry Li / Nicholas Purser / Chutima Rattanasopa / Samuel Maiwald / David T Krist / Daniel C Scott / Barbara Steigenberger / J Rajan Prabu / Brenda A Schulman / Gary Kleiger /   要旨: E3 ubiquitin ligases, in collaboration with E2 ubiquitin-conjugating enzymes, modify proteins with poly-ubiquitin chains. Cullin-RING ligase (CRL) E3s use Cdc34/UBE2R-family E2s to build Lys48-linked ...E3 ubiquitin ligases, in collaboration with E2 ubiquitin-conjugating enzymes, modify proteins with poly-ubiquitin chains. Cullin-RING ligase (CRL) E3s use Cdc34/UBE2R-family E2s to build Lys48-linked poly-ubiquitin chains to control an enormous swath of eukaryotic biology. Yet the molecular mechanisms underlying this exceptional linkage specificity and millisecond kinetics of poly-ubiquitylation remain unclear. Here we obtain cryogenic-electron microscopy (cryo-EM) structures that provide pertinent insight into how such poly-ubiquitin chains are forged. The CRL RING domain not only activates the E2-bound ubiquitin but also shapes the conformation of a distinctive UBE2R2 loop, positioning both the ubiquitin to be transferred and the substrate-linked acceptor ubiquitin within the active site. The structures also reveal how the ubiquitin-like protein NEDD8 uniquely activates CRLs during chain formation. NEDD8 releases the RING domain from the CRL, but unlike previous CRL-E2 structures, does not contact UBE2R2. These findings suggest how poly-ubiquitylation may be accomplished by many E2s and E3s. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17799.map.gz emd_17799.map.gz | 16.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17799-v30.xml emd-17799-v30.xml emd-17799.xml emd-17799.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17799_fsc.xml emd_17799_fsc.xml | 6.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17799.png emd_17799.png | 26.7 KB | ||
| マスクデータ |  emd_17799_msk_1.map emd_17799_msk_1.map | 18.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17799.cif.gz emd-17799.cif.gz | 4 KB | ||
| その他 |  emd_17799_additional_1.map.gz emd_17799_additional_1.map.gz emd_17799_half_map_1.map.gz emd_17799_half_map_1.map.gz emd_17799_half_map_2.map.gz emd_17799_half_map_2.map.gz | 1.5 MB 13.8 MB 13.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17799 http://ftp.pdbj.org/pub/emdb/structures/EMD-17799 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17799 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17799 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17799_validation.pdf.gz emd_17799_validation.pdf.gz | 630.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17799_full_validation.pdf.gz emd_17799_full_validation.pdf.gz | 630.4 KB | 表示 | |
| XML形式データ |  emd_17799_validation.xml.gz emd_17799_validation.xml.gz | 11.4 KB | 表示 | |
| CIF形式データ |  emd_17799_validation.cif.gz emd_17799_validation.cif.gz | 15.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17799 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17799 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17799 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17799 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17799.map.gz / 形式: CCP4 / 大きさ: 18.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17799.map.gz / 形式: CCP4 / 大きさ: 18.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.885 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17799_msk_1.map emd_17799_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Relion PostProcessed Map
| ファイル | emd_17799_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Relion PostProcessed Map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17799_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17799_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CUL2-RBX1-ELOB/C-FEM1C-SIL1 conformation 2
| 全体 | 名称: CUL2-RBX1-ELOB/C-FEM1C-SIL1 conformation 2 |
|---|---|
| 要素 |
|
-超分子 #1: CUL2-RBX1-ELOB/C-FEM1C-SIL1 conformation 2
| 超分子 | 名称: CUL2-RBX1-ELOB/C-FEM1C-SIL1 conformation 2 / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 190 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.6 µm |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)