+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Orrella dioscoreae BcsD | |||||||||
 マップデータ マップデータ | Sharpened cryo-EM map of Orrella dioscoreae BcsD | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Bacterial cytoskeleton / bacterial cellulose / bacterial secretion / bacterial biofilms / STRUCTURAL PROTEIN | |||||||||
| 機能・相同性 | Cellulose synthase operon protein D, bacterial / Cellulose synthase subunit D superfamily / Cellulose synthase subunit D / cellulose biosynthetic process / Cellulose synthase operon protein D 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Orrella dioscoreae (バクテリア) Orrella dioscoreae (バクテリア) | |||||||||
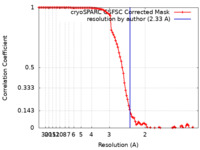
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.33 Å | |||||||||
 データ登録者 データ登録者 | Puygrenier L / Decossas M / Krasteva PV | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Curr Biol / 年: 2024 ジャーナル: Curr Biol / 年: 2024タイトル: Structures and roles of BcsD and partner scaffold proteins in proteobacterial cellulose secretion. 著者: Thibault G Sana / Areti Notopoulou / Lucie Puygrenier / Marion Decossas / Sandra Moreau / Aurélien Carlier / Petya V Krasteva /   要旨: Cellulose is the world's most abundant biopolymer, and similar to its role as a cell wall component in plants, it is a prevalent constituent of the extracellular matrix in bacterial biofilms. ...Cellulose is the world's most abundant biopolymer, and similar to its role as a cell wall component in plants, it is a prevalent constituent of the extracellular matrix in bacterial biofilms. Although bacterial cellulose (BC) was first described in the 19 century, it was only recently revealed that it is produced by several distinct types of Bcs secretion systems that feature multiple accessory subunits in addition to a catalytic BcsAB synthase tandem. We recently showed that crystalline cellulose secretion in the Gluconacetobacter genus (α-Proteobacteria) is driven by a supramolecular BcsH-BcsD scaffold-the "cortical belt"-which stabilizes the synthase nanoarrays through an unexpected inside-out mechanism for secretion system assembly. Interestingly, while bcsH is specific for Gluconacetobacter, bcsD homologs are widespread in Proteobacteria. Here, we examine BcsD homologs and their gene neighborhoods from several plant-colonizing β- and γ-Proteobacteria proposed to secrete a variety of non-crystalline and/or chemically modified cellulosic polymers. We provide structural and mechanistic evidence that through different quaternary structure assemblies BcsD acts with proline-rich BcsH, BcsP, or BcsO partners across the proteobacterial clade to form synthase-interacting intracellular scaffolds that, in turn, determine the biofilm strength and architecture in species with strikingly different physiology and secreted biopolymers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17735.map.gz emd_17735.map.gz | 96.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17735-v30.xml emd-17735-v30.xml emd-17735.xml emd-17735.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17735_fsc.xml emd_17735_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17735.png emd_17735.png | 50 KB | ||
| Filedesc metadata |  emd-17735.cif.gz emd-17735.cif.gz | 6.1 KB | ||
| その他 |  emd_17735_additional_1.map.gz emd_17735_additional_1.map.gz emd_17735_additional_2.map.gz emd_17735_additional_2.map.gz emd_17735_half_map_1.map.gz emd_17735_half_map_1.map.gz emd_17735_half_map_2.map.gz emd_17735_half_map_2.map.gz | 48.6 MB 91.3 MB 94.6 MB 94.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17735 http://ftp.pdbj.org/pub/emdb/structures/EMD-17735 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17735 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17735 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17735_validation.pdf.gz emd_17735_validation.pdf.gz | 796.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17735_full_validation.pdf.gz emd_17735_full_validation.pdf.gz | 796 KB | 表示 | |
| XML形式データ |  emd_17735_validation.xml.gz emd_17735_validation.xml.gz | 18.3 KB | 表示 | |
| CIF形式データ |  emd_17735_validation.cif.gz emd_17735_validation.cif.gz | 23.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17735 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17735 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17735 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17735 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17735.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17735.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened cryo-EM map of Orrella dioscoreae BcsD | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.657 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened cryo-EM map of Orrella dioscoreae BcsD
| ファイル | emd_17735_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened cryo-EM map of Orrella dioscoreae BcsD | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Deep EMhancer sharpened cryo-EM map of Orrella dioscoreae BcsD
| ファイル | emd_17735_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Deep EMhancer sharpened cryo-EM map of Orrella dioscoreae BcsD | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: EM half-map for O. dioscoreae BcsD
| ファイル | emd_17735_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half-map for O. dioscoreae BcsD | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: EM half-map for O. dioscoreae BcsD
| ファイル | emd_17735_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EM half-map for O. dioscoreae BcsD | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Orrella dioscoreae BcsD
| 全体 | 名称: Orrella dioscoreae BcsD |
|---|---|
| 要素 |
|
-超分子 #1: Orrella dioscoreae BcsD
| 超分子 | 名称: Orrella dioscoreae BcsD / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Orrella dioscoreae (バクテリア) Orrella dioscoreae (バクテリア) |
| 分子量 | 理論値: 69.7 KDa |
-分子 #1: Cellulose synthase operon protein D
| 分子 | 名称: Cellulose synthase operon protein D / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Orrella dioscoreae (バクテリア) Orrella dioscoreae (バクテリア) |
| 分子量 | 理論値: 17.448371 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSMENPLFD YYRNRQAPLQ WRGALGALAQ SLTNHFSPEQ LRTLLREAGQ HFASQHPVQA AETVQSMQDA MNGVWTTQDW GWVDIHDLD SFLTLTHYAA PLESAFGAQN LAWSAAFLEG VYEQWFRQLG ASDALHVRQS EESDVRKAIV LRLGR UniProtKB: Cellulose synthase operon protein D |
-分子 #2: water
| 分子 | 名称: water / タイプ: ligand / ID: 2 / コピー数: 16 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 100 mM NaCl, 20 mM HEPES pH 8.0 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | The sample was monodispersed, size-exclusion purified protein. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 19118 / 平均電子線量: 51.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.4 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 詳細 | Iterative model building and refinement in Coot, Namdinator and Phenix. |
| 精密化 | 空間: REAL / プロトコル: OTHER |
| 得られたモデル |  PDB-8pkd: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)