+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Chaetomium thermophilum Get1/Get2 heterotetramer in complex with a Get3 dimer (amphipol) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | membrane protein insertion / GET pathway / tail anchored membrane protein / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GET complex / tail-anchored membrane protein insertion into ER membrane / protein insertion into ER membrane / post-translational protein targeting to endoplasmic reticulum membrane / retrograde vesicle-mediated transport, Golgi to endoplasmic reticulum / protein-membrane adaptor activity / endoplasmic reticulum membrane / ATP hydrolysis activity / ATP binding / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Thermochaetoides thermophila (菌類) / Thermochaetoides thermophila (菌類) /  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) | |||||||||
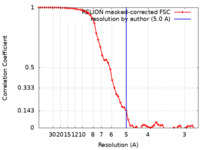
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.0 Å | |||||||||
 データ登録者 データ登録者 | McDowell MA / Wild K / Sinning I | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: The GET insertase exhibits conformational plasticity and induces membrane thinning. 著者: Melanie A McDowell / Michael Heimes / Giray Enkavi / Ákos Farkas / Daniel Saar / Klemens Wild / Blanche Schwappach / Ilpo Vattulainen / Irmgard Sinning /   要旨: The eukaryotic guided entry of tail-anchored proteins (GET) pathway mediates the biogenesis of tail-anchored (TA) membrane proteins at the endoplasmic reticulum. In the cytosol, the Get3 chaperone ...The eukaryotic guided entry of tail-anchored proteins (GET) pathway mediates the biogenesis of tail-anchored (TA) membrane proteins at the endoplasmic reticulum. In the cytosol, the Get3 chaperone captures the TA protein substrate and delivers it to the Get1/Get2 membrane protein complex (GET insertase), which then inserts the substrate via a membrane-embedded hydrophilic groove. Here, we present structures, atomistic simulations and functional data of human and Chaetomium thermophilum Get1/Get2/Get3. The core fold of the GET insertase is conserved throughout eukaryotes, whilst thinning of the lipid bilayer occurs in the vicinity of the hydrophilic groove to presumably lower the energetic barrier of membrane insertion. We show that the gating interaction between Get2 helix α3' and Get3 drives conformational changes in both Get3 and the Get1/Get2 membrane heterotetramer. Thus, we provide a framework to understand the conformational plasticity of the GET insertase and how it remodels its membrane environment to promote substrate insertion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16817.map.gz emd_16817.map.gz | 14.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16817-v30.xml emd-16817-v30.xml emd-16817.xml emd-16817.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16817_fsc.xml emd_16817_fsc.xml | 5.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16817.png emd_16817.png | 47.9 KB | ||
| Filedesc metadata |  emd-16817.cif.gz emd-16817.cif.gz | 6.7 KB | ||
| その他 |  emd_16817_half_map_1.map.gz emd_16817_half_map_1.map.gz emd_16817_half_map_2.map.gz emd_16817_half_map_2.map.gz | 13.8 MB 13.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16817 http://ftp.pdbj.org/pub/emdb/structures/EMD-16817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16817 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16817_validation.pdf.gz emd_16817_validation.pdf.gz | 717.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16817_full_validation.pdf.gz emd_16817_full_validation.pdf.gz | 716.9 KB | 表示 | |
| XML形式データ |  emd_16817_validation.xml.gz emd_16817_validation.xml.gz | 10.7 KB | 表示 | |
| CIF形式データ |  emd_16817_validation.cif.gz emd_16817_validation.cif.gz | 14.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16817 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16817 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16817 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16817 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16817.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16817.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.375 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_16817_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
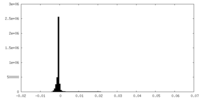
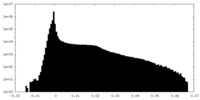
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16817_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
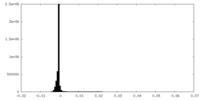
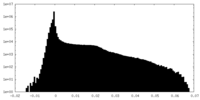
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Chaetomium thermophilum Get1/Get2 heterotetramer in complex with ...
| 全体 | 名称: Chaetomium thermophilum Get1/Get2 heterotetramer in complex with a Get3 dimer |
|---|---|
| 要素 |
|
-超分子 #1: Chaetomium thermophilum Get1/Get2 heterotetramer in complex with ...
| 超分子 | 名称: Chaetomium thermophilum Get1/Get2 heterotetramer in complex with a Get3 dimer タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: Get2-Get1 was expressed as a fusion protein in S. cerevisiae and Get3 was expressed in E. coli. The complex components were purified and reconstituted in vitro. |
|---|---|
| 分子量 | 理論値: 165 KDa |
-超分子 #2: Tail-anchored protein insertase Get1 and Get2
| 超分子 | 名称: Tail-anchored protein insertase Get1 and Get2 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila (菌類) Thermochaetoides thermophila (菌類) |
-超分子 #3: Dimeric ATPase Get3
| 超分子 | 名称: Dimeric ATPase Get3 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila (菌類) Thermochaetoides thermophila (菌類) |
-分子 #1: ATPase GET3
| 分子 | 名称: ATPase GET3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: 加水分解酵素; 酸無水物に作用 |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) |
| 分子量 | 理論値: 37.127391 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEPTLQSILD QRSLRWIFVG GKGGVGKTTT SCSLAIQLAK VRRSVLLLST DPAHNLSDAF SQKFGKEARL VEGFDNLYAM EIDPNGSMQ DLLAGQTGDG DAGMGGVGVM QDLAYAIPGI DEAMSFAEVL KQVNSLSYET IVFDTAPTGH TLRFLQFPTV L EKALAKVS ...文字列: MEPTLQSILD QRSLRWIFVG GKGGVGKTTT SCSLAIQLAK VRRSVLLLST DPAHNLSDAF SQKFGKEARL VEGFDNLYAM EIDPNGSMQ DLLAGQTGDG DAGMGGVGVM QDLAYAIPGI DEAMSFAEVL KQVNSLSYET IVFDTAPTGH TLRFLQFPTV L EKALAKVS QLSGQYGSLL NGILGGSGTL PNGQTLSDVM EKLDSLRVTI SEVNAQFKDE RLTTFVCVCI PEFLSLYETE RM IQELANY GIDTHCIVVN QLLFPKPGSD CEQCTARRRM QKKYLDQIEE LYDEEFNVVK MPLLVEEVRG KERLEKFSEM LIK PFVPPE WSHPQFEK UniProtKB: Arsenite translocating ATPase-like protein |
-分子 #2: Protein GET2,Protein GET1
| 分子 | 名称: Protein GET2,Protein GET1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Thermochaetoides thermophila DSM 1495 (菌類) Thermochaetoides thermophila DSM 1495 (菌類) |
| 分子量 | 理論値: 45.573727 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGRPTPLWRF LHTLLAVALG LAVIMLSPFG GTKLERDRAA AAVAGSASER EWLASLTDSY PLVKTGLGGG LFWAFATGEA ILLGTRWLF LSKKKKAATA AAKVNNNNGE GDDAELDSVE QAIELALEFF PAIRQPVEYL RPKVAVAMRY VDVGMTLWRD V MLALFVLG ...文字列: MGRPTPLWRF LHTLLAVALG LAVIMLSPFG GTKLERDRAA AAVAGSASER EWLASLTDSY PLVKTGLGGG LFWAFATGEA ILLGTRWLF LSKKKKAATA AAKVNNNNGE GDDAELDSVE QAIELALEFF PAIRQPVEYL RPKVAVAMRY VDVGMTLWRD V MLALFVLG AVAWWRAGSG SENLYFQSGS GSMSLLLVIF LLELVVQLVN TIGAKTINNL LWRFYLSIPG SPLAKDFAEQ RA KQKEYLQ VRHDLNATSS QDEFAKWARL QRKHDKLMDE LEKKKSQLDA HRTSFSRKLT IYRWILTRGM QWFLCFWFSS QPM FWLPYG WFPYWVEWLV SFPNAPMGSV SIVVWQSACS GVLALVIEAV MAVVRYTGGT GMQKQRQPVP AAGGAPGTSK KDLG SGSLE VLFQ UniProtKB: Uncharacterized protein, Protein GET1 |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.4 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 279 K / 装置: FEI VITROBOT MARK IV | ||||||
| 詳細 | Complex stabilised in A835 amphipol |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 5599 / 平均露光時間: 4.0 sec. / 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X