+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | COPII inner coat reprocessed with relion4.0 | |||||||||
 マップデータ マップデータ | COPII inner coat | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | COPII / Sec23 Sec24 Sar1 / PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Antigen Presentation: Folding, assembly and peptide loading of class I MHC / Cargo concentration in the ER / regulation of COPII vesicle coating / positive regulation of ER to Golgi vesicle-mediated transport / mitochondria-associated endoplasmic reticulum membrane contact site / COPII-mediated vesicle transport / COPII-coated vesicle budding / nuclear envelope organization / vesicle organization / COPII vesicle coat ...Antigen Presentation: Folding, assembly and peptide loading of class I MHC / Cargo concentration in the ER / regulation of COPII vesicle coating / positive regulation of ER to Golgi vesicle-mediated transport / mitochondria-associated endoplasmic reticulum membrane contact site / COPII-mediated vesicle transport / COPII-coated vesicle budding / nuclear envelope organization / vesicle organization / COPII vesicle coat / positive regulation of protein exit from endoplasmic reticulum / membrane organization / mitochondrial fission / mitochondrial membrane organization / endoplasmic reticulum exit site / endoplasmic reticulum to Golgi vesicle-mediated transport / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / intracellular protein transport / Golgi membrane / GTPase activity / endoplasmic reticulum membrane / GTP binding / Golgi apparatus / endoplasmic reticulum / mitochondrion / zinc ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
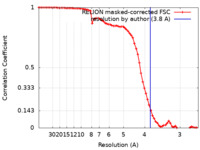
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Zanetti G / Pyle E | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2022 ジャーナル: Elife / 年: 2022タイトル: A Bayesian approach to single-particle electron cryo-tomography in RELION-4.0. 著者: Jasenko Zivanov / Joaquín Otón / Zunlong Ke / Andriko von Kügelgen / Euan Pyle / Kun Qu / Dustin Morado / Daniel Castaño-Díez / Giulia Zanetti / Tanmay A M Bharat / John A G Briggs / Sjors H W Scheres /     要旨: We present a new approach for macromolecular structure determination from multiple particles in electron cryo-tomography (cryo-ET) data sets. Whereas existing subtomogram averaging approaches are ...We present a new approach for macromolecular structure determination from multiple particles in electron cryo-tomography (cryo-ET) data sets. Whereas existing subtomogram averaging approaches are based on 3D data models, we propose to optimise a regularised likelihood target that approximates a function of the 2D experimental images. In addition, analogous to Bayesian polishing and contrast transfer function (CTF) refinement in single-particle analysis, we describe the approaches that exploit the increased signal-to-noise ratio in the averaged structure to optimise tilt-series alignments, beam-induced motions of the particles throughout the tilt-series acquisition, defoci of the individual particles, as well as higher-order optical aberrations of the microscope. Implementation of our approaches in the open-source software package RELION aims to facilitate their general use, particularly for those researchers who are already familiar with its single-particle analysis tools. We illustrate for three applications that our approaches allow structure determination from cryo-ET data to resolutions sufficient for de novo atomic modelling. #1:  ジャーナル: Biorxiv / 年: 2022 ジャーナル: Biorxiv / 年: 2022タイトル: A Bayesian approach to single-particle electron cryo-tomography in RELION-4.0 著者: Zivanov J / Oton J / Ke Z / von Kugelgen A / Pyle E / Qu K / Morado D / Castano-Diez D / Zanetti G / Bharat TAM / Briggs JAG / Scheres SHW | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15949.map.gz emd_15949.map.gz | 18.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15949-v30.xml emd-15949-v30.xml emd-15949.xml emd-15949.xml | 20.6 KB 20.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15949_fsc.xml emd_15949_fsc.xml | 7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15949.png emd_15949.png | 156 KB | ||
| マスクデータ |  emd_15949_msk_1.map emd_15949_msk_1.map | 28.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-15949.cif.gz emd-15949.cif.gz | 6.5 KB | ||
| その他 |  emd_15949_half_map_1.map.gz emd_15949_half_map_1.map.gz emd_15949_half_map_2.map.gz emd_15949_half_map_2.map.gz | 14.1 MB 14.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15949 http://ftp.pdbj.org/pub/emdb/structures/EMD-15949 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15949 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15949 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15949_validation.pdf.gz emd_15949_validation.pdf.gz | 912.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15949_full_validation.pdf.gz emd_15949_full_validation.pdf.gz | 912.4 KB | 表示 | |
| XML形式データ |  emd_15949_validation.xml.gz emd_15949_validation.xml.gz | 14.4 KB | 表示 | |
| CIF形式データ |  emd_15949_validation.cif.gz emd_15949_validation.cif.gz | 18.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15949 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15949 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15949 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15949 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15949.map.gz / 形式: CCP4 / 大きさ: 28.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15949.map.gz / 形式: CCP4 / 大きさ: 28.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | COPII inner coat | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_15949_msk_1.map emd_15949_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half2
| ファイル | emd_15949_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half1
| ファイル | emd_15949_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : COPII coat assembled on membrane, inner coat
| 全体 | 名称: COPII coat assembled on membrane, inner coat |
|---|---|
| 要素 |
|
-超分子 #1: COPII coat assembled on membrane, inner coat
| 超分子 | 名称: COPII coat assembled on membrane, inner coat / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|
-超分子 #2: Protein transport protein SEC23
| 超分子 | 名称: Protein transport protein SEC23 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1, #4 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Protein transport protein SEC24
| 超分子 | 名称: Protein transport protein SEC24 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #4: Small COPII coat GTPase SAR1
| 超分子 | 名称: Small COPII coat GTPase SAR1 / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3, #5 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: inner COPII coat complex
| 分子 | 名称: inner COPII coat complex / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Spodoptera (蝶・蛾) Spodoptera (蝶・蛾) |
| 配列 | 文字列: DFETNEDING VRFTWNVFPS TRSDANSNVV PVGCLYTPLK EYDELNVAPY NPVVCSGPHC KSILNPYCVI DPRNSSWSCP ICNSRNHLPP QYTNLSQENM PLELQSTTIE YITNKPVTVP PIFFFVVDLT SETENLDSLK ESIITSLSLL PPNALIGLIT YGNVVQLHDL ...文字列: DFETNEDING VRFTWNVFPS TRSDANSNVV PVGCLYTPLK EYDELNVAPY NPVVCSGPHC KSILNPYCVI DPRNSSWSCP ICNSRNHLPP QYTNLSQENM PLELQSTTIE YITNKPVTVP PIFFFVVDLT SETENLDSLK ESIITSLSLL PPNALIGLIT YGNVVQLHDL SSETIDRCNV FRGDREYQLE ALTEMLTGQK PTGPGGAASH LPNAMNKVTP FSLNRFFLP LEQVEFKLNQ LLENLSPDQW SVPAGHRPLR ATGSALNIAS LLLQGCYKNI PARIILFASG PGTVAPGLIV NSELKDPLRS HHDIDSDHAQ HYKKACKFYN QIAQRVAANG HTVDIFAGCY DQIGMSEMKQ LTDSTGGVLL LTDAFSTAIF KQSYLRLFAK DEEGYLKMAF NGNMAVKTSK DLKVQGLIGH ASAVKKTDAN NISESEIGIG A TSTWKMAS LSPYHSYAIF FEIANTAANS NPMMSAPGSA DRPHLAYTQF ITTYQHSSGT NRIRVTTVAN QLLPFGTPAI AASFDQEAAA VLMARIAVHK AETDDGADVI RWLDRTLIKL CQKYADYNKD DPQSFRLAPN FSLYPQFTYY LRRSQFLSVF NNSPDETAFY RHIFTREDTT NSLIMIQPTL TSFSMEDDPQ PVLLDSISVK PNTILLLDTF FF ILIYHGE QIAQWRKAGY QDDPQYADFK ALLEEPKLEA AELLVDRFPL PRFIDTEAGG SQARFLLSKL NPSDNYQDMA RGGSTIVLTD DVSLQNFMTH LQQVAVSGQA MSHHKKRVY PQAQLQYGQN ATPLQQPAQF MPPQDPAAAG MSYGQMGMPP QGAVPSMGQQ QFLTPAQEQL HQQIDQATTS MNDMHLHNVP LVDPNAYMQP QVPVQMGTPL QQQQQPMAAP AYGQPSAAMG QNMRPMNQLY PIDLLTELPP PITDLTLPPP PLVIPPERML VPSELSNASP DYIRSTLNAV PKNSSLLKKS KLPFGLVIRP YQHLYDDIDP P PLNEDGLI VRCRRCRSYM NPFVTFIEQG RRWRCNFCRL ANDVPMQMDQ SDPNDPKSRY DRNEIKCAVM EYMAPKEYTL RQPPPATYCF LIDVSQSSIK SGLLATTINT LLQNLDSIPN HDERTRISIL CVDNAIHYFK IPLDSENNEE SADQINMMDI ADLEEPFLPR PNSMVVSLKA CRQNIETLLT KIPQIFQSNL ITNFALGPAL KSAYHLIGGV GG KIIVVSG TLPNLGIGKL QRRNESGVVN TSKETAQLLS CQDSFYKNFT IDCSKVQITV DLFLASEDYM DVASLSNLSR FTAGQTHFYP GFSGKNPNDI VKFSTEFAKH ISMDFCMETV MRARGSTGLR MSRFYGHFFN RSSDLCAFST MPRDQSYLFE VNVDESIMAD YCYVQVAVLL SLNNSQRRIR IITLAMPTTE SLAEVYASAD QLAIASFYNS KAV EKALNS SLDDARVLIN KSVQDILATY KKEIVVSNTA GGAPLRLCAN LRMFPLLMHS LTKHMAFRSG IVPSDHRASA LNNLESLPLK YLIKNIYPDV YSLHDMADEA GLPVQTEDGE ATGTIVLPQP INATSSLFER YGLYLIDNGN ELFLWMGGDA VPALVFDVFG TQDIFDIPIG KQEIPVVENS EFNQRVRNII NQLRNHDDVI TYQSLYIVRG ASLS EPVNH ASAREVATLR LWASSTLVED KILNNESYRE FLQIMKARIS K MAGWDIFG WFRDVLASLG LWNKHGKLLF LGLDNAGKTT LLHMLKNDRL ATLQPTWHPT SEELAIGNIK FTTFDLGGHI QARRLWKDYF PEVNGIVFLV DAADPERFDE ARVELDALFN IAELKDVPFV ILGNKIDAPN AVSEAELRSA LGLLNTTGSQ RIEGQRPVEV FMCSVVMRNG YLEAFQWLSQ YI |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 3.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 3.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X