+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Poliovirus type 3 (strain Saukett) stabilised virus-like particle (PV3 SC8). | |||||||||
 マップデータ マップデータ | Half map after RELION 3D refinement. Contour level recommended based on viewing in UCSF chimeraX | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Capsid protein / VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont genome entry into host cell via pore formation in plasma membrane / symbiont-mediated suppression of host gene expression / viral capsid / virion attachment to host cell / structural molecule activity 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human poliovirus 3 (ポリオウイルス) Human poliovirus 3 (ポリオウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Bahar MW / Fry EE / Stuart DI | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Viruses / 年: 2022 ジャーナル: Viruses / 年: 2022タイトル: Production and Characterisation of Stabilised PV-3 Virus-like Particles Using . 著者: Lee Sherry / Keith Grehan / Jessica J Swanson / Mohammad W Bahar / Claudine Porta / Elizabeth E Fry / David I Stuart / David J Rowlands / Nicola J Stonehouse /  要旨: Following the success of global vaccination programmes using the live-attenuated oral and inactivated poliovirus vaccines (OPV and IPV), wild poliovirus (PV) is now only endemic in Afghanistan and ...Following the success of global vaccination programmes using the live-attenuated oral and inactivated poliovirus vaccines (OPV and IPV), wild poliovirus (PV) is now only endemic in Afghanistan and Pakistan. However, the continued use of these vaccines poses potential risks to the eradication of PV. The production of recombinant PV virus-like particles (VLPs), which lack the viral genome offer great potential as next-generation vaccines for the post-polio world. We have previously reported production of PV VLPs using , however, these VLPs were in the non-native conformation (C Ag), which would not produce effective protection against PV. Here, we build on this work and show that it is possible to produce wt PV-3 and thermally stabilised PV-3 (referred to as PV-3 SC8) VLPs in the native conformation (D Ag) using . We show that the PV-3 SC8 VLPs provide a much-improved D:C antigen ratio as compared to wt PV-3, whilst exhibiting greater thermostability than the current IPV vaccine. Finally, we determine the cryo-EM structure of the yeast-derived PV-3 SC8 VLPs and compare this to previously published PV-3 D Ag structures, highlighting the similarities between these recombinantly expressed VLPs and the infectious virus, further emphasising their potential as a next-generation vaccine candidate for PV. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15543.map.gz emd_15543.map.gz | 209.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15543-v30.xml emd-15543-v30.xml emd-15543.xml emd-15543.xml | 23.4 KB 23.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15543_fsc.xml emd_15543_fsc.xml | 15.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15543.png emd_15543.png | 112.1 KB | ||
| Filedesc metadata |  emd-15543.cif.gz emd-15543.cif.gz | 7.5 KB | ||
| その他 |  emd_15543_half_map_1.map.gz emd_15543_half_map_1.map.gz emd_15543_half_map_2.map.gz emd_15543_half_map_2.map.gz | 276.6 MB 276.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15543 http://ftp.pdbj.org/pub/emdb/structures/EMD-15543 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15543 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15543 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15543_validation.pdf.gz emd_15543_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15543_full_validation.pdf.gz emd_15543_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_15543_validation.xml.gz emd_15543_validation.xml.gz | 23.8 KB | 表示 | |
| CIF形式データ |  emd_15543_validation.cif.gz emd_15543_validation.cif.gz | 31.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15543 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15543 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15543 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15543 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8anwMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15543.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15543.map.gz / 形式: CCP4 / 大きさ: 347.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map after RELION 3D refinement. Contour level recommended based on viewing in UCSF chimeraX | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map after RELION 3D refinement. Contour level...
| ファイル | emd_15543_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map after RELION 3D refinement. Contour level recommended based on viewing in UCSF chimeraX. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
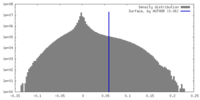
| 密度ヒストグラム |
-ハーフマップ: Half map after RELION 3D refinement. Contour level...
| ファイル | emd_15543_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map after RELION 3D refinement. Contour level recommended based on viewing in UCSF chimeraX. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human poliovirus 3
| 全体 | 名称:  Human poliovirus 3 (ポリオウイルス) Human poliovirus 3 (ポリオウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Human poliovirus 3
| 超分子 | 名称: Human poliovirus 3 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: Recombinantly expressed virus-like particle of PV3 (strain Saukett). NCBI-ID: 12086 / 生物種: Human poliovirus 3 / Sci species strain: Saukett / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 5.85 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Virus shell 1 / 直径: 310.0 Å / T番号(三角分割数): 1 |
-分子 #1: Capsid protein, VP1
| 分子 | 名称: Capsid protein, VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human poliovirus 3 (ポリオウイルス) Human poliovirus 3 (ポリオウイルス) |
| 分子量 | 理論値: 33.562785 KDa |
| 組換発現 | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: GIEDLITEVA QGALTLSLPK QQDSLPDTKA SGPAHSKEVP ALTAVETGAT NPLVPSDTVQ TRHVIQRRSR SESTIESFFA RGACVAIIE VDNEEPTTRA QKLFAMWRIT YKDTVQLRRK LEFFTYSRFD MELTFVVTAN FTNTNNGHAL NQVYQIMYIP P GAPTPKSW ...文字列: GIEDLITEVA QGALTLSLPK QQDSLPDTKA SGPAHSKEVP ALTAVETGAT NPLVPSDTVQ TRHVIQRRSR SESTIESFFA RGACVAIIE VDNEEPTTRA QKLFAMWRIT YKDTVQLRRK LEFFTYSRFD MELTFVVTAN FTNTNNGHAL NQVYQIMYIP P GAPTPKSW DDYTWQTSSN PSIFYTYGAA PARISVPYVG LANAYSHFYD GFAKVPLKTD ANDQIGDSLY SAMTVDDFGV LA IRVVNDH NPTKVTSKVR IYMKPKHVRV WCPRPPRAVP YYGPGVDYKD NLNPLSEKGL TTY UniProtKB: Genome polyprotein |
-分子 #2: Capsid protein, VP0
| 分子 | 名称: Capsid protein, VP0 / タイプ: protein_or_peptide / ID: 2 詳細: Sequence is given for the VP0 polypeptide. Mutations are numbered according to sequence numbering for mature polypeptides VP2 and VP4. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human poliovirus 3 (ポリオウイルス) Human poliovirus 3 (ポリオウイルス) |
| 分子量 | 理論値: 37.623023 KDa |
| 組換発現 | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: MGAQVSSQKV GAHENSNRAY GGSTINYTTI NYYKDSASNA ASKQDYSQDP SKFTEPLKDV LIKTAPALNS PNVEACGYSD RVLQLTIGN STITTQEAAN SVVAYGRWPE FIRDDEANPV DQPTEPDVAT CRFYTLDTVM WGKESKGWWW KLPDALRDMG L FGQNMYYH ...文字列: MGAQVSSQKV GAHENSNRAY GGSTINYTTI NYYKDSASNA ASKQDYSQDP SKFTEPLKDV LIKTAPALNS PNVEACGYSD RVLQLTIGN STITTQEAAN SVVAYGRWPE FIRDDEANPV DQPTEPDVAT CRFYTLDTVM WGKESKGWWW KLPDALRDMG L FGQNMYYH YLGRSGYTVH VQCNASKFHQ GALGVFAIPE YCLAGDSDKQ RYTSYANANP GEKGGKFYSQ FNRDTAVTSP KR EFCPVDY LLGCGVLLGN AFVYPHQIIN LRTNNSATIV LPYVNAMAID SMVKHNNWGI AILPLSPLDF AQESSVEIPI TVT IAPMCS EFNGLRNVTA PKFQ UniProtKB: Genome polyprotein |
-分子 #3: Capsid protein, VP3
| 分子 | 名称: Capsid protein, VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human poliovirus 3 (ポリオウイルス) Human poliovirus 3 (ポリオウイルス) |
| 分子量 | 理論値: 26.3151 KDa |
| 組換発現 | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: GLPVLNTPGS NQYLTSDNYQ SPCAIPEFDV TPPIDIPGEV KNMMELAEID TMIPLNLENT KRNTMDMYRV TLSDSADLSQ PILCFSLSP ASDPRLSHTM LGEVLNYYTH WAGSLKFTFL FCGSMMATGK ILVAYAPPGA QPPTSRKEAM LGTHVIWDLG L QSSCTMVV ...文字列: GLPVLNTPGS NQYLTSDNYQ SPCAIPEFDV TPPIDIPGEV KNMMELAEID TMIPLNLENT KRNTMDMYRV TLSDSADLSQ PILCFSLSP ASDPRLSHTM LGEVLNYYTH WAGSLKFTFL FCGSMMATGK ILVAYAPPGA QPPTSRKEAM LGTHVIWDLG L QSSCTMVV PWISNVTYRQ TTQDSFTEGG YISMFYQTRI VVPLSTPKSM SMLGFVSACN DFSVRLLRDT THISQSALPQ UniProtKB: Genome polyprotein |
-分子 #4: SPHINGOSINE
| 分子 | 名称: SPHINGOSINE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: SPH |
|---|---|
| 分子量 | 理論値: 299.492 Da |
| Chemical component information |  ChemComp-SPH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7 / 構成要素:
| ||||||
| グリッド | モデル: EMS Lacey Carbon / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 支持フィルム - Film thickness: 3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR 詳細: The specific type of grid used was Ultra-thin carbon support film, 3nm - on lacey carbon AGS187-4 from Agar Scientific. | ||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: Double blotting with 4 ul of sample, followed by 4 second blot, before plunging.. | ||||||
| 詳細 | Virus-like particle purified by sucrose density gradient purification from Pichia pastoris cells. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-20 / 撮影したグリッド数: 1 / 実像数: 3717 / 平均露光時間: 3.0 sec. / 平均電子線量: 35.0 e/Å2 / 詳細: Pixel sampling of 0.82 A/pixel. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 60975 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Initial model was rigid body fitted using UCSF chimera and Coot. Global minimization and B-factor refinement was performed in real space using phenix_real.space.refine. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Correlation coefficient |
| 得られたモデル |  PDB-8anw: |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)