+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | human MutSalpha mutant (MSH2-V63E/MSH6) on DNA containing a GT mismatch | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Bruekner SR / Sixma TK | |||||||||
| 資金援助 |  オランダ, European Union, 2件 オランダ, European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res. / 年: 2023 ジャーナル: Nucleic Acids Res. / 年: 2023タイトル: Unexpected moves: a conformational change in MutSalpha enables high affinity DNA mismatch binding 著者: Bruekner SR / Pieters W / Fish A / Liaci AM / Scheffers S / Rayner E / Kaldenbach D / Drost L / Dekker M / van Hees-Stuivenberg S / Delzenne-Goette E / de Konink C / Houllleberghs H / Dubbink ...著者: Bruekner SR / Pieters W / Fish A / Liaci AM / Scheffers S / Rayner E / Kaldenbach D / Drost L / Dekker M / van Hees-Stuivenberg S / Delzenne-Goette E / de Konink C / Houllleberghs H / Dubbink HJ / AlSaegh A / de Wind N / Forster F / te Riele H / Sixma TK | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15518.map.gz emd_15518.map.gz | 140.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15518-v30.xml emd-15518-v30.xml emd-15518.xml emd-15518.xml | 29.5 KB 29.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15518_fsc.xml emd_15518_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15518.png emd_15518.png | 146.2 KB | ||
| その他 |  emd_15518_additional_1.map.gz emd_15518_additional_1.map.gz emd_15518_additional_2.map.gz emd_15518_additional_2.map.gz emd_15518_half_map_1.map.gz emd_15518_half_map_1.map.gz emd_15518_half_map_2.map.gz emd_15518_half_map_2.map.gz | 141 MB 141.1 MB 140.8 MB 141 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15518 http://ftp.pdbj.org/pub/emdb/structures/EMD-15518 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15518 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15518 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15518_validation.pdf.gz emd_15518_validation.pdf.gz | 822.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15518_full_validation.pdf.gz emd_15518_full_validation.pdf.gz | 822.3 KB | 表示 | |
| XML形式データ |  emd_15518_validation.xml.gz emd_15518_validation.xml.gz | 19.5 KB | 表示 | |
| CIF形式データ |  emd_15518_validation.cif.gz emd_15518_validation.cif.gz | 25.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15518 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15518 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15518 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15518 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15518.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15518.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.836 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_15518_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
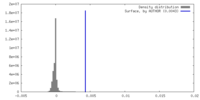
| 密度ヒストグラム |
-追加マップ: #2
| ファイル | emd_15518_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
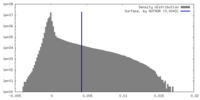
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15518_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
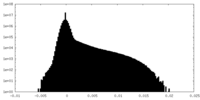
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15518_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
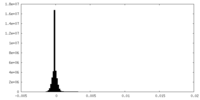
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : human MutSalpha mutant (MSH2-V63E/MSH6) on mismatched DNA
| 全体 | 名称: human MutSalpha mutant (MSH2-V63E/MSH6) on mismatched DNA |
|---|---|
| 要素 |
|
-超分子 #1: human MutSalpha mutant (MSH2-V63E/MSH6) on mismatched DNA
| 超分子 | 名称: human MutSalpha mutant (MSH2-V63E/MSH6) on mismatched DNA タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all 詳細: MSH2 has a valine to glutamate mutation at position 63 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 31 KDa |
-超分子 #2: MSH2 V63E
| 超分子 | 名称: MSH2 V63E / タイプ: complex / ID: 2 / キメラ: Yes / 親要素: 1 / 含まれる分子: #1 詳細: MSH2 has a valine to glutamate mutation at postition 63 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: MSH6
| 超分子 | 名称: MSH6 / タイプ: complex / ID: 3 / キメラ: Yes / 親要素: 1 / 含まれる分子: #2 / 詳細: MSH6 has an N-terminal 10His-TwinStrep tag |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #4: DNA (50-mer)
| 超分子 | 名称: DNA (50-mer) / タイプ: complex / ID: 4 / キメラ: Yes / 親要素: 1 / 含まれる分子: #3-#4 / 詳細: contains a GT mismatch at position 25 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: MSH2 V63E
| 分子 | 名称: MSH2 V63E / タイプ: protein_or_peptide / ID: 1 / 詳細: valine 63 is mutated to glutamate / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAVQPKETLQ LESAAEVGFV RFFQGMPEKP TTTVRLFDRG DFYTAHGEDA LLAAREVFKT QGEIKYMGPA GAKNLQSVVL SKMNFESFVK DLLLVRQYRV EVYKNRAGNK ASKENDWYLA YKASPGNLSQ FEDILFGNND MSASIGVVGV KMSAVDGQRQ VGVGYVDSIQ ...文字列: MAVQPKETLQ LESAAEVGFV RFFQGMPEKP TTTVRLFDRG DFYTAHGEDA LLAAREVFKT QGEIKYMGPA GAKNLQSVVL SKMNFESFVK DLLLVRQYRV EVYKNRAGNK ASKENDWYLA YKASPGNLSQ FEDILFGNND MSASIGVVGV KMSAVDGQRQ VGVGYVDSIQ RKLGLCEFPD NDQFSNLEAL LIQIGPKECV LPGGETAGDM GKLRQIIQRG GILITERKKA DFSTKDIYQD LNRLLKGKKG EQMNSAVLPE MENQVAVSSL SAVIKFLELL SDDSNFGQFE LTTFDFSQYM KLDIAAVRAL NLFQGSVEDT TGSQSLAALL NKCKTPQGQR LVNQWIKQPL MDKNRIEERL NLVEAFVEDA ELRQTLQEDL LRRFPDLNRL AKKFQRQAAN LQDCYRLYQG INQLPNVIQA LEKHEGKHQK LLLAVFVTPL TDLRSDFSKF QEMIETTLDM DQVENHEFLV KPSFDPNLSE LREIMNDLEK KMQSTLISAA RDLGLDPGKQ IKLDSSAQFG YYFRVTCKEE KVLRNNKNFS TVDIQKNGVK FTNSKLTSLN EEYTKNKTEY EEAQDAIVKE IVNISSGYVE PMQTLNDVLA QLDAVVSFAH VSNGAPVPYV RPAILEKGQG RIILKASRHA CVEVQDEIAF IPNDVYFEKD KQMFHIITGP NMGGKSTYIR QTGVIVLMAQ IGCFVPCESA EVSIVDCILA RVGAGDSQLK GVSTFMAEML ETASILRSAT KDSLIIIDEL GRGTSTYDGF GLAWAISEYI ATKIGAFCMF ATHFHELTAL ANQIPTVNNL HVTALTTEET LTMLYQVKKG VCDQSFGIHV AELANFPKHV IECAKQKALE LEEFQYIGES QGYDIMEPAA KKCYLEREQG EKIIQEFLSK VKQMPFTEMS EENITIKLKQ LKAEVIAKNN SFVNEIISRI KVTT |
-分子 #2: MSH6
| 分子 | 名称: MSH6 / タイプ: protein_or_peptide / ID: 2 / 詳細: N-terminal 10His-TwinStrep tag / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAHHHHHHHH HHGSAASWSH PQFEKGGGSG GGSGGGSWSH PQFEKGALEV LFQGPSRQST LYSFFPKSPA LSDANKASAR ASREGGRAAA APEASPSPGG DAAWSEAGPG PRPLARSASP PKAKNLNGGL RRSVAPAAPT SCDFSPGDLV WAKMEGYPWW PCLVYNHPFD ...文字列: MAHHHHHHHH HHGSAASWSH PQFEKGGGSG GGSGGGSWSH PQFEKGALEV LFQGPSRQST LYSFFPKSPA LSDANKASAR ASREGGRAAA APEASPSPGG DAAWSEAGPG PRPLARSASP PKAKNLNGGL RRSVAPAAPT SCDFSPGDLV WAKMEGYPWW PCLVYNHPFD GTFIREKGKS VRVHVQFFDD SPTRGWVSKR LLKPYTGSKS KEAQKGGHFY SAKPEILRAM QRADEALNKD KIKRLELAVC DEPSEPEEEE EMEVGTTYVT DKSEEDNEIE SEEEVQPKTQ GSRRSSRQIK KRRVISDSES DIGGSDVEFK PDTKEEGSSD EISSGVGDSE SEGLNSPVKV ARKRKRMVTG NGSLKRKSSR KETPSATKQA TSISSETKNT LRAFSAPQNS ESQAHVSGGG DDSSRPTVWY HETLEWLKEE KRRDEHRRRP DHPDFDASTL YVPEDFLNSC TPGMRKWWQI KSQNFDLVIC YKVGKFYELY HMDALIGVSE LGLVFMKGNW AHSGFPEIAF GRYSDSLVQK GYKVARVEQT ETPEMMEARC RKMAHISKYD RVVRREICRI ITKGTQTYSV LEGDPSENYS KYLLSLKEKE EDSSGHTRAY GVCFVDTSLG KFFIGQFSDD RHCSRFRTLV AHYPPVQVLF EKGNLSKETK TILKSSLSCS LQEGLIPGSQ FWDASKTLRT LLEEEYFREK LSDGIGVMLP QVLKGMTSES DSIGLTPGEK SELALSALGG CVFYLKKCLI DQELLSMANF EEYIPLDSDT VSTTRSGAIF TKAYQRMVLD AVTLNNLEIF LNGTNGSTEG TLLERVDTCH TPFGKRLLKQ WLCAPLCNHY AINDRLDAIE DLMVVPDKIS EVVELLKKLP DLERLLSKIH NVGSPLKSQN HPDSRAIMYE ETTYSKKKII DFLSALEGFK VMCKIIGIME EVADGFKSKI LKQVISLQTK NPEGRFPDLT VELNRWDTAF DHEKARKTGL ITPKAGFDSD YDQALADIRE NEQSLLEYLE KQRNRIGCRT IVYWGIGRNR YQLEIPENFT TRNLPEEYEL KSTKKGCKRY WTKTIEKKLA NLINAEERRD VSLKDCMRRL FYNFDKNYKD WQSAVECIAV LDVLLCLANY SRGGDGPMCR PVILLPEDTP PFLELKGSRH PCITKTFFGD DFIPNDILIG CEEEEQENGK AYCVLVTGPN MGGKSTLMRQ AGLLAVMAQM GCYVPAEVCR LTPIDRVFTR LGASDRIMSG ESTFFVELSE TASILMHATA HSLVLVDELG RGTATFDGTA IANAVVKELA ETIKCRTLFS THYHSLVEDY SQNVAVRLGH MACMVENECE DPSQETITFL YKFIKGACPK SYGFNAARLA NLPEEVIQKG HRKAREFEKM NQSLRLFREV CLASERSTVD AEAVHKLLTL IKEL |
-分子 #3: DNA (50-mer)
| 分子 | 名称: DNA (50-mer) / タイプ: dna / ID: 3 / 詳細: contains a GT mismatch / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: CTTAGCTTAG GATCATCGAG GATCGAGCTC GGTGCAATTC AGCGGTACCC |
-分子 #4: DNA (50-mer)
| 分子 | 名称: DNA (50-mer) / タイプ: dna / ID: 4 / 詳細: contains a GT mismatch / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: GGGTACCGCT GAATTGCACC GAGCTTGATC CTCGATGATC CTAAGCTAAG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.01 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 2 s blotting time with force 10. | |||||||||||||||
| 詳細 | 2 uM MutSalpha V63E with 2-fold excess mismatched DNA |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 詳細 | Data collected on Krios 1 at the Netherlands Center for Electron Nanoscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 4456 / 平均露光時間: 1.9 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)