[日本語] English
 万見
万見- EMDB-15322: Complex III2 from Yarrowia lipolytica,antimycin A bound, int-position -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Complex III2 from Yarrowia lipolytica,antimycin A bound, int-position | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | oxidoreductase / electron transport chain / inhibitor / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitochondrial processing peptidase complex / mitochondrial processing peptidase / matrix side of mitochondrial inner membrane / protein processing involved in protein targeting to mitochondrion / mitochondrial respiratory chain complex III assembly / : / quinol-cytochrome-c reductase / ubiquinol-cytochrome-c reductase activity / mitochondrial electron transport, ubiquinol to cytochrome c / mitochondrial crista ...mitochondrial processing peptidase complex / mitochondrial processing peptidase / matrix side of mitochondrial inner membrane / protein processing involved in protein targeting to mitochondrion / mitochondrial respiratory chain complex III assembly / : / quinol-cytochrome-c reductase / ubiquinol-cytochrome-c reductase activity / mitochondrial electron transport, ubiquinol to cytochrome c / mitochondrial crista / nuclear periphery / metalloendopeptidase activity / mitochondrial intermembrane space / 2 iron, 2 sulfur cluster binding / oxidoreductase activity / heme binding / mitochondrion / proteolysis / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Yarrowia lipolytica (酵母) Yarrowia lipolytica (酵母) | |||||||||
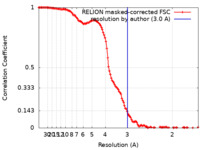
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Wieferig JP / Kuhlbrandt W | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: IUCrJ / 年: 2023 ジャーナル: IUCrJ / 年: 2023タイトル: Analysis of the conformational heterogeneity of the Rieske iron-sulfur protein in complex III by cryo-EM. 著者: Jan Philip Wieferig / Werner Kühlbrandt /  要旨: Movement of the Rieske domain of the iron-sulfur protein is essential for intramolecular electron transfer within complex III (CIII) of the respiratory chain as it bridges a gap in the cofactor chain ...Movement of the Rieske domain of the iron-sulfur protein is essential for intramolecular electron transfer within complex III (CIII) of the respiratory chain as it bridges a gap in the cofactor chain towards the electron acceptor cytochrome c. We present cryo-EM structures of CIII from Yarrowia lipolytica at resolutions up to 2.0 Å under different conditions, with different redox states of the cofactors of the high-potential chain. All possible permutations of three primary positions were observed, indicating that the two halves of the dimeric complex act independently. Addition of the substrate analogue decylubiquinone to CIII with a reduced high-potential chain increased the occupancy of the Q site. The extent of Rieske domain interactions through hydrogen bonds to the cytochrome b and cytochrome c subunits varied depending on the redox state and substrate. In the absence of quinols, the reduced Rieske domain interacted more closely with cytochrome b and cytochrome c than in the oxidized state. Upon addition of the inhibitor antimycin A, the heterogeneity of the cd-helix and ef-loop increased, which may be indicative of a long-range effect on the Rieske domain. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
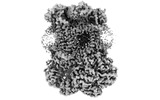
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15322.map.gz emd_15322.map.gz | 161.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15322-v30.xml emd-15322-v30.xml emd-15322.xml emd-15322.xml | 27.4 KB 27.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15322_fsc.xml emd_15322_fsc.xml | 12.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15322.png emd_15322.png | 65.4 KB | ||
| Filedesc metadata |  emd-15322.cif.gz emd-15322.cif.gz | 7.7 KB | ||
| その他 |  emd_15322_half_map_1.map.gz emd_15322_half_map_1.map.gz emd_15322_half_map_2.map.gz emd_15322_half_map_2.map.gz | 140.6 MB 140.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15322 http://ftp.pdbj.org/pub/emdb/structures/EMD-15322 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15322 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15322 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15322_validation.pdf.gz emd_15322_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15322_full_validation.pdf.gz emd_15322_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_15322_validation.xml.gz emd_15322_validation.xml.gz | 20 KB | 表示 | |
| CIF形式データ |  emd_15322_validation.cif.gz emd_15322_validation.cif.gz | 25.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15322 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15322 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15322 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15322 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8abiMC  8ab6C  8ab7C  8ab8C  8ab9C  8abaC  8abbC  8abeC  8abfC  8abgC  8abhC  8abjC  8abkC  8ablC  8abmC  8ac3C  8ac4C  8ac5C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15322.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15322.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.831 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_15322_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_15322_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Complex III2, with antmiycin A, int-position
+超分子 #1: Complex III2, with antmiycin A, int-position
+分子 #1: Cytochrome b
+分子 #2: Cytochrome b-c1 complex subunit Rieske, mitochondrial
+分子 #3: Cytochrome b-c1 complex subunit 7
+分子 #4: YALI0F24673p
+分子 #5: YALI0A14806p
+分子 #6: Cytochrome b-c1 complex subunit 2, mitochondrial
+分子 #7: YALI0A17468p
+分子 #8: Cytochrome b-c1 complex subunit 8
+分子 #9: Complex III subunit 9
+分子 #10: YALI0C12210p
+分子 #11: PROTOPORPHYRIN IX CONTAINING FE
+分子 #12: 1,2-DIACYL-SN-GLYCERO-3-PHOSPHOCHOLINE
+分子 #13: PHOSPHATIDYLETHANOLAMINE
+分子 #14: CARDIOLIPIN
+分子 #15: [(2R,3S,6S,7R,8R)-3-[(3-formamido-2-oxidanyl-phenyl)carbonylamino...
+分子 #16: FE2/S2 (INORGANIC) CLUSTER
+分子 #17: DODECYL-BETA-D-MALTOSIDE
+分子 #18: 1,2-DIMYRISTOYL-SN-GLYCERO-3-PHOSPHATE
+分子 #19: HEME C
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)