+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Full AAV3B-VP1KO virion | |||||||||
 マップデータ マップデータ | Sharpened cryo-EM map of the full AAV3B-VP1KO particle | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Phospholipase A2-like domain / Phospholipase A2-like domain / Parvovirus coat protein VP2 / Parvovirus coat protein VP1/VP2 / Parvovirus coat protein VP2 / Capsid/spike protein, ssDNA virus / T=1 icosahedral viral capsid / structural molecule activity / Capsid protein VP1 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Adeno-associated virus - 3 (アデノ随伴ウイルス) Adeno-associated virus - 3 (アデノ随伴ウイルス) | |||||||||
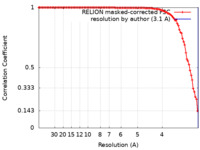
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Arriaga I / Abrescia NGA | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Hum Gene Ther / 年: 2022 ジャーナル: Hum Gene Ther / 年: 2022タイトル: Cellular and Structural Characterization of VP1 and VP2 Knockout Mutants of AAV3B Serotype and Implications for AAV Manufacturing. 著者: Iker Arriaga / Aitor Navarro / Amaia Etxabe / César Trigueros / R Jude Samulski / Philippe Moullier / Achille François / Nicola G A Abrescia /   要旨: AAV virion biology is still lacking a complete understanding of the role that the various structural subunits (VP1, 2, and 3) play in virus assembly, infectivity, and therapeutic delivery for ...AAV virion biology is still lacking a complete understanding of the role that the various structural subunits (VP1, 2, and 3) play in virus assembly, infectivity, and therapeutic delivery for clinical indications. In this study, we focus on the less studied adeno-associated virus AAV3B and generate a collection of AAV plasmid substrates that assemble virion particles deficient specifically in VP1, VP2, or VP1 and 2 structural subunits. Using a collection of biological and structural assays, we observed that virions devoid of VP1, VP2, or VP1 and 2 efficiently assembled virion particles, indistinguishable by cryoelectron microscopy (cryo-EM) from that of wild type (WT), but unique in virion transduction (WT > VP2 > VP1 > VP1 and 2 mutants). We also observed that the missing structural subunit was mostly compensated by additional VP3 protomers in the formed virion particle. Using cryo-EM analysis, virions fell into three classes, namely full, empty, and partially filled, based on comparison of density values within the capsid. Further, we characterize virions described as "broken" or "disassembled" particles, and provide structural information that supports the particle dissolution occurring through the two-fold symmetry sites. Finally, we highlight the unique value of employing cryo-EM as an essential tool for release criteria with respect to AAV manufacturing. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15286.map.gz emd_15286.map.gz | 96.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15286-v30.xml emd-15286-v30.xml emd-15286.xml emd-15286.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15286_fsc.xml emd_15286_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15286.png emd_15286.png | 196 KB | ||
| その他 |  emd_15286_additional_1.map.gz emd_15286_additional_1.map.gz emd_15286_half_map_1.map.gz emd_15286_half_map_1.map.gz emd_15286_half_map_2.map.gz emd_15286_half_map_2.map.gz | 79.1 MB 79.1 MB 79.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15286 http://ftp.pdbj.org/pub/emdb/structures/EMD-15286 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15286 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15286 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_15286_validation.pdf.gz emd_15286_validation.pdf.gz | 648.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_15286_full_validation.pdf.gz emd_15286_full_validation.pdf.gz | 647.7 KB | 表示 | |
| XML形式データ |  emd_15286_validation.xml.gz emd_15286_validation.xml.gz | 17.4 KB | 表示 | |
| CIF形式データ |  emd_15286_validation.cif.gz emd_15286_validation.cif.gz | 23 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15286 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15286 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15286 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-15286 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8a9uMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15286.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15286.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened cryo-EM map of the full AAV3B-VP1KO particle | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.556 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Reconstruction of the final iteration of the 3D...
| ファイル | emd_15286_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of the final iteration of the 3D auto refinement of the full AAV3B-VP1KO particle | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half-1 map of the 3D auto refinement...
| ファイル | emd_15286_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half-1 map of the 3D auto refinement of the full AAV3B-VP1KO particle | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half-2 map of the 3D auto refinement...
| ファイル | emd_15286_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half-2 map of the 3D auto refinement of the full AAV3B-VP1KO particle | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Adeno-associated virus - 3
| 全体 | 名称:  Adeno-associated virus - 3 (アデノ随伴ウイルス) Adeno-associated virus - 3 (アデノ随伴ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Adeno-associated virus - 3
| 超分子 | 名称: Adeno-associated virus - 3 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: For the recombinant production of the VP1KO virion, we used the Pro10TM cell line. NCBI-ID: 46350 / 生物種: Adeno-associated virus - 3 / Sci species strain: 3B / ウイルスタイプ: VIRION / ウイルス・単離状態: SEROTYPE / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| Host system | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| ウイルス殻 | Shell ID: 1 / 直径: 260.0 Å / T番号(三角分割数): 1 |
-分子 #1: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 1 詳細: For pXR3b-VP1KO, the ATG start codon of VP1 was changed to TGA, and the GAT codon of Asp4 was changed to GAC to remove an alternative ATG. Thus this sample only has VP2 and VP3. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 66.728281 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TAPGKKRPVD QSPQEPDSSS GVGKSGKQPA RKRLNFGQTG DSESVPDPQP LGEPPAAPTS LGSNTMASGG GAPMADNNEG ADGVGNSSG NWHCDSQWLG DRVITTSTRT WALPTYNNHL YKQISSQSGA SNDNHYFGYS TPWGYFDFNR FHCHFSPRDW Q RLINNNWG ...文字列: TAPGKKRPVD QSPQEPDSSS GVGKSGKQPA RKRLNFGQTG DSESVPDPQP LGEPPAAPTS LGSNTMASGG GAPMADNNEG ADGVGNSSG NWHCDSQWLG DRVITTSTRT WALPTYNNHL YKQISSQSGA SNDNHYFGYS TPWGYFDFNR FHCHFSPRDW Q RLINNNWG FRPKKLSFKL FNIQVKEVTQ NDGTTTIANN LTSTVQVFTD SEYQLPYVLG SAHQGCLPPF PADVFMVPQY GY LTLNNGS QAVGRSSFYC LEYFPSQMLR TGNNFQFSYT FEDVPFHSSY AHSQSLDRLM NPLIDQYLYY LNRTQGTTSG TTN QSRLLF SQAGPQSMSL QARNWLPGPC YRQQRLSKTA NDNNNSNFPW TAASKYHLNG RDSLVNPGPA MASHKDDEEK FFPM HGNLI FGKEGTTASN AELDNVMITD EEEIRTTNPV ATEQYGTVAN NLQSSNTAPT TRTVNDQGAL PGMVWQDRDV YLQGP IWAK IPHTDGHFHP SPLMGGFGLK HPPPQIMIKN TPVPANPPTT FSPAKFASFI TQYSTGQVSV EIEWELQKEN SKRWNP EIQ YTSNYNKSVN VDFTVDTNGV YSEPRPIGTR YLTRNL |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 85 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK III 詳細: Parameters: 2 seconds of blot time, -2 offset value, after 45 seconds of incubation. Quantifoil Cu 300-mesh R1.2/1.3 holey-carbon grids with an extra layer of carbon on top added in-house.. |
| 詳細 | VP1KO 4.23E+13 vg/ml |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 実像数: 2384 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 92000 |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)