+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EM map of the LARGE1 dual glycosyltransferase with its coiled-coil stem region | |||||||||
 マップデータ マップデータ | Local filtered map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | matriglycan / xylose / glucuronic acid / polymerase / TRANSFERASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報post-embryonic hindlimb morphogenesis / Defective LARGE causes MDDGA6 and MDDGB6 / xylosyltransferase activity / 転移酵素; グリコシル基を移すもの / walking behavior / : / principal sensory nucleus of trigeminal nerve development / striated muscle cell development / connective tissue development / skeletal muscle organ development ...post-embryonic hindlimb morphogenesis / Defective LARGE causes MDDGA6 and MDDGB6 / xylosyltransferase activity / 転移酵素; グリコシル基を移すもの / walking behavior / : / principal sensory nucleus of trigeminal nerve development / striated muscle cell development / connective tissue development / skeletal muscle organ development / glycosphingolipid biosynthetic process / O-linked glycosylation / glucuronosyltransferase activity / UDP-xylosyltransferase activity / localization of cell / protein O-linked mannosylation / reactive gliosis / glycoprotein biosynthetic process / N-acetylglucosamine metabolic process / neuromuscular process controlling posture / retina layer formation / water transport / plasma membrane organization / acetylglucosaminyltransferase activity / neuromuscular synaptic transmission / retina vasculature development in camera-type eye / basement membrane organization / skeletal muscle fiber differentiation / skeletal muscle tissue regeneration / nerve development / hexosyltransferase activity / dentate gyrus development / protein O-linked glycosylation / astrocyte differentiation / synaptic assembly at neuromuscular junction / cardiac muscle cell development / acetylcholine receptor signaling pathway / protein targeting to membrane / 転移酵素; グリコシル基を移すもの; 六炭糖残基を移すもの / muscle cell cellular homeostasis / glycosyltransferase activity / blood vessel development / protein glycosylation / macrophage differentiation / 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの / response to light stimulus / behavioral fear response / skeletal muscle fiber development / striated muscle contraction / response to mechanical stimulus / potassium ion transmembrane transport / cytoskeleton organization / myelination / post-translational protein modification / protein localization to plasma membrane / determination of adult lifespan / long-term synaptic potentiation / sensory perception of sound / intracellular protein transport / neuron migration / bone development / neuromuscular junction / multicellular organism growth / memory / manganese ion binding / gene expression / protein-containing complex assembly / Golgi membrane / Golgi apparatus / protein-containing complex / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
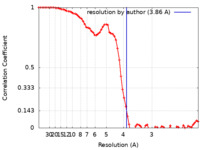
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.86 Å | |||||||||
 データ登録者 データ登録者 | Diskin R / Katz M | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2022 ジャーナル: PLoS One / 年: 2022タイトル: Structural basis for matriglycan synthesis by the LARGE1 dual glycosyltransferase. 著者: Michael Katz / Ron Diskin /  要旨: LARGE1 is a bifunctional glycosyltransferase responsible for generating a long linear polysaccharide termed matriglycan that links the cytoskeleton and the extracellular matrix and is required for ...LARGE1 is a bifunctional glycosyltransferase responsible for generating a long linear polysaccharide termed matriglycan that links the cytoskeleton and the extracellular matrix and is required for proper muscle function. This matriglycan polymer is made with an alternating pattern of xylose and glucuronic acid monomers. Mutations in the LARGE1 gene have been shown to cause life-threatening dystroglycanopathies through the inhibition of matriglycan synthesis. Despite its major role in muscle maintenance, the structure of the LARGE1 enzyme and how it assembles in the Golgi are unknown. Here we present the structure of LARGE1, obtained by a combination of X-ray crystallography and single-particle cryo-EM. We found that LARGE1 homo-dimerizes in a configuration that is dictated by its coiled-coil stem domain. The structure shows that this enzyme has two canonical GT-A folds within each of its catalytic domains. In the context of its dimeric structure, the two types of catalytic domains are brought into close proximity from opposing monomers to allow efficient shuttling of the substrates between the two domains. Together, with putative retention of matriglycan by electrostatic interactions, this dimeric organization offers a possible mechanism for the ability of LARGE1 to synthesize long matriglycan chains. The structural information further reveals the mechanisms in which disease-causing mutations disrupt the activity of LARGE1. Collectively, these data shed light on how matriglycan is synthesized alongside the functional significance of glycosyltransferase oligomerization. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14987.map.gz emd_14987.map.gz | 1.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14987-v30.xml emd-14987-v30.xml emd-14987.xml emd-14987.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14987_fsc.xml emd_14987_fsc.xml | 6.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14987.png emd_14987.png | 60.1 KB | ||
| マスクデータ |  emd_14987_msk_1.map emd_14987_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-14987.cif.gz emd-14987.cif.gz | 4.9 KB | ||
| その他 |  emd_14987_additional_1.map.gz emd_14987_additional_1.map.gz emd_14987_half_map_1.map.gz emd_14987_half_map_1.map.gz emd_14987_half_map_2.map.gz emd_14987_half_map_2.map.gz | 14.9 MB 28.2 MB 28.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14987 http://ftp.pdbj.org/pub/emdb/structures/EMD-14987 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14987 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14987 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_14987_validation.pdf.gz emd_14987_validation.pdf.gz | 665.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_14987_full_validation.pdf.gz emd_14987_full_validation.pdf.gz | 665.1 KB | 表示 | |
| XML形式データ |  emd_14987_validation.xml.gz emd_14987_validation.xml.gz | 14 KB | 表示 | |
| CIF形式データ |  emd_14987_validation.cif.gz emd_14987_validation.cif.gz | 18 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14987 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14987 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14987 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-14987 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7zvjC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14987.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14987.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local filtered map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||
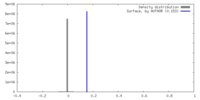
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_14987_msk_1.map emd_14987_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
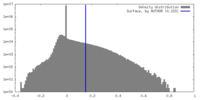
| 密度ヒストグラム |
-追加マップ: Unfiltered map
| ファイル | emd_14987_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
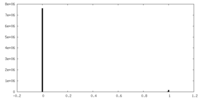
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_14987_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_14987_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
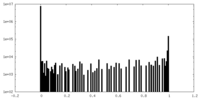
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : LARGE1
| 全体 | 名称: LARGE1 |
|---|---|
| 要素 |
|
-超分子 #1: LARGE1
| 超分子 | 名称: LARGE1 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 180 KDa |
-分子 #1: LARGE1
| 分子 | 名称: LARGE1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: HHHHHHGSGG LFSGSFEDGK PVSLSPLESQ AHSPRYTASS QR ERESLEV RMREVEEENR ALRRQLSLAQ GRAPSHRRGN HSKTYSMEEG TGDSENLRAG IVA GNSSEC GQQPVVEKCE TIHVAIVCAG YNASRDVVTL VKSVLFHRRN PLHFHLIADS IAEQ ILATL ...文字列: HHHHHHGSGG LFSGSFEDGK PVSLSPLESQ AHSPRYTASS QR ERESLEV RMREVEEENR ALRRQLSLAQ GRAPSHRRGN HSKTYSMEEG TGDSENLRAG IVA GNSSEC GQQPVVEKCE TIHVAIVCAG YNASRDVVTL VKSVLFHRRN PLHFHLIADS IAEQ ILATL FQTWMVPAVR VDFYNADELK SEVSWIPNKH YSGIYGLMKL VLTKTLPANL ERVIV LDTD ITFATDIAEL WAVFHKFKGQ QVLGLVENQS DWYLGNLWKN HRPWPALGRG YNTGVI LLL LDKLRKMKWE QMWRLTAERE LMGMLSTSLA DQDIFNAVIK QNPFLVYQLP CFWNVQL SD HTRSEQCYRD VSDLKVIHWN SPKKLRVKNK HVEFFRNLYL TFLEYDGNLL RRELFGCP S EADVNSENLQ KQLSELDEDD LCYEFRRERF TVHRTHLYFL HYEYEPAADS TDVTLVAQL SMDRLQMLEA ICKHWEGPIS LALYLSDAEA QQFLRYAQGS EVLMSRHNVG YHIVYKEGQF YPVNLLRNV AMKHISTPYM FLSDIDFLPM YGLYEYLRKS VIQLDLANTK KAMIVPAFET L RYRLSFPK SKAELLSMLD MGTLFTFRYH VWTKGHAPTN FAKWRTATTP YRVEWEADFE PY VVVRRDC PEYDRRFVGF GWNKVAHIME LDVQEYEFIV LPNAYMIHMP HAPSFDITKF RSN KQYRIC LKTLKEEFQQ DMSRRYGFAA LKYLTAENNS UniProtKB: Xylosyl- and glucuronyltransferase LARGE1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 77.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)