+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13928 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 80S-bound human SKI complex in the closed state | ||||||||||||||||||
 マップデータ マップデータ | porstprocessed_map_hSKI_ribosome-bound_closed | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | multiprotein complex / RNA helicase / DExH-box helicase / ATPase / RNA binding / RNA BINDING PROTEIN | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Ski complex / mRNA decay by 3' to 5' exoribonuclease / Cdc73/Paf1 complex / nuclear-transcribed mRNA catabolic process, 3'-5' exonucleolytic nonsense-mediated decay / negative regulation of myeloid cell differentiation / 3'-5' RNA helicase activity / Association of TriC/CCT with target proteins during biosynthesis / nuclear-transcribed mRNA catabolic process / RNA Polymerase II Transcription Elongation / Formation of RNA Pol II elongation complex ...Ski complex / mRNA decay by 3' to 5' exoribonuclease / Cdc73/Paf1 complex / nuclear-transcribed mRNA catabolic process, 3'-5' exonucleolytic nonsense-mediated decay / negative regulation of myeloid cell differentiation / 3'-5' RNA helicase activity / Association of TriC/CCT with target proteins during biosynthesis / nuclear-transcribed mRNA catabolic process / RNA Polymerase II Transcription Elongation / Formation of RNA Pol II elongation complex / RNA Polymerase II Pre-transcription Events / rescue of stalled ribosome / transcription elongation by RNA polymerase II / euchromatin / Wnt signaling pathway / E3 ubiquitin ligases ubiquitinate target proteins / RNA helicase activity / RNA helicase / ATP hydrolysis activity / RNA binding / nucleoplasm / ATP binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Koegel A / Keidel A | ||||||||||||||||||
| 資金援助 |  ドイツ, 5件 ドイツ, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2022 ジャーナル: Mol Cell / 年: 2022タイトル: The human SKI complex regulates channeling of ribosome-bound RNA to the exosome via an intrinsic gatekeeping mechanism. 著者: Alexander Kögel / Achim Keidel / Fabien Bonneau / Ingmar B Schäfer / Elena Conti /  要旨: The superkiller (SKI) complex is the cytoplasmic co-factor and regulator of the RNA-degrading exosome. In human cells, the SKI complex functions mainly in co-translational surveillance-decay ...The superkiller (SKI) complex is the cytoplasmic co-factor and regulator of the RNA-degrading exosome. In human cells, the SKI complex functions mainly in co-translational surveillance-decay pathways, and its malfunction is linked to a severe congenital disorder, the trichohepatoenteric syndrome. To obtain insights into the molecular mechanisms regulating the human SKI (hSKI) complex, we structurally characterized several of its functional states in the context of 80S ribosomes and substrate RNA. In a prehydrolytic ATP form, the hSKI complex exhibits a closed conformation with an inherent gating system that effectively traps the 80S-bound RNA into the hSKI2 helicase subunit. When active, hSKI switches to an open conformation in which the gating is released and the RNA 3' end exits the helicase. The emerging picture is that the gatekeeping mechanism and architectural remodeling of hSKI underpin a regulated RNA channeling system that is mechanistically conserved among the cytoplasmic and nuclear helicase-exosome complexes. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13928.map.gz emd_13928.map.gz | 13.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13928-v30.xml emd-13928-v30.xml emd-13928.xml emd-13928.xml | 24.5 KB 24.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13928_fsc.xml emd_13928_fsc.xml | 11.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13928.png emd_13928.png | 72.4 KB | ||
| マスクデータ |  emd_13928_msk_1.map emd_13928_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13928.cif.gz emd-13928.cif.gz | 8.2 KB | ||
| その他 |  emd_13928_additional_1.map.gz emd_13928_additional_1.map.gz emd_13928_half_map_1.map.gz emd_13928_half_map_1.map.gz emd_13928_half_map_2.map.gz emd_13928_half_map_2.map.gz | 82 MB 98.3 MB 98.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13928 http://ftp.pdbj.org/pub/emdb/structures/EMD-13928 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13928 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13928 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13928_validation.pdf.gz emd_13928_validation.pdf.gz | 875.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13928_full_validation.pdf.gz emd_13928_full_validation.pdf.gz | 874.9 KB | 表示 | |
| XML形式データ |  emd_13928_validation.xml.gz emd_13928_validation.xml.gz | 18.7 KB | 表示 | |
| CIF形式データ |  emd_13928_validation.cif.gz emd_13928_validation.cif.gz | 24.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13928 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13928 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13928 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13928 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13928.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13928.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | porstprocessed_map_hSKI_ribosome-bound_closed | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8512 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13928_msk_1.map emd_13928_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
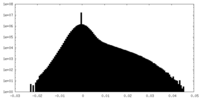
| 密度ヒストグラム |
-追加マップ: postprocessed map hSKI-40S closed
| ファイル | emd_13928_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | postprocessed_map_hSKI-40S_closed | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
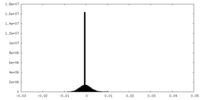
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_13928_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half_map_1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
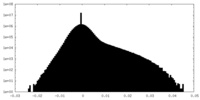
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_13928_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half_map_2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : H.s. SKI complex bound to the ribosome in closed state
| 全体 | 名称: H.s. SKI complex bound to the ribosome in closed state |
|---|---|
| 要素 |
|
-超分子 #1: H.s. SKI complex bound to the ribosome in closed state
| 超分子 | 名称: H.s. SKI complex bound to the ribosome in closed state タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 380 KDa |
-分子 #1: Helicase SKI2W
| 分子 | 名称: Helicase SKI2W / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 137.913688 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MMETERLVLP PPDPLDLPLR AVELGCTGHW ELLNLPGAPE SSLPHGLPPC APDLQQEAEQ LFLSSPAWLP LHGVEHSARK WQRKTDPWS LLAVLGAPVP SDLQAQRHPT TGQILGYKEV LLENTNLSAT TSLSLRRPPG PASQSLWGNP TQYPFWPGGM D EPTITDLN ...文字列: MMETERLVLP PPDPLDLPLR AVELGCTGHW ELLNLPGAPE SSLPHGLPPC APDLQQEAEQ LFLSSPAWLP LHGVEHSARK WQRKTDPWS LLAVLGAPVP SDLQAQRHPT TGQILGYKEV LLENTNLSAT TSLSLRRPPG PASQSLWGNP TQYPFWPGGM D EPTITDLN TREEAEEEID FEKDLLTIPP GFKKGMDFAP KDCPTPAPGL LSLSCMLEPL DLGGGDEDEN EAVGQPGGPR GD TVSASPC SAPLARASSL EDLVLKEAST AVSTPEAPEP PSQEQWAIPV DATSPVGDFY RLIPQPAFQW AFEPDVFQKQ AIL HLERHD SVFVAAHTSA GKTVVAEYAI ALAQKHMTRT IYTSPIKALS NQKFRDFRNT FGDVGLLTGD VQLHPEASCL IMTT EILRS MLYSGSDVIR DLEWVIFDEV HYINDVERGV VWEEVLIMLP DHVSIILLSA TVPNALEFAD WIGRLKRRQI YVIST VTRP VPLEHYLFTG NSSKTQGELF LLLDSRGAFH TKGYYAAVEA KKERMSKHAQ TFGAKQPTHQ GGPAQDRGVY LSLLAS LRT RAQLPVVVFT FSRGRCDEQA SGLTSLDLTT SSEKSEIHLF LQRCLARLRG SDRQLPQVLH MSELLNRGLG VHHSGIL PI LKEIVEMLFS RGLVKVLFAT ETFAMGVNMP ARTVVFDSMR KHDGSTFRDL LPGEYVQMAG RAGRRGLDPT GTVILLCK G RVPEMADLHR MMMGKPSQLQ SQFRLTYTMI LNLLRVDALR VEDMMKRSFS EFPSRKDSKA HEQALAELTK RLGALEEPD MTGQLVDLPE YYSWGEELTE TQHMIQRRIM ESVNGLKSLS AGRVVVVKNQ EHHNALGVIL QVSSNSTSRV FTTLVLCDKP LSQDPQDRG PATAEVPYPD DLVGFKLFLP EGPCDHTVVK LQPGDMAAIT TKVLRVNGEK ILEDFSKRQQ PKFKKDPPLA A VTTAVQEL LRLAQAHPAG PPTLDPVNDL QLKDMSVVEG GLRARKLEEL IQGAQCVHSP RFPAQYLKLR ERMQIQKEME RL RFLLSDQ SLLLLPEYHQ RVEVLRTLGY VDEAGTVKLA GRVACAMSSH ELLLTELMFD NALSTLRPEE IAALLSGLVC QSP GDAGDQ LPNTLKQGIE RVRAVAKRIG EVQVACGLNQ TVEEFVGELN FGLVEVVYEW ARGMPFSELA GLSGTPEGLV VRCI QRLAE MCRSLRGAAR LVGEPVLGAK METAATLLRR DIVFAASLYT Q UniProtKB: Superkiller complex protein 2 |
-分子 #2: Tetratricopeptide repeat protein 37
| 分子 | 名称: Tetratricopeptide repeat protein 37 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 178.651641 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKHHHHHHHH HHSAGLEVLF QGPDSMSSKE VKTALKSARD AIRNKEYKEA LKHCKTVLKQ EKNNYNAWVF IGVAAAELEQ PDQAQSAYK KAAELEPDQL LAWQGLANLY EKYNHINAKD DLPGVYQKLL DLYESVDKQK WCDVCKKLVD LYYQEKKHLE V ARTWHKLI ...文字列: MKHHHHHHHH HHSAGLEVLF QGPDSMSSKE VKTALKSARD AIRNKEYKEA LKHCKTVLKQ EKNNYNAWVF IGVAAAELEQ PDQAQSAYK KAAELEPDQL LAWQGLANLY EKYNHINAKD DLPGVYQKLL DLYESVDKQK WCDVCKKLVD LYYQEKKHLE V ARTWHKLI KTRQEQGAEN EELHQLWRKL TQFLAESTED QNNETQQLLF TAFENALGLS DKIPSEDHQV LYRHFIQSLS KF PHESARL KKACEGMINI YPTVQYPLEV LCLHLIESGN LTDEGQQYCC RLVEMDSKSG PGLIGLGIKA LQDKKYEDAV RNL TEGLKE SPVCTSGWYH LAEAQVKMHR PKEAVLSCSQ ALKIVDNLGA SGNSLYQRNL CLHLKAEALI KLSDYDSSEE AIRT LDQIS DADNIPGLLV LKSLAYRNKG SFDEAAKIME DLLSSYPDLA EVHALEALIH FTKKDYLQAE KCFQRALEKD TEVAE YHYQ LGLTYWFMGE ETRKDKTKAL THFLKAARLD TYMGKVFCYL GHYYRDVVGD KNRARGCYRK AFELDDTDAE SGAAAV DLS VELEDMEMAL AILTTVTQKA SAGTAKWAWL RRGLYYLKAG QHSQAVADLQ AALRADPKDF NCWESLGEAY LSRGGYT TA LKSFTKASEL NPESIYSVFK VAAIQQILGK YKEAVAQYQM IIKKKEDYVP ALKGLGECHL MMAKAALVDY LDGKAVDY I EKALEYFTCA LQHRADVSCL WKLAGDACTC LYAVAPSKVN VHVLGVLLGQ KEGKQVLKKN ELLHLGGRCY GRALKLMST SNTWCDLGIN YYRQAQHLAE TGSNMNDLKE LLEKSLHCLK KAVRLDSNNH LYWNALGVVA CYSGIGNYAL AQHCFIKSIQ SEQINAVAW TNLGVLYLTN ENIEQAHEAF KMAQSLDPSY LMCWIGQALI AEAVGSYDTM DLFRHTTELN MHTEGALGYA Y WVCTTLQD KSNRETELYQ YNILQMNAIP AAQVILNKYV ERIQNYAPAF TMLGYLNEHL QLKKEAANAY QRAILLLQTA ED QDTYNVA IRNYGRLLCS TGEYDKAIQA FKSTPLEVLE DIIGFALALF MKGLYKESSK AYERALSIVE SEQDKAHILT ALA ITEYKQ GKTDVAKTLL FKCSILKEPT TESLQALCAL GLAMQDATLS KAALNELLKH IKHKDSNYQR CLLTSAIYAL QGRS VAVQK QISKAVHSNP GDPALWSLLS RVVAQYAQRN AKGGVVAGNV AHILDSNHGK KALLYTAVNQ LAMGSSSAED EKNTA LKTI QKAALLSPGD PAIWAGLMAA CHADDKLALV NNTQPKRIDL YLALLSAVSA SIKDEKFFEN YNQSLEKWSL SQAVTG LID TGRISEAETL CTKNLKSNPD QPAVILLLRQ VQCKPLLESQ KPLPDAVLEE LQKTVMSNST SVPAWQWLAH VYQSQGM MR AAEMCYRKSL QLASQRGSWS GKLSSLLRLA LLALKVCMAN ISNDHWPSLV QEATTEALKL CFCPLAVLLQ ALLQFKRK M GARETRRLLE RVVYQPGYPK SIASTARWYL LRHLYAKDDY ELIDVLVNNA KTHGDTRALE LNQRLSSQ UniProtKB: Superkiller complex protein 3 |
-分子 #3: WD repeat-containing protein 61
| 分子 | 名称: WD repeat-containing protein 61 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 33.617465 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTNQYGILFK QEQAHDDAIW SVAWGTNKKE NSETVVTGSL DDLVKVWKWR DERLDLQWSL EGHQLGVVSV DISHTLPIAA SSSLDAHIR LWDLENGKQI KSIDAGPVDA WTLAFSPDSQ YLATGTHVGK VNIFGVESGK KEYSLDTRGK FILSIAYSPD G KYLASGAI ...文字列: MTNQYGILFK QEQAHDDAIW SVAWGTNKKE NSETVVTGSL DDLVKVWKWR DERLDLQWSL EGHQLGVVSV DISHTLPIAA SSSLDAHIR LWDLENGKQI KSIDAGPVDA WTLAFSPDSQ YLATGTHVGK VNIFGVESGK KEYSLDTRGK FILSIAYSPD G KYLASGAI DGIINIFDIA TGKLLHTLEG HAMPIRSLTF SPDSQLLVTA SDDGYIKIYD VQHANLAGTL SGHASWVLNV AF CPDDTHF VSSSSDKSVK VWDVGTRTCV HTFFDHQDQV WGVKYNGNGS KIVSVGDDQE IHIYDCPI UniProtKB: Superkiller complex protein 8 |
-分子 #4: RNA (5'-R(P*UP*UP*UP*UP*UP*U)-3')
| 分子 | 名称: RNA (5'-R(P*UP*UP*UP*UP*UP*U)-3') / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.792037 KDa |
| 配列 | 文字列: UUUUUU |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均露光時間: 5.0 sec. / 平均電子線量: 55.8 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE / 当てはまり具合の基準: CORRELATION COEFFICIENT |
|---|---|
| 得られたモデル |  PDB-7qdz: |
 ムービー
ムービー コントローラー
コントローラー































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)