+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single Particle Cryo-EM structure of photosynthetic A8B8 glyceraldehyde-3-phosphate dehydrogenase hexadecamer (major conformer) from Spinacia oleracia. | |||||||||
 マップデータ マップデータ | A8B8 photosynthetic glyceraldehyde 3-phosphate dehydrogenase (GAPDH) hexadecamer - major oligomer. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (NADP+) / グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (NADP+) /  glyceraldehyde-3-phosphate dehydrogenase (NADP+) (phosphorylating) activity / reductive pentose-phosphate cycle / glyceraldehyde-3-phosphate dehydrogenase (NADP+) (phosphorylating) activity / reductive pentose-phosphate cycle /  アポプラスト / glyceraldehyde-3-phosphate dehydrogenase (NAD+) (phosphorylating) activity / アポプラスト / glyceraldehyde-3-phosphate dehydrogenase (NAD+) (phosphorylating) activity /  葉緑体 / glucose metabolic process / NAD binding / 葉緑体 / glucose metabolic process / NAD binding /  NADP binding NADP binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Spinacia oleracea (ホウレンソウ) / Spinacia oleracea (ホウレンソウ) /   spinach (ホウレンソウ) spinach (ホウレンソウ) | |||||||||
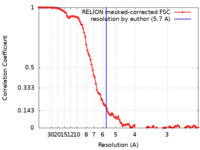
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.7 Å クライオ電子顕微鏡法 / 解像度: 5.7 Å | |||||||||
 データ登録者 データ登録者 | Marotta R / Fermani S / Sparla F / Trost P / Del Giudice A | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Acta Crystallogr D Struct Biol / 年: 2022 ジャーナル: Acta Crystallogr D Struct Biol / 年: 2022タイトル: Unravelling the regulation pathway of photosynthetic AB-GAPDH. 著者: Roberto Marotta / Alessandra Del Giudice / Libero Gurrieri / Silvia Fanti / Paolo Swuec / Luciano Galantini / Giuseppe Falini / Paolo Trost / Simona Fermani / Francesca Sparla /  要旨: Oxygenic phototrophs perform carbon fixation through the Calvin-Benson cycle. Different mechanisms adjust the cycle and the light-harvesting reactions to rapid environmental changes. Photosynthetic ...Oxygenic phototrophs perform carbon fixation through the Calvin-Benson cycle. Different mechanisms adjust the cycle and the light-harvesting reactions to rapid environmental changes. Photosynthetic glyceraldehyde 3-phosphate dehydrogenase (GAPDH) is a key enzyme in the cycle. In land plants, different photosynthetic GAPDHs exist: the most abundant isoform is formed by AB heterotetramers and the least abundant by A homotetramers. Regardless of the subunit composition, GAPDH is the major consumer of photosynthetic NADPH and its activity is strictly regulated. While A-GAPDH is regulated by CP12, AB-GAPDH is autonomously regulated through the C-terminal extension (CTE) of its B subunits. Reversible inhibition of AB-GAPDH occurs via the oxidation of a cysteine pair located in the CTE and the substitution of NADP(H) with NAD(H) in the cofactor-binding site. These combined conditions lead to a change in the oligomerization state and enzyme inhibition. SEC-SAXS and single-particle cryo-EM analysis were applied to reveal the structural basis of this regulatory mechanism. Both approaches revealed that spinach (AB)-GAPDH oligomers with n = 1, 2, 4 and 5 co-exist in a dynamic system. B subunits mediate the contacts between adjacent tetramers in AB and AB oligomers. The CTE of each B subunit penetrates into the active site of a B subunit of the adjacent tetramer, which in turn moves its CTE in the opposite direction, effectively preventing the binding of the substrate 1,3-bisphosphoglycerate in the B subunits. The whole mechanism is made possible, and eventually controlled, by pyridine nucleotides. In fact, NAD(H), by removing NADP(H) from A subunits, allows the entrance of the CTE into the active site of the B subunit, hence stabilizing inhibited oligomers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13826.map.gz emd_13826.map.gz | 95.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13826-v30.xml emd-13826-v30.xml emd-13826.xml emd-13826.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13826_fsc.xml emd_13826_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13826.png emd_13826.png | 92.3 KB | ||
| その他 |  emd_13826_half_map_1.map.gz emd_13826_half_map_1.map.gz emd_13826_half_map_2.map.gz emd_13826_half_map_2.map.gz | 79.4 MB 79.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13826 http://ftp.pdbj.org/pub/emdb/structures/EMD-13826 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13826 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13826 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13826.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13826.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | A8B8 photosynthetic glyceraldehyde 3-phosphate dehydrogenase (GAPDH) hexadecamer - major oligomer. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.21 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: A8B8 photosynthetic glyceraldehyde 3-phosphate dehydrogenase (GAPDH) hexadecamer -...
| ファイル | emd_13826_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | A8B8 photosynthetic glyceraldehyde 3-phosphate dehydrogenase (GAPDH) hexadecamer - major oligomer. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
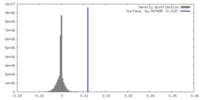
| 密度ヒストグラム |
-ハーフマップ: A8B8 photosynthetic glyceraldehyde 3-phosphate dehydrogenase (GAPDH) hexadecamer -...
| ファイル | emd_13826_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | A8B8 photosynthetic glyceraldehyde 3-phosphate dehydrogenase (GAPDH) hexadecamer - major oligomer. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
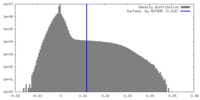
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : A8B8 photosynthetic glyceraldehyde 3-phosphate dehydrogenase hete...
| 全体 | 名称: A8B8 photosynthetic glyceraldehyde 3-phosphate dehydrogenase hetero-hexdecamer (major conformer) complexed with NAD. |
|---|---|
| 要素 |
|
-超分子 #1: A8B8 photosynthetic glyceraldehyde 3-phosphate dehydrogenase hete...
| 超分子 | 名称: A8B8 photosynthetic glyceraldehyde 3-phosphate dehydrogenase hetero-hexdecamer (major conformer) complexed with NAD. タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Spinacia oleracea (ホウレンソウ) Spinacia oleracea (ホウレンソウ) |
-分子 #1: Glyceraldehyde-3-phosphate dehydrogenase B, chloroplastic
| 分子 | 名称: Glyceraldehyde-3-phosphate dehydrogenase B, chloroplastic タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO EC番号:  グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (NADP+) グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (NADP+) |
|---|---|
| 由来(天然) | 生物種:   spinach (ホウレンソウ) spinach (ホウレンソウ) |
| 分子量 | 理論値: 48.183789 KDa |
| 配列 | 文字列: MASHAALAPS RIPASTRLAS KASQQYSFLT QCSFKRLDVA DFSGLRSSNS VTFTREASFH DVIAAQLTTK PTGAAPVRGE TVAKLKVAI NGFGRIGRNF LRCWHGRKDS PLDVVVVNDS GGVKSATHLL KYDSILGTFK ADVKIIDNET FSIDGKPIKV V SNRDPLKL ...文字列: MASHAALAPS RIPASTRLAS KASQQYSFLT QCSFKRLDVA DFSGLRSSNS VTFTREASFH DVIAAQLTTK PTGAAPVRGE TVAKLKVAI NGFGRIGRNF LRCWHGRKDS PLDVVVVNDS GGVKSATHLL KYDSILGTFK ADVKIIDNET FSIDGKPIKV V SNRDPLKL PWAELGIDIV IEGTGVFVDG PGAGKHIQAG AKKVIITAPA KGSDIPTYVV GVNEKDYGHD VANIISNASC TT NCLAPFV KVLDEELGIV KGTMTTTHSY TGDQRLLDAS HRDLRRARAA ALNIVPTSTG AAKAVSLVLP QLKGKLNGIA LRV PTPNVS VVDLVVNIEK VGVTAEDVNN AFRKAAAGPL KGVLDVCDIP LVSVDFRCSD FSSTIDSSLT MVMGGDMVKV VAWY DNEWG YSQRVVDLAD LVANKWPGLE GSVASGDPLE DFCKDNPADE ECKLYE |
-分子 #2: Glyceraldehyde-3-phosphate dehydrogenase A, chloroplastic
| 分子 | 名称: Glyceraldehyde-3-phosphate dehydrogenase A, chloroplastic タイプ: protein_or_peptide / ID: 2 / コピー数: 8 / 光学異性体: LEVO EC番号:  グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (NADP+) グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (NADP+) |
|---|---|
| 由来(天然) | 生物種:   spinach (ホウレンソウ) spinach (ホウレンソウ) |
| 分子量 | 理論値: 43.159098 KDa |
| 配列 | 文字列: MASNMLSIAN PSLRVYNKGF SEFSGLHTSS LPFGRKGSDD LMAFVSFQTN AVGGKRSSQN GVVEAKLKVA INGFGRIGRN FLRCWHGRK DSPLDVVVIN DTGGVKQASH LLKYDSILGT FDADVKTAGD SAISVDGKVI KVVSDRNPVN LPWGDMGIDL V IEGTGVFV ...文字列: MASNMLSIAN PSLRVYNKGF SEFSGLHTSS LPFGRKGSDD LMAFVSFQTN AVGGKRSSQN GVVEAKLKVA INGFGRIGRN FLRCWHGRK DSPLDVVVIN DTGGVKQASH LLKYDSILGT FDADVKTAGD SAISVDGKVI KVVSDRNPVN LPWGDMGIDL V IEGTGVFV DRDGAGKHLQ AGAKKVLITA PGKGDIPTYV VGVNEEGYTH ADTIISNASC TTNCLAPFVK VLDQKFGIIK GT MTTTHSY TGDQRLLDAS HRDLRRARAA CLNIVPTSTG AAKAVALVLP NLKGKLNGIA LRVPTPNVSV VDLVVQVSKK TFA EEVNAA FRESADNELK GILSVCDEPL VSIDFRCTDV SSTIDSSLTM VMGDDMVKVI AWYDNEWGYS QRVVDLADIV ANKW Q(UNK) |
-分子 #3: NICOTINAMIDE-ADENINE-DINUCLEOTIDE
| 分子 | 名称: NICOTINAMIDE-ADENINE-DINUCLEOTIDE / タイプ: ligand / ID: 3 / コピー数: 16 / 式: NAD |
|---|---|
| 分子量 | 理論値: 663.425 Da |
| Chemical component information |  ChemComp-NAD: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 42.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)