+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of double-stranded DNA-bound MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATP (B1 map) | |||||||||||||||
 マップデータ マップデータ | Body 1 of multi-body refinement of MD-(ATP) | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | Helicase / Activation / Kinase / Phosphorylation / REPLICATION | |||||||||||||||
| 生物種 |  | |||||||||||||||
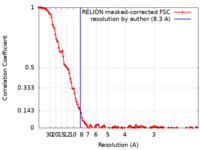
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.3 Å | |||||||||||||||
 データ登録者 データ登録者 | Saleh A / Noguchi Y / Aramayo R / Ivanova ME / Speck C | |||||||||||||||
| 資金援助 |  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: The structural basis of Cdc7-Dbf4 kinase dependent targeting and phosphorylation of the MCM2-7 double hexamer. 著者: Almutasem Saleh / Yasunori Noguchi / Ricardo Aramayo / Marina E Ivanova / Kathryn M Stevens / Alex Montoya / S Sunidhi / Nicolas Lopez Carranza / Marcin J Skwark / Christian Speck /  要旨: The controlled assembly of replication forks is critical for genome stability. The Dbf4-dependent Cdc7 kinase (DDK) initiates replisome assembly by phosphorylating the MCM2-7 replicative helicase at ...The controlled assembly of replication forks is critical for genome stability. The Dbf4-dependent Cdc7 kinase (DDK) initiates replisome assembly by phosphorylating the MCM2-7 replicative helicase at the N-terminal tails of Mcm2, Mcm4 and Mcm6. At present, it remains poorly understood how DDK docks onto the helicase and how the kinase targets distal Mcm subunits for phosphorylation. Using cryo-electron microscopy and biochemical analysis we discovered that an interaction between the HBRCT domain of Dbf4 with Mcm2 serves as an anchoring point, which supports binding of DDK across the MCM2-7 double-hexamer interface and phosphorylation of Mcm4 on the opposite hexamer. Moreover, a rotation of DDK along its anchoring point allows phosphorylation of Mcm2 and Mcm6. In summary, our work provides fundamental insights into DDK structure, control and selective activation of the MCM2-7 helicase during DNA replication. Importantly, these insights can be exploited for development of novel DDK inhibitors. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13657.map.gz emd_13657.map.gz | 138.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13657-v30.xml emd-13657-v30.xml emd-13657.xml emd-13657.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13657_fsc.xml emd_13657_fsc.xml | 13 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13657.png emd_13657.png | 48.8 KB | ||
| マスクデータ |  emd_13657_msk_1.map emd_13657_msk_1.map | 178 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-13657.cif.gz emd-13657.cif.gz | 4.4 KB | ||
| その他 |  emd_13657_additional_1.map.gz emd_13657_additional_1.map.gz | 19 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13657 http://ftp.pdbj.org/pub/emdb/structures/EMD-13657 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13657 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13657 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13657_validation.pdf.gz emd_13657_validation.pdf.gz | 579.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13657_full_validation.pdf.gz emd_13657_full_validation.pdf.gz | 578.9 KB | 表示 | |
| XML形式データ |  emd_13657_validation.xml.gz emd_13657_validation.xml.gz | 13.2 KB | 表示 | |
| CIF形式データ |  emd_13657_validation.cif.gz emd_13657_validation.cif.gz | 17.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13657 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13657 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13657 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13657 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13657.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13657.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Body 1 of multi-body refinement of MD-(ATP) | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.085 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_13657_msk_1.map emd_13657_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
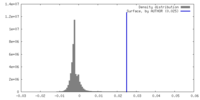
| 密度ヒストグラム |
-追加マップ: Post-processed map of Body 1 of multi-body refinement...
| ファイル | emd_13657_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Post-processed map of Body 1 of multi-body refinement of MD-(ATP) (B-factor of -100 applied) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
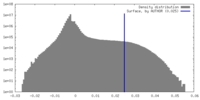
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MCM2-7 double hexamer bound to double-stranded DNA and two copies...
| 全体 | 名称: MCM2-7 double hexamer bound to double-stranded DNA and two copies of Cdc7-Dbf4 |
|---|---|
| 要素 |
|
-超分子 #1: MCM2-7 double hexamer bound to double-stranded DNA and two copies...
| 超分子 | 名称: MCM2-7 double hexamer bound to double-stranded DNA and two copies of Cdc7-Dbf4 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#9 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.54 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: AIR / 詳細: 15 mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 1.5 seconds and blot force +2. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 実像数: 3416 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)