+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13206 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Tetrameric building block of the human GID complex. | |||||||||
 マップデータ マップデータ | Postprocessed map of the four building blocks of the tetrameric hGID complex, consisting of WDR26, RanBP9, TWA1, and MAEA or RMND5A. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
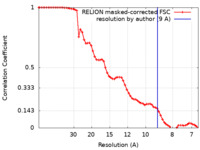
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 9.0 Å | |||||||||
 データ登録者 データ登録者 | Mohamed WI / Park SL / Rabl J / Leitner A / Boehringer D / Peter M | |||||||||
| 資金援助 | European Union,  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: EMBO Rep / 年: 2021 ジャーナル: EMBO Rep / 年: 2021タイトル: The human GID complex engages two independent modules for substrate recruitment. 著者: Weaam I Mohamed / Sophia L Park / Julius Rabl / Alexander Leitner / Daniel Boehringer / Matthias Peter /  要旨: The human GID (hGID) complex is a conserved E3 ubiquitin ligase regulating diverse biological processes, including glucose metabolism and cell cycle progression. However, the biochemical function and ...The human GID (hGID) complex is a conserved E3 ubiquitin ligase regulating diverse biological processes, including glucose metabolism and cell cycle progression. However, the biochemical function and substrate recognition of the multi-subunit complex remain poorly understood. Using biochemical assays, cross-linking mass spectrometry, and cryo-electron microscopy, we show that hGID engages two distinct modules for substrate recruitment, dependent on either WDR26 or GID4. WDR26 and RanBP9 cooperate to ubiquitinate HBP1 in vitro, while GID4 is dispensable for this reaction. In contrast, GID4 functions as an adaptor for the substrate ZMYND19, which surprisingly lacks a Pro/N-end degron. GID4 substrate binding and ligase activity is regulated by ARMC8α, while the shorter ARMC8β isoform assembles into a stable hGID complex that is unable to recruit GID4. Cryo-EM reconstructions of these hGID complexes reveal the localization of WDR26 within a ring-like, tetrameric architecture and suggest that GID4 and WDR26/Gid7 utilize different, non-overlapping binding sites. Together, these data advance our mechanistic understanding of how the hGID complex recruits cognate substrates and provides insights into the regulation of its E3 ligase activity. #1:  ジャーナル: Biorxiv / 年: 2021 ジャーナル: Biorxiv / 年: 2021タイトル: The human GID complex engages two independent modules for substrate recruitment 著者: Mohamed WI / Park SL / Rabl J / Leitner A / Boehringer D / Peter M | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13206.map.gz emd_13206.map.gz | 1.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13206-v30.xml emd-13206-v30.xml emd-13206.xml emd-13206.xml | 27.6 KB 27.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13206_fsc.xml emd_13206_fsc.xml | 6.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13206.png emd_13206.png | 47.7 KB | ||
| マスクデータ |  emd_13206_msk_1.map emd_13206_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| その他 |  emd_13206_additional_1.map.gz emd_13206_additional_1.map.gz emd_13206_half_map_1.map.gz emd_13206_half_map_1.map.gz emd_13206_half_map_2.map.gz emd_13206_half_map_2.map.gz | 17.1 MB 17.1 MB 17.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13206 http://ftp.pdbj.org/pub/emdb/structures/EMD-13206 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13206 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13206 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13206_validation.pdf.gz emd_13206_validation.pdf.gz | 422.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13206_full_validation.pdf.gz emd_13206_full_validation.pdf.gz | 422 KB | 表示 | |
| XML形式データ |  emd_13206_validation.xml.gz emd_13206_validation.xml.gz | 12.4 KB | 表示 | |
| CIF形式データ |  emd_13206_validation.cif.gz emd_13206_validation.cif.gz | 16.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13206 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13206 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13206 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13206 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13206.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13206.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed map of the four building blocks of the tetrameric hGID complex, consisting of WDR26, RanBP9, TWA1, and MAEA or RMND5A. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.36 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
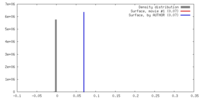
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13206_msk_1.map emd_13206_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
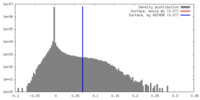
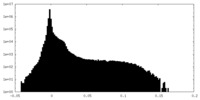
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_13206_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
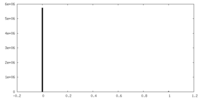
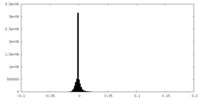
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13206_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
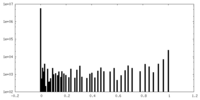
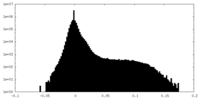
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13206_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
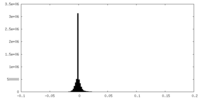
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : hGID
| 全体 | 名称: hGID |
|---|---|
| 要素 |
|
-超分子 #1: hGID
| 超分子 | 名称: hGID / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Complex of subunits WDR26, RanBP9, TWA1, MAEA, and RMND5A. The map contains one subunit of WDR26, RanBP9, TWA1, and one subunit of MAEA or RMND5A. |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  組換株: Sf9 / 組換細胞: Sf9 |
-分子 #1: RanBP9
| 分子 | 名称: RanBP9 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDWSHPQFEK SAVDENLYFQ GGGRMSGQPP PPPPQQQQQQ QQLSPPPPAA LAPVSGVVLP APPAVSAGSS PAGSPGGGAG GEGLGAAAAA LLLHPPPPPP PATAAPPPPP PPPPPPASAA APASGPPAPP GLAAGPGPAG GAPTPALVAG SSAAAPFPHG DSALNEQEKE ...文字列: MDWSHPQFEK SAVDENLYFQ GGGRMSGQPP PPPPQQQQQQ QQLSPPPPAA LAPVSGVVLP APPAVSAGSS PAGSPGGGAG GEGLGAAAAA LLLHPPPPPP PATAAPPPPP PPPPPPASAA APASGPPAPP GLAAGPGPAG GAPTPALVAG SSAAAPFPHG DSALNEQEKE LQRRLKRLYP AVDEQETPLP RSWSPKDKFS YIGLSQNNLR VHYKGHGKTP KDAASVRATH PIPAACGIYY FEVKIVSKGR DGYMGIGLSA QGVNMNRLPG WDKHSYGYHG DDGHSFCSSG TGQPYGPTFT TGDVIGCCVN LINNTCFYTK NGHSLGIAFT DLPPNLYPTV GLQTPGEVVD ANFGQHPFVF DIEDYMREWR TKIQAQIDRF PIGDREGEWQ TMIQKMVSSY LVHHGYCATA EAFARSTDQT VLEELASIKN RQRIQKLVLA GRMGEAIETT QQLYPSLLER NPNLLFTLKV RQFIEMVNGT DSEVRCLGGR SPKSQDSYPV SPRPFSSPSM SPSHGMNIHN LASGKGSTAH FSGFESCSNG VISNKAHQSY CHSNKHQSSN LNVPELNSIN MSRSQQVNNF TSNDVDMETD HYSNGVGETS SNGFLNGSSK HDHEMEDCDT EMEVDSSQLR RQLCGGSQAA IERMIHFGRE LQAMSEQLRR DCGKNTANKK MLKDAFSLLA YSDPWNSPVG NQLDPIQREP VCSALNSAIL ETHNLPKQPP LALAMGQATQ CLGLMARSGI GSCAFATVED YLH |
-分子 #2: WDR26
| 分子 | 名称: WDR26 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SAVDENLYFQ GGGRSQSDED VIRLIGQHLN GLGLNQTVDL LMQESGCRLE HPSATKFRNH VMEGDWDKAE NDLNELKPLV HSPHAIVVRG ALEISQTLLG IIVRMKFLLL QQKYLEYLED GKVLEALQVL RCELTPLKYN TERIHVLSGY LMCSHAEDLR ...文字列: MGSSHHHHHH SAVDENLYFQ GGGRSQSDED VIRLIGQHLN GLGLNQTVDL LMQESGCRLE HPSATKFRNH VMEGDWDKAE NDLNELKPLV HSPHAIVVRG ALEISQTLLG IIVRMKFLLL QQKYLEYLED GKVLEALQVL RCELTPLKYN TERIHVLSGY LMCSHAEDLR AKAEWEGKGT ASRSKLLDKL QTYLPPSVML PPRRLQTLLR QAVELQRDRC LYHNTKLDNN LDSVSLLIDH VCSRRQFPCY TQQILTEHCN EVWFCKFSND GTKLATGSKD TTVIIWQVDP DTHLLKLLKT LEGHAYGVSY IAWSPDDNYL VACGPDDCSE LWLWNVQTGE LRTKMSQSHE DSLTSVAWNP DGKRFVTGGQ RGQFYQCDLD GNLLDSWEGV RVQCLWCLSD GKTVLASDTH QRIRGYNFED LTDRNIVQED HPIMSFTISK NGRLALLNVA TQGVHLWDLQ DRVLVRKYQG VTQGFYTIHS CFGGHNEDFI ASGSEDHKVY IWHKRSELPI AELTGHTRTV NCVSWNPQIP SMMASASDDG TVRIWGPAPF IDHQNIEEEC SSMDS |
-分子 #3: Twa1
| 分子 | 名称: Twa1 / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SAVDENLYFQ GGGRMSYAEK PDEITKDEWM EKLNNLHVQR ADMNRLIMNY LVTEGFKEAA EKFRMESGIE PSVDLETLDE RIKIREMILK GQIQEAIALI NSLHPELLDT NRYLYFHLQQ QHLIELIRQR ETEAALEFAQ TQLAEQGEES RECLTEMERT ...文字列: MGSSHHHHHH SAVDENLYFQ GGGRMSYAEK PDEITKDEWM EKLNNLHVQR ADMNRLIMNY LVTEGFKEAA EKFRMESGIE PSVDLETLDE RIKIREMILK GQIQEAIALI NSLHPELLDT NRYLYFHLQQ QHLIELIRQR ETEAALEFAQ TQLAEQGEES RECLTEMERT LALLAFDSPE ESPFGDLLHT MQRQKVWSEV NQAVLDYENR ESTPKLAKLL KLLLWAQNEL DQKKVKYPKM TDLSKGVIEE PK |
-分子 #4: MAEA
| 分子 | 名称: MAEA / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDYKDDDDKS AVDENLYFQG GGRMAVQESA AQLSMTLKVQ EYPTLKVPYE TLNKRFRAAQ KNIDRETSHV TMVVAELEKT LSGCPAVDSV VSLLDGVVEK LSVLKRKAVE SIQAEDESAK LCKRRIEHLK EHSSDQPAAA SVWKRKRMDR MMVEHLLRCG YYNTAVKLAR ...文字列: MDYKDDDDKS AVDENLYFQG GGRMAVQESA AQLSMTLKVQ EYPTLKVPYE TLNKRFRAAQ KNIDRETSHV TMVVAELEKT LSGCPAVDSV VSLLDGVVEK LSVLKRKAVE SIQAEDESAK LCKRRIEHLK EHSSDQPAAA SVWKRKRMDR MMVEHLLRCG YYNTAVKLAR QSGIEDLVNI EMFLTAKEVE ESLERRETAT CLAWCHDNKS RLRKMKSCLE FSLRIQEFIE LIRQNKRLDA VRHARKHFSQ AEGSQLDEVR QAMGMLAFPP DTHISPYKDL LDPARWRMLI QQFRYDNYRL HQLGNNSVFT LTLQAGLSAI KTPQCYKEDG SSKSPDCPVC SRSLNKLAQP LPMAHCANSR LVCKISGDVM NENNPPMMLP NGYVYGYNSL LSIRQDDKVV CPRTKEVFHF SQAEKVYIM |
-分子 #5: RMND5A
| 分子 | 名称: RMND5A / タイプ: protein_or_peptide / ID: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SAVDENLYFQ GGGRMDQCVT VERELEKVLH KFSGYGQLCE RGLEELIDYT GGLKHEILQS HGQDAELSGT LSLVLTQCCK RIKDTVQKLA SDHKDIHSSV SRVGKAIDKN FDSDISSVGI DGCWQADSQR LLNEVMVEHF FRQGMLDVAE ELCQESGLSV ...文字列: MGSSHHHHHH SAVDENLYFQ GGGRMDQCVT VERELEKVLH KFSGYGQLCE RGLEELIDYT GGLKHEILQS HGQDAELSGT LSLVLTQCCK RIKDTVQKLA SDHKDIHSSV SRVGKAIDKN FDSDISSVGI DGCWQADSQR LLNEVMVEHF FRQGMLDVAE ELCQESGLSV DPSQKEPFVE LNRILEALKV RVLRPALEWA VSNREMLIAQ NSSLEFKLHR LYFISLLMGG TTNQREALQY AKNFQPFALN HQKDIQVLMG SLVYLRQGIE NSPYVHLLDA NQWADICDIF TRDACALLGL SVESPLSVSF SAGCVALPAL INIKAVIEQR QCTGVWNQKD ELPIEVDLGK KCWYHSIFAC PILRQQTTDN NPPMKLVCGH IISRDALNKM FNGSKLKCPY CPMEQSPGDA KQIFF |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: 50 mM HEPES pH 7.4, 200 mM NaCl, 1mM TCEP and 0.01% NP40 | |||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 1.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 詳細: 15mA | |||||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV | |||||||||||||||
| 詳細 | The complex was purified with gel filtration and subsequently crosslinked using the GraFix method. |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 撮影 | Image recording ID: 1 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 2 / 実像数: 26017 / 平均露光時間: 1.5 sec. / 平均電子線量: 78.0 e/Å2 / 詳細: magnification: 105000x, 0.84A/pix |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #1~
電子顕微鏡法 #1~
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TITAN KRIOS |
| 撮影 | Image recording ID: 2 フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 2 / 実像数: 19599 / 平均露光時間: 8.5 sec. / 平均電子線量: 78.0 e/Å2 / 詳細: magnification: 165000, 0.84A/pix |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)