+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1318 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Three-dimensional structure of the respiratory chain supercomplex I1III2IV1 from bovine heart mitochondria. | |||||||||
 マップデータ マップデータ | This is a side view along the mitochondiral membrane plane | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 32.0 Å | |||||||||
 データ登録者 データ登録者 | Schafer E / Dencher NA / Vonck J / Parcej DN | |||||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2007 ジャーナル: Biochemistry / 年: 2007タイトル: Three-dimensional structure of the respiratory chain supercomplex I1III2IV1 from bovine heart mitochondria. 著者: Eva Schäfer / Norbert A Dencher / Janet Vonck / David N Parcej /  要旨: The respiratory chain complexes can arrange into multienzyme assemblies, so-called supercomplexes. We present the first 3D map of a respiratory chain supercomplex. It was determined by random conical ...The respiratory chain complexes can arrange into multienzyme assemblies, so-called supercomplexes. We present the first 3D map of a respiratory chain supercomplex. It was determined by random conical tilt electron microscopy analysis of a bovine supercomplex consisting of complex I, dimeric complex III, and complex IV (I1III2IV1). Within this 3D map the positions and orientations of all the individual complexes in the supercomplex were determined unambiguously. Furthermore, the ubiquinone and cytochrome c binding sites of each complex in the supercomplex could be located. The mobile electron carrier binding site of each complex was found to be in proximity to the binding site of the succeeding complex in the respiratory chain. This provides structural evidence for direct substrate channeling in the supercomplex assembly with short diffusion distances for the mobile electron carriers. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1318.map.gz emd_1318.map.gz | 6.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1318-v30.xml emd-1318-v30.xml emd-1318.xml emd-1318.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1318.gif 1318.gif | 35.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1318 http://ftp.pdbj.org/pub/emdb/structures/EMD-1318 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1318 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1318 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1318_validation.pdf.gz emd_1318_validation.pdf.gz | 212.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1318_full_validation.pdf.gz emd_1318_full_validation.pdf.gz | 211.4 KB | 表示 | |
| XML形式データ |  emd_1318_validation.xml.gz emd_1318_validation.xml.gz | 5.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1318 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1318 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1318 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1318 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1318.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1318.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a side view along the mitochondiral membrane plane | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.667 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
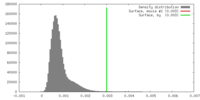
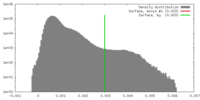
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : bovine supercomplex I1III2IV1
| 全体 | 名称: bovine supercomplex I1III2IV1 |
|---|---|
| 要素 |
|
-超分子 #1000: bovine supercomplex I1III2IV1
| 超分子 | 名称: bovine supercomplex I1III2IV1 / タイプ: sample / ID: 1000 詳細: membranes are solubilised in digitonin, monodisperse sample Number unique components: 3 |
|---|---|
| 分子量 | 実験値: 1.7 MDa / 理論値: 1.7 MDa |
-分子 #1: complex I
| 分子 | 名称: complex I / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #2: complex III
| 分子 | 名称: complex III / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 集合状態: dimer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #3: complex IV
| 分子 | 名称: complex IV / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 集合状態: monomer / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.1 詳細: 0.1 % (w/v) digitonin, 25 mM tricine, 7.5 mM bis-tris, 25 mM aminocaproic acid, 10 % (w/v) glycerol |
| 染色 | タイプ: NEGATIVE / 詳細: 2 % (w/v) ammonium molybdate, deep stain method |
| グリッド | 詳細: 400 mesh Cu grid |
| 凍結 | 凍結剤: NONE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM120T |
|---|---|
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: ZEISS SCAI / 実像数: 20 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 45000 |
| 試料ステージ | 試料ホルダー: n.a / 試料ホルダーモデル: OTHER / Tilt angle max: 50 |
- 画像解析
画像解析
| 詳細 | particles were selected manually |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 32.0 Å / 解像度の算出法: FSC 3 SIGMA CUT-OFF / ソフトウェア - 名称: SPIDER / 詳細: final maps calculated from two / 使用した粒子像数: 1023 |
| 最終 2次元分類 | クラス数: 3 |
-原子モデル構築 1
| 詳細 | The complexes were fitted by hand in Chimera |
|---|
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)