+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-13171 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human Signal Peptidase Complex Paralog A (SPC-A) | |||||||||
 マップデータ マップデータ | Amphipol has been largely masked out | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報signal peptidase complex / signal peptidase I / : / signal peptide processing / protein targeting to ER / Synthesis, secretion, and inactivation of Glucose-dependent Insulinotropic Polypeptide (GIP) / virion assembly / SRP-dependent cotranslational protein targeting to membrane / Synthesis, secretion, and deacylation of Ghrelin / Synthesis, secretion, and inactivation of Glucagon-like Peptide-1 (GLP-1) ...signal peptidase complex / signal peptidase I / : / signal peptide processing / protein targeting to ER / Synthesis, secretion, and inactivation of Glucose-dependent Insulinotropic Polypeptide (GIP) / virion assembly / SRP-dependent cotranslational protein targeting to membrane / Synthesis, secretion, and deacylation of Ghrelin / Synthesis, secretion, and inactivation of Glucagon-like Peptide-1 (GLP-1) / peptidase activity / membrane => GO:0016020 / viral protein processing / serine-type endopeptidase activity / endoplasmic reticulum membrane / endoplasmic reticulum / proteolysis 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
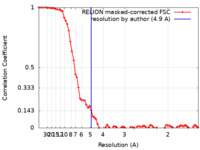
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.9 Å | |||||||||
 データ登録者 データ登録者 | Liaci AM / Foerster F | |||||||||
| 資金援助 |  オランダ, 1件 オランダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2021 ジャーナル: Mol Cell / 年: 2021タイトル: Structure of the human signal peptidase complex reveals the determinants for signal peptide cleavage. 著者: A Manuel Liaci / Barbara Steigenberger / Paulo Cesar Telles de Souza / Sem Tamara / Mariska Gröllers-Mulderij / Patrick Ogrissek / Siewert J Marrink / Richard A Scheltema / Friedrich Förster /    要旨: The signal peptidase complex (SPC) is an essential membrane complex in the endoplasmic reticulum (ER), where it removes signal peptides (SPs) from a large variety of secretory pre-proteins with ...The signal peptidase complex (SPC) is an essential membrane complex in the endoplasmic reticulum (ER), where it removes signal peptides (SPs) from a large variety of secretory pre-proteins with exquisite specificity. Although the determinants of this process have been established empirically, the molecular details of SP recognition and removal remain elusive. Here, we show that the human SPC exists in two functional paralogs with distinct proteolytic subunits. We determined the atomic structures of both paralogs using electron cryo-microscopy and structural proteomics. The active site is formed by a catalytic triad and abuts the ER membrane, where a transmembrane window collectively formed by all subunits locally thins the bilayer. Molecular dynamics simulations indicate that this unique architecture generates specificity for SPs based on the length of their hydrophobic segments. #1:  ジャーナル: BioRxiv / 年: 2020 ジャーナル: BioRxiv / 年: 2020タイトル: Structure of the Human Signal Peptidase Complex Reveals the Determinants for Signal Peptide Cleavage 著者: Liaci AM / Steigenberger B / Tamara S / Telles de Souza PC / Grollers-Mulderij M / Ogrissek P / Marrik SJ / Scheltema RA / Foerster F | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_13171.map.gz emd_13171.map.gz | 3.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-13171-v30.xml emd-13171-v30.xml emd-13171.xml emd-13171.xml | 30.7 KB 30.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_13171_fsc.xml emd_13171_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_13171.png emd_13171.png | 104.7 KB | ||
| マスクデータ |  emd_13171_msk_1.map emd_13171_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_13171_half_map_1.map.gz emd_13171_half_map_1.map.gz emd_13171_half_map_2.map.gz emd_13171_half_map_2.map.gz | 28.3 MB 28.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-13171 http://ftp.pdbj.org/pub/emdb/structures/EMD-13171 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13171 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-13171 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_13171_validation.pdf.gz emd_13171_validation.pdf.gz | 783.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_13171_full_validation.pdf.gz emd_13171_full_validation.pdf.gz | 783.1 KB | 表示 | |
| XML形式データ |  emd_13171_validation.xml.gz emd_13171_validation.xml.gz | 12.7 KB | 表示 | |
| CIF形式データ |  emd_13171_validation.cif.gz emd_13171_validation.cif.gz | 17.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13171 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13171 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13171 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-13171 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_13171.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_13171.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Amphipol has been largely masked out | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.81 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_13171_msk_1.map emd_13171_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_13171_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_13171_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Signal peptidase complex paralog A (SPC-A)
| 全体 | 名称: Signal peptidase complex paralog A (SPC-A) |
|---|---|
| 要素 |
|
-超分子 #1: Signal peptidase complex paralog A (SPC-A)
| 超分子 | 名称: Signal peptidase complex paralog A (SPC-A) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / Organelle: Endoplasmic Reticulum / 細胞中の位置: Endoplasmic Reticulum Homo sapiens (ヒト) / Organelle: Endoplasmic Reticulum / 細胞中の位置: Endoplasmic Reticulum |
| 分子量 | 理論値: 20.3 KDa |
-超分子 #2: Signal peptidase complex catalytic subunit A (SEC11A)
| 超分子 | 名称: Signal peptidase complex catalytic subunit A (SEC11A) タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / Organelle: Endoplasmic Reticulum / 細胞中の位置: Endoplasmic Reticulum Homo sapiens (ヒト) / Organelle: Endoplasmic Reticulum / 細胞中の位置: Endoplasmic Reticulum |
-超分子 #3: Signal peptidase complex subunit 3 (SPC22/23)
| 超分子 | 名称: Signal peptidase complex subunit 3 (SPC22/23) / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / Organelle: Endoplasmic Reticulum / 細胞中の位置: Endoplasmic Reticulum Homo sapiens (ヒト) / Organelle: Endoplasmic Reticulum / 細胞中の位置: Endoplasmic Reticulum |
-超分子 #4: Signal peptidase complex subunit 2 (SPC25)
| 超分子 | 名称: Signal peptidase complex subunit 2 (SPC25) / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / Organelle: Endoplasmic Reticulum / 細胞中の位置: Endoplasmic Reticulum Homo sapiens (ヒト) / Organelle: Endoplasmic Reticulum / 細胞中の位置: Endoplasmic Reticulum |
-超分子 #5: Signal peptidase complex subunit 1 (SPC12)
| 超分子 | 名称: Signal peptidase complex subunit 1 (SPC12) / タイプ: complex / ID: 5 / 親要素: 1 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / Organelle: Endoplasmic Reticulum / 細胞中の位置: Endoplasmic Reticulum Homo sapiens (ヒト) / Organelle: Endoplasmic Reticulum / 細胞中の位置: Endoplasmic Reticulum |
-分子 #1: Signal peptidase complex catalytic subunit SEC11A
| 分子 | 名称: Signal peptidase complex catalytic subunit SEC11A / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: signal peptidase I |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.376088 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: PMLSLDFLDD VRRMNKRQLY YQVLNFGMIV SSALMIWKGL MVITGSESPI VVVLSGSMEP AFHRGDLLFL TNRVEDPIRV GEIVVFRIE GREIPIVHRV LKIHEKQNGH IKFLTKGDNN AVDDRGLYKQ GQHWLEKKDV VGRARGFVPY IGIVTILMND Y PKFKYAVL ...文字列: PMLSLDFLDD VRRMNKRQLY YQVLNFGMIV SSALMIWKGL MVITGSESPI VVVLSGSMEP AFHRGDLLFL TNRVEDPIRV GEIVVFRIE GREIPIVHRV LKIHEKQNGH IKFLTKGDNN AVDDRGLYKQ GQHWLEKKDV VGRARGFVPY IGIVTILMND Y PKFKYAVL FLLGLFVLVH REGGSPGGSG GGSAENLYFQ |
-分子 #2: Signal peptidase complex subunit 3
| 分子 | 名称: Signal peptidase complex subunit 3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.348457 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: PMNTVLSRAN SLFAFSLSVM AALTFGCFIT TAFKDRSVPV RLHVSRIMLK NVEDFTGPRE RSDLGFITFD ITADLENIFD WNVKQLFLY LSAEYSTKNN ALNQVVLWDK IVLRGDNPKL LLKDMKTKYF FFDDGNGLKG NRNVTLTLSW NVVPNAGILP L VTGSGHVS ...文字列: PMNTVLSRAN SLFAFSLSVM AALTFGCFIT TAFKDRSVPV RLHVSRIMLK NVEDFTGPRE RSDLGFITFD ITADLENIFD WNVKQLFLY LSAEYSTKNN ALNQVVLWDK IVLRGDNPKL LLKDMKTKYF FFDDGNGLKG NRNVTLTLSW NVVPNAGILP L VTGSGHVS VPFPDTYEIT KSYGSGEGRG SLLTCGDVEE NPG |
-分子 #3: Signal peptidase complex subunit 2
| 分子 | 名称: Signal peptidase complex subunit 2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 27.215236 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAAAAVQGGR SGGSGGCSGA GGASNCGTGS GRSGLLDKWK IDDKPVKIDK WDGSAVKNSL DDSAKKVLLE KYKYVENFGL IDGRLTICT ISCFFAIVAL IWDYMHPFPE SKPVLALCVI SYFVMMGILT IYTSYKEKSI FLVAHRKDPT GMDPDDIWQL S SSLKRFDD ...文字列: MAAAAVQGGR SGGSGGCSGA GGASNCGTGS GRSGLLDKWK IDDKPVKIDK WDGSAVKNSL DDSAKKVLLE KYKYVENFGL IDGRLTICT ISCFFAIVAL IWDYMHPFPE SKPVLALCVI SYFVMMGILT IYTSYKEKSI FLVAHRKDPT GMDPDDIWQL S SSLKRFDD KYTLKLTFIS GRTKQQREAE FTKSIAKFFD HSGTLVMDAY EPEISRLHDS LAIERKIKGS GQCTNYALLK LA GDVESNP G |
-分子 #4: Signal peptidase complex subunit 1
| 分子 | 名称: Signal peptidase complex subunit 1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO EC番号: 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 20.488445 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: PMARGGDTGC TGPSETSASG AAAIALPGLE GPATDAQCQT LPLTVLKSRS PSPRSLPPAL SCPPPQPAML EHLSSLPTQM DYKGQKLAE QMFQGIILFS AIVGFIYGYV AEQFGWTVYI VMAGFAFSCL LTLPPWPIYR RHPLKWLPVQ ESSTDDKKPG E RKIKRHAK ...文字列: PMARGGDTGC TGPSETSASG AAAIALPGLE GPATDAQCQT LPLTVLKSRS PSPRSLPPAL SCPPPQPAML EHLSSLPTQM DYKGQKLAE QMFQGIILFS AIVGFIYGYV AEQFGWTVYI VMAGFAFSCL LTLPPWPIYR RHPLKWLPVQ ESSTDDKKPG E RKIKRHAK NNGSGATNFS LLKQAGDVEE NPG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製 #1
試料調製 #1
| Preparation ID | 1 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 濃度 | 0.5 mg/mL | |||||||||||||||
| 緩衝液 | pH: 7.8 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - Film type ID: 1 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot for 4s with blot force 0 before plunging.. | |||||||||||||||
| 詳細 | grid condition 1 (frozen without fluorinated fos-choline-8) |
- 試料調製 #2
試料調製 #2
| Preparation ID | 2 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 濃度 | 4 mg/mL | |||||||||||||||
| 緩衝液 | pH: 7.8 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - Film type ID: 1 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot for 4s with blot force 0 before plunging.. | |||||||||||||||
| 詳細 | grid condition 2 (frozen with 1.5 mM fluorinated fos-choline-8) |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 温度 | 最低: 93.0 K / 最高: 93.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 詳細 | 200 kV Talos Arctica at Utrecht University, the Netherlands. Same settings were used for both grid conditions. |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #0 - 検出モード: COUNTING / #0 - 撮影したグリッド数: 1 / #0 - 実像数: 5203 / #0 - 平均露光時間: 9.0 sec. / #0 - 平均電子線量: 63.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #1 - 検出モード: COUNTING / #1 - デジタル化 - サイズ - 横: 3838 pixel / #1 - デジタル化 - サイズ - 縦: 3710 pixel / #1 - 撮影したグリッド数: 1 / #1 - 実像数: 2083 / #1 - 平均露光時間: 10.2 sec. / #1 - 平均電子線量: 63.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 30.0 µm / 最大 デフォーカス(補正後): 4.8 µm / 最小 デフォーカス(補正後): 0.3 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
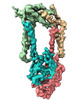
| 得られたモデル |  PDB-7p2p: |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X