+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mouse RNF213:UBE2L3 transthiolation intermediate, chemically stabilized | |||||||||||||||
 マップデータ マップデータ | full reconstructed density | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lipid ubiquitination / lipid droplet formation / negative regulation of non-canonical Wnt signaling pathway / cell cycle phase transition / ubiquitin-protein transferase activator activity / xenophagy / Antigen processing: Ubiquitination & Proteasome degradation / protein K11-linked ubiquitination / 転移酵素; アシル基を移すもの; アミノアシル基を移すもの / cellular response to glucocorticoid stimulus ...lipid ubiquitination / lipid droplet formation / negative regulation of non-canonical Wnt signaling pathway / cell cycle phase transition / ubiquitin-protein transferase activator activity / xenophagy / Antigen processing: Ubiquitination & Proteasome degradation / protein K11-linked ubiquitination / 転移酵素; アシル基を移すもの; アミノアシル基を移すもの / cellular response to glucocorticoid stimulus / sprouting angiogenesis / positive regulation of ubiquitin-protein transferase activity / positive regulation of protein targeting to mitochondrion / E2 ubiquitin-conjugating enzyme / cellular response to steroid hormone stimulus / immune system process / ubiquitin conjugating enzyme activity / protein K63-linked ubiquitination / regulation of lipid metabolic process / protein autoubiquitination / ubiquitin ligase complex / lipid droplet / Synthesis of active ubiquitin: roles of E1 and E2 enzymes / positive regulation of protein ubiquitination / Regulation of TNFR1 signaling / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / RING-type E3 ubiquitin transferase / protein modification process / Regulation of necroptotic cell death / protein polyubiquitination / ubiquitin-protein transferase activity / ubiquitin protein ligase activity / Antigen processing: Ubiquitination & Proteasome degradation / presynapse / E3 ubiquitin ligases ubiquitinate target proteins / ubiquitin-dependent protein catabolic process / angiogenesis / cell population proliferation / transcription coactivator activity / protein ubiquitination / defense response to bacterium / ubiquitin protein ligase binding / regulation of DNA-templated transcription / nucleolus / enzyme binding / ATP hydrolysis activity / RNA binding / nucleoplasm / ATP binding / membrane / nucleus / metal ion binding / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
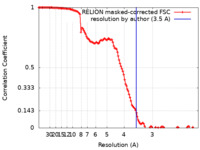
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||||||||
 データ登録者 データ登録者 | Ahel J / Clausen T | |||||||||||||||
| 資金援助 |  オーストリア, オーストリア,  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: E3 ubiquitin ligase RNF213 employs a non-canonical zinc finger active site and is allosterically regulated by ATP 著者: Ahel J / Fletcher AJ / Grabarczyk D / Roitinger E / Deszcz L / Lehner A / Virdee S / Clausen T | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12931.map.gz emd_12931.map.gz | 131.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12931-v30.xml emd-12931-v30.xml emd-12931.xml emd-12931.xml | 26.5 KB 26.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12931_fsc.xml emd_12931_fsc.xml | 12.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12931.png emd_12931.png | 188.7 KB | ||
| マスクデータ |  emd_12931_msk_1.map emd_12931_msk_1.map | 166.4 MB |  マスクマップ マスクマップ | |
| その他 |  emd_12931_half_map_1.map.gz emd_12931_half_map_1.map.gz emd_12931_half_map_2.map.gz emd_12931_half_map_2.map.gz | 131.7 MB 131.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12931 http://ftp.pdbj.org/pub/emdb/structures/EMD-12931 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12931 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12931 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_12931_validation.pdf.gz emd_12931_validation.pdf.gz | 765.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12931_full_validation.pdf.gz emd_12931_full_validation.pdf.gz | 765.4 KB | 表示 | |
| XML形式データ |  emd_12931_validation.xml.gz emd_12931_validation.xml.gz | 19.8 KB | 表示 | |
| CIF形式データ |  emd_12931_validation.cif.gz emd_12931_validation.cif.gz | 26.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12931 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12931 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12931 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12931 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7oikMC  7oimC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
| 電子顕微鏡画像生データ |  EMPIAR-10711 (タイトル: Transmission electron micrographs of mouse RNF213 in a chemically-stabilized complex with human UBE2L3 EMPIAR-10711 (タイトル: Transmission electron micrographs of mouse RNF213 in a chemically-stabilized complex with human UBE2L3Data size: 2.1 TB Data #1: unaligned multi-frame micrographs [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12931.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12931.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | full reconstructed density | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.072 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_12931_msk_1.map emd_12931_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: unfiltered half map 2 of the full reconstructed density
| ファイル | emd_12931_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unfiltered half map 2 of the full reconstructed density | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: unfiltered half map 1 of the full reconstructed density
| ファイル | emd_12931_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unfiltered half map 1 of the full reconstructed density | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RNF213-ABP(UBE2L3-Ub)
| 全体 | 名称: RNF213-ABP(UBE2L3-Ub) |
|---|---|
| 要素 |
|
-超分子 #1: RNF213-ABP(UBE2L3-Ub)
| 超分子 | 名称: RNF213-ABP(UBE2L3-Ub) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: chemically stabilized complex between mouse RNF213 and a modified human UBE2L3 ABP (activity based probe), coupled to modified human ubiquitin |
|---|---|
| 分子量 | 理論値: 580 KDa |
-超分子 #2: RNF213-ABP(UBE2L3-Ub)
| 超分子 | 名称: RNF213-ABP(UBE2L3-Ub) / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 / 詳細: E3 ubiquitin-protein ligase RNF213 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
-超分子 #3: RNF213-ABP(UBE2L3-Ub)
| 超分子 | 名称: RNF213-ABP(UBE2L3-Ub) / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 / 詳細: Ubiquitin-conjugating enzyme E2 L3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
-分子 #1: E3 ubiquitin-protein ligase RNF213
| 分子 | 名称: E3 ubiquitin-protein ligase RNF213 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: RING-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 586.5455 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MECPQCGHVS SEKAPKFCSE CGQKLPSAAT VQGDLKNDNT LVVSSTPEGK TEQGAVLREE EVLLSSTDPG KELEKPEESD SNASWTTQM SKKEKRRRKR QGTISSSEAP SSGLWSLDMP PSPGSHNSAL PQNQAQQGGA ASQPGHPLDT ENMPMEDGFV H TEGSGSPL ...文字列: MECPQCGHVS SEKAPKFCSE CGQKLPSAAT VQGDLKNDNT LVVSSTPEGK TEQGAVLREE EVLLSSTDPG KELEKPEESD SNASWTTQM SKKEKRRRKR QGTISSSEAP SSGLWSLDMP PSPGSHNSAL PQNQAQQGGA ASQPGHPLDT ENMPMEDGFV H TEGSGSPL QGQAAERTDA QSNLAPSDLA EVKDLNTSKP SVDKGLPLDG GPALSAFKGH PKMTDASQKA PLPESKGETS GQ EKKVPPI DAAASPVKTA GKETGEDVRK PKPSPVSPVA SKHGDQEAEL KGKLATPVRK SNEGGNTQPE DQRKPGEGRN FAA AVKTQQ AAAPQQAAAP EPTSAFNPRD TVTVYFHAIV SRHFGFNPEE HKVYVRGGEG LGQKGWTDAC EMYCTQDLHD LGSL VEGKM DIPRQSLDKP IPYKYVIHRG GSSKDTVEYE FIYEQAQKKG EHVNRCLRVV STSLGNGDWH QYDDIICMRS TGFFQ QAKN RILDSTRKEL LKGKKQAAVV MLDRIFSVLQ PWSDINLQSF MTQFLQFYSV VREPMIHDGR ARKWTSLQYE EKEVWT NLW EHVKKQMAPF LEGKSGESLP ADCPVRSKLT LGLSILFMVE AAEFTVPKKD LDSLCYLLIP SAGSPEALHS DLSPVLR IR QRWRIYLTNL CLRCIDERCD RWLGILPLLH TCMQKSPPKK NSKSQPEDTW AGLEGISFSE FRDKAPTRSQ PLQFMQSK M ALLRVDEYLF RSWLSVVPLE SLSSYLENSI DYLSDVPVRV LDCLQGISYR LPGLRKISNQ NMKKDVENVF KMLMHLVDI YQHRIFGENL LQIYLTECLT LHETVCNITA NHQFFEIPAL SAELICKLLE LSPPGHTDEG LPEKSYEDLV TSTLQEALAT TRNWLRSLF KSRMLSISSA YVRLTYSEEM AVWRRLVEIG FPEKHGWKGS LLGDMEGRLK QEPPRLQISF FCSSQCRDGG L HDSVSRSF EKCVIEAVSS ACQSQTSVLE GLSCQDLQKF GTLLSAVITK SWPVHNGEPV FDVDEIFKYL LKWPDVRQLF EL CGTNEKI IDNITEEGRQ LMATAESVFQ KVAGELENGT IVVGQLELIL EHQSQFLDIW NLNRRRLPSQ EKACDVRSLL KRR RDDLLF LKQEKRYVES LLRQLGRVKH LVQVDFGNIE IIHSQDLSNK KLNEAVIKLP NSSSYKRETH YCLSPDIREM ASKL DSLKD SHIFQDFWQE TAESLNTLDK DPRELKVSLP EVLEYLYNPC YDNFYTLYEN LKSGKITFAE VDAIFKDFVD KYDEL KNDL KFMCTMNPQD QKGWISERVG QIKEYHTLHQ AVSSAKVILQ VRRALGVTGD FSVLNPLLNF ADSFEDFGNE KLDQIS PQF IKAKQLLQDI SEPRQRCLEE LARQTELVAW LHKALEDINE LKVFVDLASI SAGENDIDVD RVACFHDAVQ GYASLLY KM DERTNFSDFM NHLQELWRAL DNDQHLPDKL KDSARNLEWL KTVKESHGSV ELSSLSLATA INSRGVYVIE APKDGQKI S PDTVLRLLLP DGHGYPEALR TYSTEELKEL LNKLMLMSGK KDHNSNTEVE KFSEVFSNMQ RLVHVFIKLH CAGNMLFRT WTAKVYCCPD GGIFMNFGLE LLSQLTEKGD VIQLLGALCR QMEDFLDNWK TVVAQKRAEH FYLNFYTAEQ LVYLSSELRK PRPSEAALM MLSFIKGKCT VQDLVQATSA CESKADRYCL REVMKKLPQQ LLSEPSLMGK LQVIMMQSLV YMSAFLPHCL D LDALGRCL AHLATMGGTP VERPLPKGLQ AGQPNLILCG HSEVLPAALA IYMQAPRQPL PTFDEVLLCT PATTIEEVEL LL RRCLTSG SQGHKVYSLL FADQLSYEVG CQAEEFFQSL CTRAHREDYQ LVILCDAARE HCYIPSTFSQ YKVPLVPQAP LPN IQAYLQ SHYQVPKRLL SAATVFRDGL CVGIVTSERA GVGKSLYVNT LHTKLKAKLR DETVPLKIIR LTEPHLDENQ VLSA LLPFL KEKYQKMPVI FHIDISTSVQ TGIPIFLFKL LILQYLMDIN GKIWRRSPGH LYLVEIPQGL SVQPKRSSKL NARAP LFKF LDLFPKVTCR PPKEVIDMEL TPERSHTDPA MDPVEFCSEA FQRPYQYLKR FHQQQNLDTF QYEKGSVEGS PEECLQ HFL IYCGLINPSW SELRNFAWFL NCQLKDCEAS IFCKSAFTGD TLRGFKNFVV TFMILMARDF ATPTLHTSDQ SPGRQSV TI GEVVEEDLAP FSLRKRWESE PHPYVFFNGD HMTMTFIGFH LETNNNGYVD AINPSNGKVI KKDVMTKELF DGLRLQRV P FNIDFDNLPR YEKLERLCLA LGIEWPIDPD ETYELTTDNM LKILAIEMRF RCGIPVIIMG ETGCGKTRLI KFLSDLKRG SVEAETMKLV KVHGGTTPSM IYSKVKEAER TAFSNKAQHK LDTILFFDEA NTTEAVSCIK EILCDRTVDG EHLHEDSGLH IIAACNPYR KHSQEMILRL ESAGLGYRVS AEETADRLGS IPLRQLVYRV HALPPSLIPL VWDFGQLNDS AEKLYIQQIV Q RLVDSVSV NPSETCVIAD VLSASQMFMR KRENECGFVS LRDVERCVKV FRWFHDHSDM LLKELDKFLH ESSDSTHTFE RD PVLWSLV MAIGVCYHAS LEEKASYRTA IARCFPKPYN SSRAILDEVT HVQDLFLRGA PIRTNIARNL ALKENVFMMV ICI ELKIPL FLVGKPGSSK SLAKIIVADA MQGQAAFSEL FRCLKQVHLV SFQCSPHSTP QGIISTFKQC ARFQQGKDLG QYVS VVVLD EVGLAEDSPK MPLKTLHPLL EDGCIEDDPA PYKKVGFVGI SNWALDPAKM NRGIFVSRGS PNEKELIESA EGICS SDRL VQDKIRGYFA PFAKAYETVC QKQDKEFFGL RDYYSLIKMV FAKAKASKRG LSPQDITHAV LRNFSGKDNI QALSIF TAS LPEARYKEEV STVELIKQNI YPGPQASSRG LDGAESRYLL VLTRNYVALQ ILQQTFFEGQ QPEIIFGSSF PQDQEYT QI CRNINRVKIC METGKMVVLL NLQNLYESLY DALNQYYVYL GGQKYVDLGL GTHRVKCRVH TAFRLIVIEE KDVVYKQF P VPLINRLEKH YLDMNTVLQP WQKSIVQELQ QWAHEFADVK ADQFIARHKY SPADVFIGYH SDACASVVLQ AVERQGCRD LTEELYRKVS EEARSILLDC ATPDAVVRLS GSSLGSFTAK QLSQEYYYAQ QHNSFVDFLQ AHLRMTHHEC RAVFTEITTF SRLLTGNDC DVLASELRGL ASKPVVLSLQ QYDTEYSFLK DVRSWLTNPG KRKVLVIQAD FDDGTRSAQL VASAKYTAIN E INKTQGTK DFVFVYFVTK LSRMGSGTSY VGFHGGLWRS VHIDDLRRST IMASDVTKLQ NVTISQLFKP EDKPEQEEME IE TSQSKEL AEEQMEVEDS EEMKKASDPR SCDCSQFLDT TRLVQSCVQG AVGMLRDQNE SCARNMRRVT ILLDLLNEDN TRN ASFLRE SKMRLHVLLN KQEENQVRSL KEWVTREAAN QDALQEAGTF RHTLWKRVQD VVTPILASMI AHIDRDGNLE LLAQ PDSPA WVQDLWMFIY SDIKFLNISL VLNNTRSNSE MSFILVQSHM NLLKDAYNAV PFSWRIRDYL EELWVQAQYI TDTEG LSKK FVEIFQKTPL GVFLAQFPVA QQQKLLQSYL KDFLLLTMKV SSREELMFLQ MALWSCLREL QEASGTPDET YKFPLS LPW VHLAFQHFRT RLQNFSRILT IHPQVLSSLS QAAEKHSLAG CEMTLDAFAA MACAEMLKGD LLKPSPKAWL QLVKNLS TP LELVCSEGYL CDSGSMTRSV IQEVRALWNR IFSIALFVEH VLLGTESHIP ELSPLVTTYV SLLDKCLEED SNLKTCRP F VAVMTTLCDC KDKASKKFSR FGIQPCFICH GDAQDPVCLP CDHVYCLRCI QTWLIPGQMM CPYCLTDLPD KFSPTVSQD HRKAIEKHAQ FRHMCNSFFV DLVSTMCFKD NTPPEKSVID TLLSLLFVQK ELLRDASQKH REHTKSLSPF DDVVDQTPVI RSVLLKLLL KYSFHEVKDY IQNYLTQLEK KAFLTEDKTE LYLLFISCLE DSVHQKTSAG CRNLEQVLRE EGHFLRTYSP G LQGQEPVR IASVEYLQEV ARVRLCLDLA ADFLSELQEG SELAEDKRRF LKHVEEFCTR VNNDWHRVYL VRKLSSQRGM EF VQSFSKQ GHPCQWVFPR KVIAQQKDHV SLMDRYLVHG NEYKAVRDAT AKAVLECKTL DIGNALMACR SPKPQQTAYL LLA LYTEVA ALYRSPNGSL HPEAKQLEAV NKFIKESKIL SDPNIRCFAR SLVDNTLPLL KIRSANSILK GTVTEMAVHV ATIL LCGHN QILKPLRNLA FYPVNMANAF LPTMPEDLLV HARTWRGLEN VTWYTCPRGH PCSVGECGRP MQESTCLDCG LPVGG LNHT PHEGFSAIRN NEDRTQTGHV LGSPQSSGVA EVSDRGQSPV VFILTRLLTH LAMLVGATHN PQALTVIIKP WVQDPQ GFL QQHIQRDLEQ LTKMLGRSAD ETIHVVHLIL SSLLRVQSHG VLNFNAELST KGCRNNWEKH FETLLLRELK HLDKNLP AI NALISQDERI SSNPVTKIIY GDPATFLPHL PQKSIIHCSK IWSCRRKITV EYLQHIVEQK NGKETVPVLW HFLQKEAE L RLVKFLPEIL ALQRDLVKQF QNVSRVEYSS IRGFIHSHSS DGLRKLLHDR ITIFLSTWNA LRRSLETNGE IKLPKDYCC SDLDLDAEFE VILPRRQGLG LCGTALVSYL ISLHNNMVYT VQKFSNEDNS YSVDISEVAD LHVISYEVER DLNPLILSNC QYQVQQGGE TSQEFDLEKI QRQISSRFLQ GKPRLTLKGI PTLVYRRDWN YEHLFMDIKN KMAQSSLPNL AISTISGQLQ S YSDACEAL SIIEITLGFL STAGGDPGMD LNVYIEEVLR MCDQTAQVLK AFSRCQLRHI IALWQFLSAH KSEQRLRLNK EL FREIDVQ YKEELSTQHQ RLLGTFLNEA GLDAFLLELH EMIVLKLKGP RAANSFNPNW SLKDTLVSYM ETKDSDILSE VES QFPEEI LMSSCISVWK IAATRKWDRQ SRGGGHHHHH HHHHH |
-分子 #2: Ubiquitin-conjugating enzyme E2 L3
| 分子 | 名称: Ubiquitin-conjugating enzyme E2 L3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: E2 ubiquitin-conjugating enzyme |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 19.197908 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GWSHPQFEKP GSMAASRRLM KELEEIRKSG MKNFRNIQVD EANLLTWQGL IVPDNPPYDK GAFRIEINFP AEYPFKPPKI TFKTKIYHP NIDEKGQVCL PVISAENWKP ATKTDQVIQS LIALVNDPQP EHPLRADLAE EYSKDRKKFS KNAEEFTKKY G EKRPVD |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 / 構成要素:
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R2/2 / 材質: GOLD / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||
| 凍結 | 凍結剤: ETHANE / 装置: LEICA PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 4591 / 平均露光時間: 3.92 sec. / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.5 µm / 最小 デフォーカス(公称値): -0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X