+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Trimer of dimers TrwK/VirB4unbound C1 at 4.14A - Refinement without symmetry of TrwK/VirB4unbound trimer of dimers complex (with Hcp1) from the R388 type IV secretion system. | |||||||||||||||
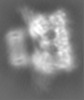
 マップデータ マップデータ | TrwK/VirB4unbound trimer of dimer with HCP without symmetry sharpened map | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |   Salmonella dublin (サルモネラ菌) / Salmonella dublin (サルモネラ菌) /   Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) | |||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.1 Å クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||||||||
 データ登録者 データ登録者 | Vadakkepat AK / Mace K / Lukoyanova N / Waksman G | |||||||||||||||
| 資金援助 |  英国, 4件 英国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: Cryo-EM structure of a type IV secretion system. 著者: Kévin Macé / Abhinav K Vadakkepat / Adam Redzej / Natalya Lukoyanova / Clasien Oomen / Nathalie Braun / Marta Ukleja / Fang Lu / Tiago R D Costa / Elena V Orlova / David Baker / Qian Cong / ...著者: Kévin Macé / Abhinav K Vadakkepat / Adam Redzej / Natalya Lukoyanova / Clasien Oomen / Nathalie Braun / Marta Ukleja / Fang Lu / Tiago R D Costa / Elena V Orlova / David Baker / Qian Cong / Gabriel Waksman /     要旨: Bacterial conjugation is the fundamental process of unidirectional transfer of DNAs, often plasmid DNAs, from a donor cell to a recipient cell. It is the primary means by which antibiotic resistance ...Bacterial conjugation is the fundamental process of unidirectional transfer of DNAs, often plasmid DNAs, from a donor cell to a recipient cell. It is the primary means by which antibiotic resistance genes spread among bacterial populations. In Gram-negative bacteria, conjugation is mediated by a large transport apparatus-the conjugative type IV secretion system (T4SS)-produced by the donor cell and embedded in both its outer and inner membranes. The T4SS also elaborates a long extracellular filament-the conjugative pilus-that is essential for DNA transfer. Here we present a high-resolution cryo-electron microscopy (cryo-EM) structure of a 2.8 megadalton T4SS complex composed of 92 polypeptides representing 8 of the 10 essential T4SS components involved in pilus biogenesis. We added the two remaining components to the structural model using co-evolution analysis of protein interfaces, to enable the reconstitution of the entire system including the pilus. This structure describes the exceptionally large protein-protein interaction network required to assemble the many components that constitute a T4SS and provides insights on the unique mechanism by which they elaborate pili. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
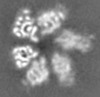
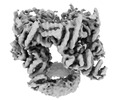
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12716.map.gz emd_12716.map.gz | 23.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12716-v30.xml emd-12716-v30.xml emd-12716.xml emd-12716.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_12716.png emd_12716.png | 95.4 KB | ||
| その他 |  emd_12716_additional_1.map.gz emd_12716_additional_1.map.gz emd_12716_half_map_1.map.gz emd_12716_half_map_1.map.gz emd_12716_half_map_2.map.gz emd_12716_half_map_2.map.gz | 22.6 MB 95.6 MB 95.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12716 http://ftp.pdbj.org/pub/emdb/structures/EMD-12716 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12716 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12716 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7o42MC  7o3jC  7o3tC  7o3vC  7o41C  7o43C  7oiuC  7q1vC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12716.map.gz / 形式: CCP4 / 大きさ: 25 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12716.map.gz / 形式: CCP4 / 大きさ: 25 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | TrwK/VirB4unbound trimer of dimer with HCP without symmetry sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.048 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: TrwK/VirB4unbound trimer of dimer with HCP without symmetry...
| ファイル | emd_12716_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
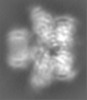
| 注釈 | TrwK/VirB4unbound trimer of dimer with HCP without symmetry unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
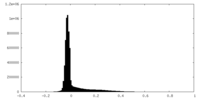
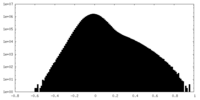
| 密度ヒストグラム |
-ハーフマップ: TrwK/VirB4unbound trimer of dimer - Half-A
| ファイル | emd_12716_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
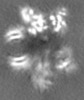
| 注釈 | TrwK/VirB4unbound trimer of dimer - Half-A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
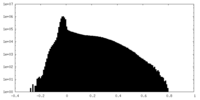
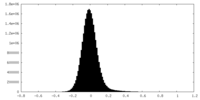
| 密度ヒストグラム |
-ハーフマップ: TrwK/VirB4unbound trimer of dimer - Half-A
| ファイル | emd_12716_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
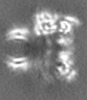
| 注釈 | TrwK/VirB4unbound trimer of dimer - Half-A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
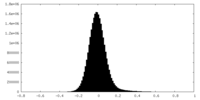
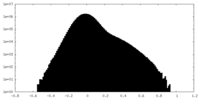
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hexameric complex of Apo-TrwK/VirB4 with Hcp1
| 全体 | 名称: Hexameric complex of Apo-TrwK/VirB4 with Hcp1 |
|---|---|
| 要素 |
|
-超分子 #1: Hexameric complex of Apo-TrwK/VirB4 with Hcp1
| 超分子 | 名称: Hexameric complex of Apo-TrwK/VirB4 with Hcp1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Salmonella dublin (サルモネラ菌) Salmonella dublin (サルモネラ菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌)組換プラスミド: IBA3C:trwM/virB3-trwK/virB4-L6-hcp1C-Strep |
-分子 #1: TrwK protein,Protein hcp1
| 分子 | 名称: TrwK protein,Protein hcp1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌) Pseudomonas aeruginosa (strain ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1) (緑膿菌)株: ATCC 15692 / DSM 22644 / CIP 104116 / JCM 14847 / LMG 12228 / 1C / PRS 101 / PAO1 |
| 分子量 | 理論値: 111.487844 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MGAIESRKLL ASETPVGQFI PYSHHVTDTI ISTKNAEYLS VWKIDGRSHQ SASEADVFQW IRELNNTLRG ISSANLSLWT HIVRRRVYE YPDAEFDNVF CRQLDEKYRE SFTGYNLMVN DLYLTVVYRP VSDKVLSFFA KRERETPDQK KHRQESCIKA L EDINRTLG ...文字列: MGAIESRKLL ASETPVGQFI PYSHHVTDTI ISTKNAEYLS VWKIDGRSHQ SASEADVFQW IRELNNTLRG ISSANLSLWT HIVRRRVYE YPDAEFDNVF CRQLDEKYRE SFTGYNLMVN DLYLTVVYRP VSDKVLSFFA KRERETPDQK KHRQESCIKA L EDINRTLG QSFKRYGAEL LSVYEKGGHA FSAPLEFLAR LVNGEHIPMP ICRDRFSDYM AVNRPMFSKW GEVGELRSLT GL RRFGMLE IREYDDATEP GQLNVLLESD YEFVLTHSFS VLSRPAAKEY LQRHQKNLID ARDVATDQIE EIDEALNQLI SGH FVMGEH HCTLTVYGET VQQVRDNLAH ASAAMLDVAV LPKPVDLALE AGYWAQLPAN WQWRPRPAPI TSLNFLSFSP FHNF MSGKP TGNPWGPAVT ILKTVSGTPL YFNFHASKEE EDATDKRLLG NTMLIGQSSS GKTVLLGFLL AQAQKFKPTI VAFDK DRGM EISIRAMGGR YLPLKTGEPS GFNPFQLPPT HANLIFLKQF VKKLAAAGGE VTHRDEEEID QAITAMMSDS IDKSLR RLS LLLQFLPNPR SDDMDARPTV HARLVKWCEG GDYGWLFDNP TDALDLSTHQ IYGFDITEFL DNPEARTPVM MYLLYRT ES MIDGRRFMYV FDEFWKPLQD EYFEDLAKNK QKTIRKQNGI FVFATQEPSD ALESNIAKTL IQQCATYIFL ANPKADYE D YTQGFKLTDS EFELVRGLGE FSRRFLIKQG DQSALAEMNL GKFRTIVDGE TVERDFDDEL LVLSGTPDNA EIAESIIAE VGDDPAVWLP IFLDRVKAER SDVGSGSGSA VDMFIKIGDV KGESKDKTHA EEIDVLAWSW GMSQSGSMHM GGGGGAGKVN VQDLSFTKY IDKSTPNLMM ACSSGKHYPQ AKLTIRKAGG ENQVEYLIIT LKEVLVSSVS TGGSGGEDRL TENVTLNFAQ V QVDYQPQK ADGAKDGGPV KYGWNIRQNV QA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 49.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: CryoSPARC ab-initio |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: OTHER / 詳細: Stochastic gradient descent (SGD) |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 214048 |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X