+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12234 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | C. thermophilum Pyruvate Dehydrogenase Complex Core from native cell extracts | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) | |||||||||
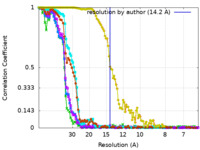
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 14.2 Å | |||||||||
 データ登録者 データ登録者 | Kyrilis FL / Semchonok DA / Skalidis I / Tueting C / Hamdi F / O'Reilly FJ / Rappsilber J / Kastritis PL | |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2021 ジャーナル: Cell Rep / 年: 2021タイトル: Integrative structure of a 10-megadalton eukaryotic pyruvate dehydrogenase complex from native cell extracts. 著者: Fotis L Kyrilis / Dmitry A Semchonok / Ioannis Skalidis / Christian Tüting / Farzad Hamdi / Francis J O'Reilly / Juri Rappsilber / Panagiotis L Kastritis /   要旨: The pyruvate dehydrogenase complex (PDHc) is a giant enzymatic assembly involved in pyruvate oxidation. PDHc components have been characterized in isolation, but the complex's quaternary structure ...The pyruvate dehydrogenase complex (PDHc) is a giant enzymatic assembly involved in pyruvate oxidation. PDHc components have been characterized in isolation, but the complex's quaternary structure has remained elusive due to sheer size, heterogeneity, and plasticity. Here, we identify fully assembled Chaetomium thermophilum α-keto acid dehydrogenase complexes in native cell extracts and characterize their domain arrangements utilizing mass spectrometry, activity assays, crosslinking, electron microscopy (EM), and computational modeling. We report the cryo-EM structure of the PDHc core and observe unique features of the previously unknown native state. The asymmetric reconstruction of the 10-MDa PDHc resolves spatial proximity of its components, agrees with stoichiometric data (60 E2p:12 E3BP:∼20 E1p: ≤ 12 E3), and proposes a minimum reaction path among component enzymes. The PDHc shows the presence of a dynamic pyruvate oxidation compartment, organized by core and peripheral protein species. Our data provide a framework for further understanding PDHc and α-keto acid dehydrogenase complex structure and function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
-検証レポート
| 文書・要旨 |  emd_12234_validation.pdf.gz emd_12234_validation.pdf.gz | 261.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_12234_full_validation.pdf.gz emd_12234_full_validation.pdf.gz | 260.6 KB | 表示 | |
| XML形式データ |  emd_12234_validation.xml.gz emd_12234_validation.xml.gz | 14.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12234 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12234 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12234 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-12234 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12234.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12234.map.gz / 形式: CCP4 / 大きさ: 59.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.03 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
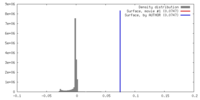
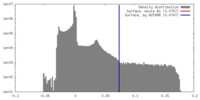
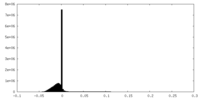
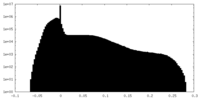
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
+追加マップ: #18
+追加マップ: #6
+追加マップ: #5
+追加マップ: #8
+追加マップ: #7
+追加マップ: #10
+追加マップ: #9
+追加マップ: #2
+追加マップ: #1
+追加マップ: #13
+追加マップ: #12
+追加マップ: #11
+追加マップ: #15
+追加マップ: #14
+追加マップ: #17
+追加マップ: #16
+追加マップ: #4
+追加マップ: #3
+ハーフマップ: #2
+ハーフマップ: #1
- 試料の構成要素
試料の構成要素
-全体 : Native core of the Pyruvate Dehydrogenase Complex from C. thermophilum
| 全体 | 名称: Native core of the Pyruvate Dehydrogenase Complex from C. thermophilum |
|---|---|
| 要素 |
|
-超分子 #1: Native core of the Pyruvate Dehydrogenase Complex from C. thermophilum
| 超分子 | 名称: Native core of the Pyruvate Dehydrogenase Complex from C. thermophilum タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) |
| 分子量 | 理論値: 3 MDa |
-分子 #1: Dihydrolipoyl transacylase, Pyruvate Dehydrogenase Complex E2 com...
| 分子 | 名称: Dihydrolipoyl transacylase, Pyruvate Dehydrogenase Complex E2 component タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: dihydrolipoyllysine-residue acetyltransferase |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) Chaetomium thermophilum var. thermophilum DSM 1495 (菌類) |
| 配列 | 文字列: MLAQVLRRQA LQHVRLARAA APSLTRWYAS YPPHTIVKMP ALSPTMTSGN IGAWQKKPGD AITPGEVLVE IETDKAQMDF EFQEEGVLAK ILKETGEKDV AVGSPIAVLV EEGTDINAFQ NFTLEDAGGD AAAPAAPAKE ELAKAETAPT PASTSAPEPE ETTSTGKLEP ...文字列: MLAQVLRRQA LQHVRLARAA APSLTRWYAS YPPHTIVKMP ALSPTMTSGN IGAWQKKPGD AITPGEVLVE IETDKAQMDF EFQEEGVLAK ILKETGEKDV AVGSPIAVLV EEGTDINAFQ NFTLEDAGGD AAAPAAPAKE ELAKAETAPT PASTSAPEPE ETTSTGKLEP ALDREPNVSF AAKKLAHELD VPLKALKGTG PGGKITEEDV KKAASAPAAA AAAPGAAYQD IPISNMRKTI ATRLKESVSE NPHFFVTSEL SVSKLLKLRQ ALNSSAEGRY KLSVNDFLIK AIAVACKRVP AVNSSWRDGV IRQFDTVDVS VAVATPTGLI TPIVKGVEAK GLETISATVK ELAKKARDGK LKPEDYQGGT ISISNMGMNP AVERFTAIIN PPQAAILAVG TTKKVAVPVE NEDGTTGVEW DDQIVVTASF DHKVVDGAVG AEWMRELKKV VENPLELLL |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 濃度: 200.0 mM / 構成要素 - 式: NH4CH2COOH / 構成要素 - 名称: Ammonium acetate |
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Acetate |
| グリッド | モデル: C-flat / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 10.0 nm / 前処理 - タイプ: GLOW DISCHARGE |
| 詳細 | fractionated native cell extract |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 温度 | 最低: 77.15 K / 最高: 120.0 K |
| 詳細 | Coma Free kept better than 200nm in EPU |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 14.0 µm / 撮影したグリッド数: 1 / 実像数: 1926 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 34739 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 36000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)