+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12228 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Red alga C.merolae Photosystem I 光化学系I 光化学系I | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 チラコイド / チラコイド /  photosynthesis, light harvesting in photosystem I / photosynthesis, light harvesting in photosystem I /  photosystem I reaction center / photosystem I reaction center /  光化学系I / 光化学系I /  光化学系I / photosynthetic electron transport in photosystem I / 光化学系I / photosynthetic electron transport in photosystem I /  色素体 / 色素体 /  chlorophyll binding / chloroplast thylakoid membrane / response to light stimulus ... chlorophyll binding / chloroplast thylakoid membrane / response to light stimulus ... チラコイド / チラコイド /  photosynthesis, light harvesting in photosystem I / photosynthesis, light harvesting in photosystem I /  photosystem I reaction center / photosystem I reaction center /  光化学系I / 光化学系I /  光化学系I / photosynthetic electron transport in photosystem I / 光化学系I / photosynthetic electron transport in photosystem I /  色素体 / 色素体 /  chlorophyll binding / chloroplast thylakoid membrane / response to light stimulus / chlorophyll binding / chloroplast thylakoid membrane / response to light stimulus /  光合成 / 光合成 /  葉緑体 / 4 iron, 4 sulfur cluster binding / membrane => GO:0016020 / 葉緑体 / 4 iron, 4 sulfur cluster binding / membrane => GO:0016020 /  electron transfer activity / electron transfer activity /  oxidoreductase activity / magnesium ion binding / oxidoreductase activity / magnesium ion binding /  生体膜 / 生体膜 /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Cyanidioschyzon merolae strain 10D (シアニディオシゾン) / Cyanidioschyzon merolae strain 10D (シアニディオシゾン) /   Red alga (シアニディオシゾン) Red alga (シアニディオシゾン) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Nelson N / Klaiman D / Hippler M | |||||||||
| 資金援助 |  イスラエル, 1件 イスラエル, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Red alga C.merolae Photosystem I 著者: Nelson N / Klaiman D / Hippler M | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12228.map.gz emd_12228.map.gz | 96.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12228-v30.xml emd-12228-v30.xml emd-12228.xml emd-12228.xml | 34.8 KB 34.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12228_fsc.xml emd_12228_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12228.png emd_12228.png | 253.4 KB | ||
| その他 |  emd_12228_half_map_1.map.gz emd_12228_half_map_1.map.gz emd_12228_half_map_2.map.gz emd_12228_half_map_2.map.gz | 80.8 MB 80.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12228 http://ftp.pdbj.org/pub/emdb/structures/EMD-12228 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12228 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12228 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12228.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12228.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.052 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #2
| ファイル | emd_12228_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
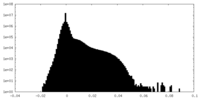
| 投影像・断面図 |
| ||||||||||||
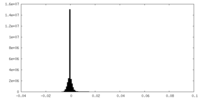
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_12228_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
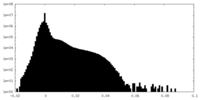
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Photosystem I
+超分子 #1: Photosystem I
+分子 #1: Similar to light harvesting protein
+分子 #2: Similar to chlorophyll a/b-binding protein, CP24
+分子 #3: Lhcr3
+分子 #4: Photosystem I P700 chlorophyll a apoprotein A1
+分子 #5: Photosystem I P700 chlorophyll a apoprotein A2
+分子 #6: Photosystem I iron-sulfur center
+分子 #7: Photosystem I p700 chlorophyll A apoprotein A2
+分子 #8: Photosystem I iron-sulfur center subunit VII
+分子 #9: PSI-F
+分子 #10: Photosystem I reaction center subunit VIII
+分子 #11: Photosystem I reaction center subunit IX
+分子 #12: PSI-K
+分子 #13: Photosystem I reaction center subunit XI
+分子 #14: Photosystem I reaction center subunit XII
+分子 #15: PsaO
+分子 #16: CHLOROPHYLL A
+分子 #17: (1~{S})-3,5,5-trimethyl-4-[(1~{E},3~{E},5~{E},7~{E},9~{E},11~{E},...
+分子 #18: (3R)-beta,beta-caroten-3-ol
+分子 #19: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #20: ERGOSTEROL
+分子 #21: BETA-CAROTENE
+分子 #22: (1S)-2-{[{[(2R)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-...
+分子 #23: DIACYL GLYCEROL
+分子 #24: DODECYL-ALPHA-D-MALTOSIDE
+分子 #25: PHOSPHATIDYLETHANOLAMINE
+分子 #26: CHLOROPHYLL A ISOMER
+分子 #27: PHYLLOQUINONE
+分子 #28: IRON/SULFUR CLUSTER
+分子 #29: 1,2-DIACYL-GLYCEROL-3-SN-PHOSPHATE
+分子 #30: Phosphatidylinositol
+分子 #31: DIGALACTOSYL DIACYL GLYCEROL (DGDG)
+分子 #32: CALCIUM ION
+分子 #33: water
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 7220 / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7blz: |
 ムービー
ムービー コントローラー
コントローラー





















 Z
Z Y
Y X
X