登録情報 データベース : EMDB / ID : EMD-11872タイトル Cryo-EM structure of Arabidopsis thaliana Complex-I (membrane core) 複合体 : Arabidopsis complex I - membrane coreリガンド : x 9種 / 機能・相同性 分子機能 ドメイン・相同性 構成要素
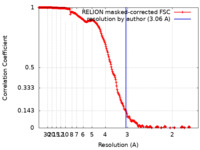
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Arabidopsis thaliana (シロイヌナズナ)手法 / / 解像度 : 3.06 Å Klusch N / Kuehlbrandt W 資金援助 Organization Grant number 国 Max Planck Society
ジャーナル : Plant Cell / 年 : 2021タイトル : A ferredoxin bridge connects the two arms of plant mitochondrial complex I.著者 : Niklas Klusch / Jennifer Senkler / Özkan Yildiz / Werner Kühlbrandt / Hans-Peter Braun / 要旨 : Mitochondrial complex I is the main site for electron transfer to the respiratory chain and generates much of the proton gradient across the inner mitochondrial membrane. Complex I is composed of two ... Mitochondrial complex I is the main site for electron transfer to the respiratory chain and generates much of the proton gradient across the inner mitochondrial membrane. Complex I is composed of two arms, which form a conserved L-shape. We report the structures of the intact, 47-subunit mitochondrial complex I from Arabidopsis thaliana and the 51-subunit complex I from the green alga Polytomella sp., both at around 2.9 Å resolution. In both complexes, a heterotrimeric γ-carbonic anhydrase domain is attached to the membrane arm on the matrix side. Two states are resolved in A. thaliana complex I, with different angles between the two arms and different conformations of the ND1 (NADH dehydrogenase subunit 1) loop near the quinol binding site. The angle appears to depend on a bridge domain, which links the peripheral arm to the membrane arm and includes an unusual ferredoxin. We propose that the bridge domain participates in regulating the activity of plant complex I. 履歴 登録 2020年10月22日 - ヘッダ(付随情報) 公開 2021年12月8日 - マップ公開 2021年12月8日 - 更新 2024年11月13日 - 現状 2024年11月13日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 ドイツ, 1件
ドイツ, 1件  引用
引用 ジャーナル: Plant Cell / 年: 2021
ジャーナル: Plant Cell / 年: 2021
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_11872.map.gz
emd_11872.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-11872-v30.xml
emd-11872-v30.xml emd-11872.xml
emd-11872.xml EMDBヘッダ
EMDBヘッダ emd_11872_fsc.xml
emd_11872_fsc.xml FSCデータファイル
FSCデータファイル emd_11872.png
emd_11872.png emd-11872.cif.gz
emd-11872.cif.gz emd_11872_half_map_1.map.gz
emd_11872_half_map_1.map.gz emd_11872_half_map_2.map.gz
emd_11872_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-11872
http://ftp.pdbj.org/pub/emdb/structures/EMD-11872 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11872
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11872 emd_11872_validation.pdf.gz
emd_11872_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_11872_full_validation.pdf.gz
emd_11872_full_validation.pdf.gz emd_11872_validation.xml.gz
emd_11872_validation.xml.gz emd_11872_validation.cif.gz
emd_11872_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11872
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11872 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11872
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11872 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_11872.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_11872.map.gz / 形式: CCP4 / 大きさ: 824 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー









































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)