登録情報 データベース : EMDB / ID : EMD-11104タイトル Bacillus subtilis RNA polymerase HelD complex 1 複合体 : Recycling complex of Bacillus subitilis RNAP with HelD機能・相同性 分子機能 ドメイン・相同性 構成要素
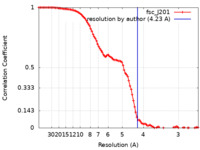
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Bacillus subtilis (枯草菌)手法 / / 解像度 : 4.23 Å Pei H / Hilal T / Huang Y / Said N / Loll B / Wahl MC 資金援助 Organization Grant number 国 German Research Foundation (DFG) HA 2549/15-2 German Research Foundation (DFG) RTG 2473-1 German Research Foundation (DFG) WA 1126/11-1 National Institutes of Health/National Center for Complementary and Integrative Health (NIH/NCCIH) GM067153
ジャーナル : Nat Commun / 年 : 2020タイトル : The δ subunit and NTPase HelD institute a two-pronged mechanism for RNA polymerase recycling.著者 : Hao-Hong Pei / Tarek Hilal / Zhuo A Chen / Yong-Heng Huang / Yuan Gao / Nelly Said / Bernhard Loll / Juri Rappsilber / Georgiy A Belogurov / Irina Artsimovitch / Markus C Wahl / 要旨 : Cellular RNA polymerases (RNAPs) can become trapped on DNA or RNA, threatening genome stability and limiting free enzyme pools, but how RNAP recycling into active states is achieved remains elusive. ... Cellular RNA polymerases (RNAPs) can become trapped on DNA or RNA, threatening genome stability and limiting free enzyme pools, but how RNAP recycling into active states is achieved remains elusive. In Bacillus subtilis, the RNAP δ subunit and NTPase HelD have been implicated in RNAP recycling. We structurally analyzed Bacillus subtilis RNAP-δ-HelD complexes. HelD has two long arms: a Gre cleavage factor-like coiled-coil inserts deep into the RNAP secondary channel, dismantling the active site and displacing RNA, while a unique helical protrusion inserts into the main channel, prying the β and β' subunits apart and, aided by δ, dislodging DNA. RNAP is recycled when, after releasing trapped nucleic acids, HelD dissociates from the enzyme in an ATP-dependent manner. HelD abundance during slow growth and a dimeric (RNAP-δ-HelD) structure that resembles hibernating eukaryotic RNAP I suggest that HelD might also modulate active enzyme pools in response to cellular cues. 履歴 登録 2020年5月29日 - ヘッダ(付随情報) 公開 2020年10月14日 - マップ公開 2020年10月14日 - 更新 2021年4月28日 - 現状 2021年4月28日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 ドイツ,
ドイツ,  米国, 4件
米国, 4件  引用
引用 ジャーナル: Nat Commun / 年: 2020
ジャーナル: Nat Commun / 年: 2020



 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_11104.map.gz
emd_11104.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-11104-v30.xml
emd-11104-v30.xml emd-11104.xml
emd-11104.xml EMDBヘッダ
EMDBヘッダ emd_11104_fsc.xml
emd_11104_fsc.xml FSCデータファイル
FSCデータファイル emd_11104.png
emd_11104.png http://ftp.pdbj.org/pub/emdb/structures/EMD-11104
http://ftp.pdbj.org/pub/emdb/structures/EMD-11104 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11104
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11104 emd_11104_validation.pdf.gz
emd_11104_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_11104_full_validation.pdf.gz
emd_11104_full_validation.pdf.gz emd_11104_validation.xml.gz
emd_11104_validation.xml.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11104
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11104 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11104
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-11104 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_11104.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_11104.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)