+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11069 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Smooth muscle myosin shutdown state heads region | ||||||||||||
 マップデータ マップデータ | Smooth muscle myosin shutdown heads region map | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | myosin / motor / inhibited state / shutdown state / smooth muscle / CONTRACTILE PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報myosin filament / actomyosin structure organization / myosin II complex / structural constituent of muscle / microfilament motor activity / myofibril / supramolecular fiber organization / ADP binding / actin filament binding / calmodulin binding ...myosin filament / actomyosin structure organization / myosin II complex / structural constituent of muscle / microfilament motor activity / myofibril / supramolecular fiber organization / ADP binding / actin filament binding / calmodulin binding / calcium ion binding / ATP binding / cytosol 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.3 Å | ||||||||||||
 データ登録者 データ登録者 | Scarff CA / Carrington G / Casas Mao D / Chalovich JM / Knight PJ / Ranson NA / Peckham M | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2020 ジャーナル: Nature / 年: 2020タイトル: Structure of the shutdown state of myosin-2. 著者: Charlotte A Scarff / Glenn Carrington / David Casas-Mao / Joseph M Chalovich / Peter J Knight / Neil A Ranson / Michelle Peckham /   要旨: Myosin-2 is essential for processes as diverse as cell division and muscle contraction. Dephosphorylation of its regulatory light chain promotes an inactive, 'shutdown' state with the filament- ...Myosin-2 is essential for processes as diverse as cell division and muscle contraction. Dephosphorylation of its regulatory light chain promotes an inactive, 'shutdown' state with the filament-forming tail folded onto the two heads, which prevents filament formation and inactivates the motors. The mechanism by which this happens is unclear. Here we report a cryo-electron microscopy structure of shutdown smooth muscle myosin with a resolution of 6 Å in the head region. A pseudo-atomic model, obtained by flexible fitting of crystal structures into the density and molecular dynamics simulations, describes interaction interfaces at the atomic level. The N-terminal extension of one regulatory light chain interacts with the tail, and the other with the partner head, revealing how the regulatory light chains stabilize the shutdown state in different ways and how their phosphorylation would allow myosin activation. Additional interactions between the three segments of the coiled coil, the motor domains and the light chains stabilize the shutdown molecule. The structure of the lever in each head is competent to generate force upon activation. This shutdown structure is relevant to all isoforms of myosin-2 and provides a framework for understanding their disease-causing mutations. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11069.map.gz emd_11069.map.gz | 6.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11069-v30.xml emd-11069-v30.xml emd-11069.xml emd-11069.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
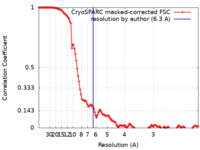
| FSC (解像度算出) |  emd_11069_fsc.xml emd_11069_fsc.xml | 13.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11069.png emd_11069.png | 31.3 KB | ||
| Filedesc metadata |  emd-11069.cif.gz emd-11069.cif.gz | 7.1 KB | ||
| その他 |  emd_11069_half_map_1.map.gz emd_11069_half_map_1.map.gz emd_11069_half_map_2.map.gz emd_11069_half_map_2.map.gz | 154.6 MB 154.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11069 http://ftp.pdbj.org/pub/emdb/structures/EMD-11069 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11069 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11069 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11069.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11069.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Smooth muscle myosin shutdown heads region map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Smooth muscle myosin shutdown heads region half map A
| ファイル | emd_11069_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Smooth muscle myosin shutdown heads region half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
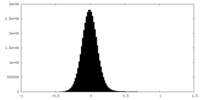
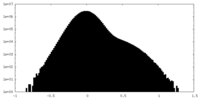
| 密度ヒストグラム |
-ハーフマップ: Smooth muscle myosin shutdown heads region half map B
| ファイル | emd_11069_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Smooth muscle myosin shutdown heads region half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
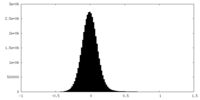
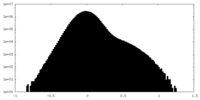
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Smooth muscle myosin in the shutdown state
| 全体 | 名称: Smooth muscle myosin in the shutdown state |
|---|---|
| 要素 |
|
-超分子 #1: Smooth muscle myosin in the shutdown state
| 超分子 | 名称: Smooth muscle myosin in the shutdown state / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Myosin heavy chain 11
| 分子 | 名称: Myosin heavy chain 11 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 229.14825 KDa |
| 配列 | 文字列: MSQKPLSDDE KFLFVDKNFV NNPLAQADWS AKKLVWVPSE KHGFEAASIK EEKGDEVTVE LQENGKKVTL SKDDIQKMNP PKFSKVEDM AELTCLNEAS VLHNLRERYF SGLIYTYSGL FCVVVNPYKQ LPIYSEKIID MYKGKKRHEM PPHIYAIADT A YRSMLQDR ...文字列: MSQKPLSDDE KFLFVDKNFV NNPLAQADWS AKKLVWVPSE KHGFEAASIK EEKGDEVTVE LQENGKKVTL SKDDIQKMNP PKFSKVEDM AELTCLNEAS VLHNLRERYF SGLIYTYSGL FCVVVNPYKQ LPIYSEKIID MYKGKKRHEM PPHIYAIADT A YRSMLQDR EDQSILCTGE SGAGKTENTK KVIQYLAVVA SSHKGKKDTS ITQGPSFSYG ELEKQLLQAN PILEAFGNAK TV KNDNSSR FGKFIRINFD VTGYIVGANI ETYLLEKSRA IRQAKDERTF HIFYYLIAGA SEQMRNDLLL EGFNNYTFLS NGH VPIPAQ QDDEMFQETL EAMRIMGFTE EEQTSILRVV SSVLQLGNIV FKKERNTDQA SMPDNTAAQK VCHLMGINVT DFTR SILTP RIKVGRDVVQ KAQTKEQADF AIEALAKAKF ERLFRWILTR VNKALDKTKR QGASFLGILD IAGFEIFEIN SFEQL CINY TNEKLQQLFN HTMFILEQEE YQREGIEWNF IDFGLDLQPC IELIERPTNP PGVLALLDEE CWFPKATDTS FVEKLI QEQ GNHPKFQKSK QLKDKTEFCI LHYAGKVSYN ASAWLTKNMD PLNDNVTSLL NQSSDKFVAD LWKDVDRIVG LDQMAKM TE SSLPSSSKTK KGMFRTVGQL YKEQLTKLMT TLRNTNPNFV RCIIPNHEKR AGKLDAHLVL EQLRCNGVLE GIRICRQG F PNRIVFQEFR QRYEILAANA IPKGFMDGKQ ACILMIKALE LDPNLYRIGQ SKIFFRTGVL AHLEEERDLK ITDVIIAFQ AQCRGYLARK AFAKRQQQLT AMKVIQRNCA AYLKLRNWQW WRLFTKVKPL LQVTRQEEEM QAKDEELQRT KERQQKAEAE LKELEQKHT QLCEEKNLLQ EKLQAETELY AEAEEMRVRL AAKKQELEEI LHEMEARIEE EEERSQQLQA EKKKMQQQML D LEEQLEEE EAARQKLQLE KVTADGKIKK MEDDILIMED QNNKLTKERK LLEERVSDLT TNLAEEEEKA KNLTKLKNKH ES MISELEV RLKKEEKTRQ ELEKTKRKLE GESSDLHEQI AELQAQIAEL KAQLAKKEEE LQAALARLED ETSQKNNALK KIR ELESHI SDLQEDLESE KAARNKAEKQ KRDLGEELEA LKTELEDTLD TTATQQELRA KREQEVTVLK RALEEETRTH EAQV QEMRQ KHTQAVEELT EQLEQFKRAK ANLDKTKQTL EKDNADLANE VRSLSQAKQD VEHKKKKLEV QLQDLQSKYT DGERV RTEL NEKVHKLQIE VENVTSLLNE AESKNIKLTK DVATLGSQLQ DTQELLQEET RQKLNVTTKL RQLEDDKNSL QEQLDE EVE AKQNLERHIS TLTIQLSDSK KKLQEFTATI ETMEEGKKKF QREIESLTQQ FEEKAASYDK LEKTKNRLQQ ELDDLVV DL DNQRQLVSNL EKKQKKFDQM LAEEKNISSK YADERDRAEA EAREKETKAL SLARALEEAL EAKEELERTN KMLKAEME D LVSSKDDVGK NVHELEKSKR TLEQQVEEMK TQLEELEDEL QAAEDAKLRL EVNMQAMKSQ FERDLQARDE QNEEKRRQL LKQLHEHETE LEDERKQRAL AAAAKKKLEV DVKDLESQVD SVNKAREEAI KQLRKLQAQM KDYQRDLDDA RAAREEIFAT ARENEKKAK NLEAELIQLQ EDLAAAERAR KQADLEKEEM AEELASATSG RTSLQDDKRR LEARIAQLEE ELDEEHSNIE A MSDRMRKA VQQAEQLNNE LATERATAQK NENARQQLER QNKELRSKLQ EMEGAVKSKF KSTIAALEAK IASLEEQLEQ EA REKQAAA KTLRQKDKKL KDALLQVEDE KKQAEQYKDQ AEKGNLRLKQ LKRQLEEAEE ESQRINANRR KLQRELDEAT ESN DALGRE VAALKSKLRR GNEPVSFAPP RRSGGRRVIE NATDGGEQEI DGRDGDLNGK ASE UniProtKB: Myosin heavy chain 11 |
-分子 #2: Myosin light chain smooth muscle isoform
| 分子 | 名称: Myosin light chain smooth muscle isoform / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 16.989145 KDa |
| 配列 | 文字列: MCDFSEEQTA EFKEAFQLFD RTGDGKILYS QCGDVMRALG QNPTNAEVMK VLGNPKSDEM NLKTLNFEQF LPMMQTIAKN KDQGCFEDY VEGLRVFDKE GNGTVMGAEI RHVLVTLGEK MTEEEVEQLV AGHEDSNGCI NYEELVRMVL SG UniProtKB: Myosin light chain smooth muscle isoform |
-分子 #3: Myosin light chain 9
| 分子 | 名称: Myosin light chain 9 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 19.872244 KDa |
| 配列 | 文字列: MSSKRAKAKT TKKRPQRATS NVFAMFDQSQ IQEFKEAFNM IDQNRDGFID KEDLHDMLAS MGKNPTDEYL EGMMSEAPGP INFTMFLTM FGEKLNGTDP EDVIRNAFAC FDEEASGFIH EDHLRELLTT MGDRFTDEEV DEMYREAPID KKGNFNYVEF T RILKHGAK DKDD UniProtKB: Myosin light chain 9 |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 2 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #6: PHOSPHATE ION
| 分子 | 名称: PHOSPHATE ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: PO4 |
|---|---|
| 分子量 | 理論値: 94.971 Da |
| Chemical component information |  ChemComp-PO4: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.45 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AMYLAMINE / 詳細: GloCube | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot force 6, blot time 3 seconds. | ||||||||||||
| 詳細 | Smooth muscle myosin from turkey gizzard, stored under liquid nitrogen, was thawed quickly. MgATP was added before dilution into the final buffer conditions stated below |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)